オートファゴソームの形成にかかわるタンパク質の構造と分子機能
2014/11/07
野田展生1・稲垣冬彦2
(1微生物化学研究会微生物化学研究所 分子構造解析部,2北海道大学大学院先端生命科学研究院 次世代ポストゲノム研究センター)
email:野田展生,稲垣冬彦
領域融合レビュー, 3, e012 (2014) DOI: 10.7875/leading.author.3.e012
Nobuo N. Noda & Fuyuhiko Inagaki: Structure and molecular function of autophagosome-forming factors.

オートファジーにおける最大のイベントであるオートファゴソームの形成は18種類の主要なAtgタンパク質が担っており,そのうちの5種類が膜の形成の始動にかかわるAtg1複合体を,8種類が膜の伸長において実働部隊として機能するAtg8結合系およびAtg12結合系を構成している.近年,これら主要なAtgタンパク質に関する構造生物学的な研究が飛躍的に進展し,その構造基盤および分子機能の一端が明らかになってきた.このレビューでは,Atg1複合体とAtg8結合系およびAtg12結合系に着目し,これまでの研究により明らかにされた構造基盤および分子機能について最新の知見にもとづき解説するとともに,今後の課題についてふれる.
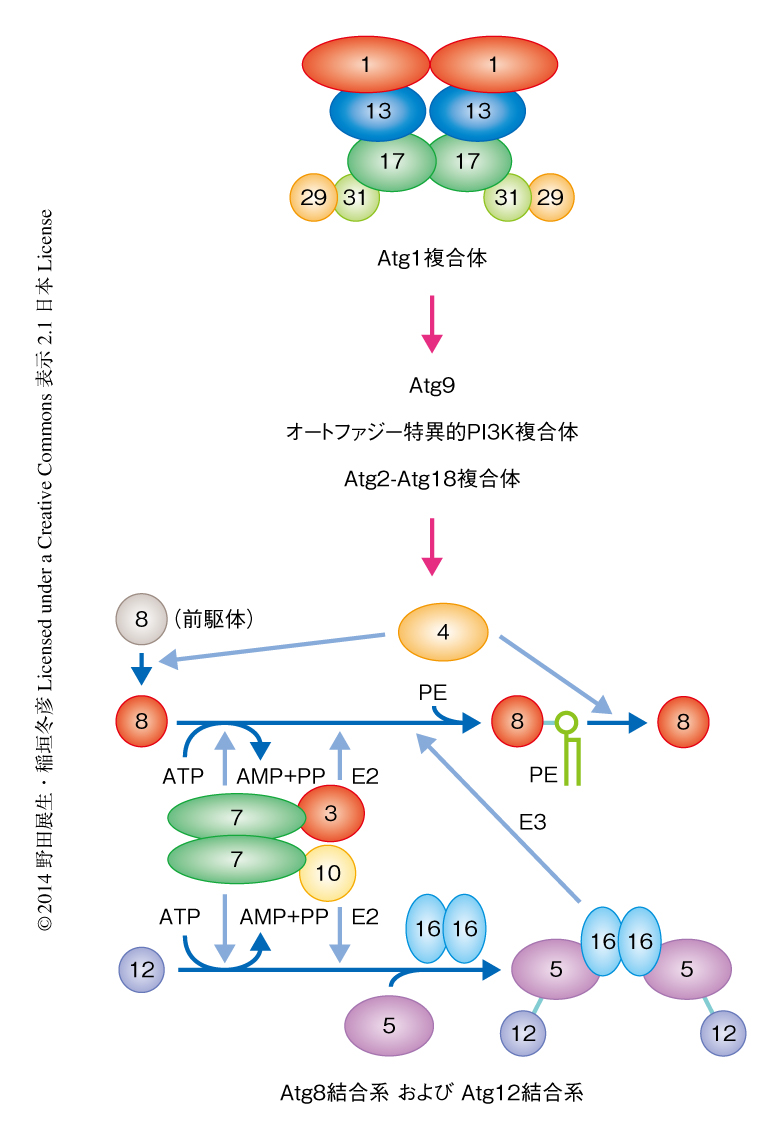
オートファジーは真核生物に保存された細胞における基本的な分解系であり,老廃物の分解やリサイクルをとおして細胞の恒常性の維持に寄与している1).オートファジーにおけるもっとも特徴的かつ重要なイベントは,二重膜からなる構造体であるオートファゴソームの形成である.オートファジーが誘導されると,突然,細胞質に隔離膜とよばれる膜構造が出現し,それが伸長して閉じることによりオートファゴソームが新生されるが,その過程でタンパク質やオルガネラなどの分解の対象がオートファゴソームの内部に隔離される.つづいて,オートファゴソームはリソソーム(酵母や植物では,液胞)と融合し,内容物は加水分解酵素のはたらきにより一網打尽に分解される.オートファジーを制御するAtgタンパク質はこれまでおもに出芽酵母を用いて同定され,その数は現時点で38種類に達している2,3).これらのうち主要なAtgタンパク質としてオートファゴソームの形成に関与するものは18種類であり,以下の6つの機能グループ,すなわち,1)Atg1複合体,2)Atg9,3)オートファジー特異的PI3K複合体(PI3K:phosphatidylinositol 3-kinase,ホスファチジルイノシトール3-キナーゼ),4)Atg2-Atg18複合体,5)Atg12結合系,6)Atg8結合系,に分類される3)(図1).これら6つの機能グループはPAS(pre-autophagosomal structure,プレオートファゴソーム構造体)に局在し,協働して機能することによりオートファゴソームの形成をひき起こす4).これらの主要なAtgタンパク質および機能グループは進化において広く保存されていることから,オートファゴソームの形成の基本的な分子機構もまた進化において保存されていると考えられる.このレビューでは,オートファゴソームの形成にかかわる主要なAtgタンパク質のうち,近年,構造生物学的な研究がいちじるしく進展したAtg1複合体とAtg8結合系およびAtg12結合系について,その構造と分子機能に関する最新の知見をまとめる.
なお,出芽酵母におけるオートファジーの研究については,荒木保弘・大隅良典, 領域融合レビュー, 1, e005, 2012,オートファジーと疾患とのかかわりについては,蔭山 俊・小松雅明, 領域融合レビュー, 3, e006, 2014 を参照されたい.
オートファジーは飢餓により強く誘導されるが,その際,まずオートファゴソームの形成の場であるPASが構築される4,5).PASはAtgタンパク質が集積して形成される構造体であるが,PASのもっとも中核はAtg1複合体が担うと考えられている5,6).すなわち,Atg1複合体はPASの構築をとおしてオートファゴソーム形成の始動装置として機能する.Atg1は主要なAtgタンパク質のなかで唯一のキナーゼであり,飢餓の際にAtg13,Atg17,Atg29,Atg31とAtg1複合体を形成する5,7).Atg1複合体はAtg9およびオートファジー特異的PI3K複合体をリクルートし,さらにこれらがAtg2-Atg18複合体,Atg8結合系,Atg12結合系をリクルートすることによりPASが完成する6)(図1).
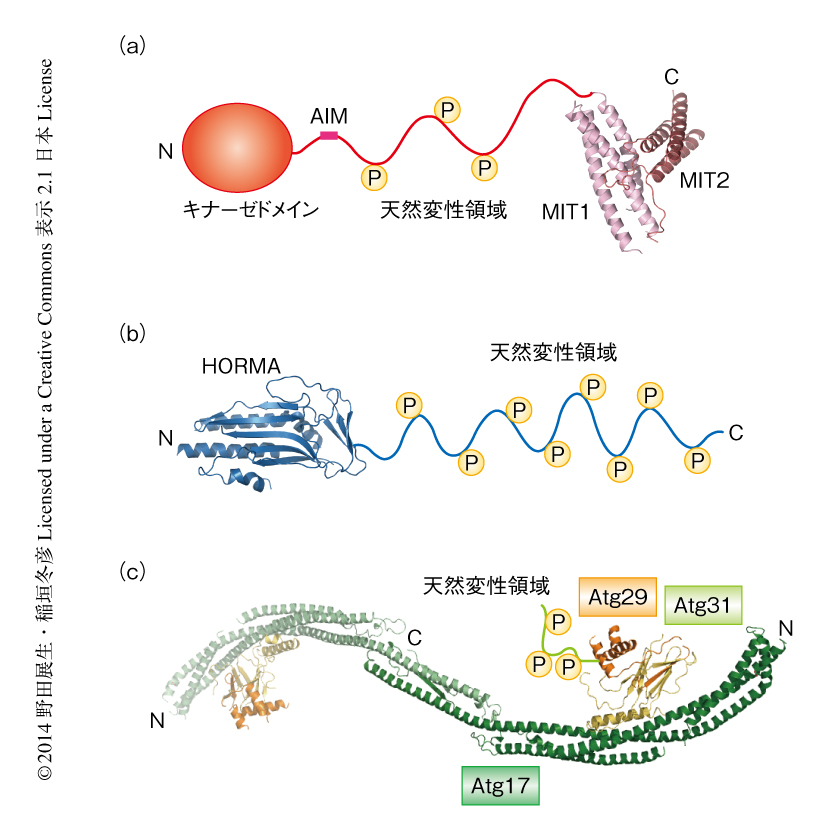
Atg1は構造的に3つの領域,すなわち,N末端側のキナーゼドメイン,C末端側の球状のドメイン,そして,両者をつなぐ天然変性領域に分けられる(図2a).キナーゼドメインの立体構造は実験的にはまだ決定されていないが,アミノ酸配列の相同性からプロテインキナーゼスーパーファミリーにおいて保存された構造をもつと予想されている.一方,C末端側の球状のドメインの構造は,Atg13のAtg1結合領域との複合体としてX線結晶解析により決定された8)(新着論文レビュー でも掲載).Atg1の球状のドメインは2つの3へリックスバンドルがタンデムにつながり,両者が互いに密接に相互作用することによりひとつの球状構造を形成している.それぞれの3へリックスバンドルはMITドメインと高い構造類似性を示したため,N末端側からMIT1およびMIT2と名づけられた8).MITドメインはMVB経路など膜輸送にかかわるタンパク質にしばしばみられるドメインであり,タンパク質どうしをつなぐ結合モジュールとして機能する.同様に,Atg1のMIT1およびMIT2はAtg13および選択的オートファジーにかかわるAtg11との結合に関与する8,9).また,Atg1のMIT1およびMIT2を含むC末端側の領域が大きな曲率をもつリポソームと結合することも報告されている10).しかし,その構造には膜の曲率を認識するような構造的な特徴はみられず,膜との結合の機構に関する詳細は不明である.Atg1の天然変性領域は約300残基からなり,アミノ酸配列から特定の立体構造をとらないことが予測される.Serに富み,それらのうちいくつかはリン酸化をうけることがわかっているが,その意義は不明である.Atg1の天然変性領域にはAtg8結合モチーフが含まれ,これを介してAtg8と直接的に結合する11,12).Atg8結合モチーフを介したAtg1とAtg8とのあいだの相互作用は両者のPASへの局在には不要であるが,Atg1が隔離膜に局在するために必要である.Atg1の隔離膜への局在は効率的なオートファジーに必要であることがわかっているが,Atg1が隔離膜において担う具体的な分子機能についてはわかっていない.
Atg13は構造的に2つの領域,すなわち,N末端側の球状のドメインとC末端側の天然変性領域とに分けられる(図2b).Atg13の球状ドメインの構造はX線結晶解析により決定された13).骨格の部分は5本のストランドからなるβシートとその片面にならんだ4本のαへリックスからなり,そのC末端側に3本のストランドからなるβシートが付加された構造をとる.骨格の部分の構造はHORMAドメインと同じトポロジーをもつことから,Atg13の球状のドメインはHORMAと命名された13).Atg13のHORMAはオートファジー特異的PI3K複合体がPASに局在するため必要であるが,両者のあいだの直接的な結合は確認されていない13).HORMAドメインをもつMad2の構造生物学的な研究により,Mad2は開いた構造と閉じた構造の2つの主要なコンホメーションの平衡状態にあり,リガンドの結合によりその平衡が閉じた構造へとかたよることがわかっている14).Atg13のHORMAの結晶構造は閉じた構造に類似した構造をとるが,これまで,開いた構造に類似した構造は報告されていない.Atg13のHORMAが同様の構造変化を示すのか,また,直接の結合相手が何であるのかは,今後,明らかにしなければならない課題である.Atg13のHORMA以外の約470残基は天然変性領域であると予測されている.Atg13の天然変性領域はSerおよびThrに富み,それらの多くがリン酸化をうけることがわかっている8).また,天然変性領域の約60残基の領域を用いてAtg1と,13残基からなる短い領域を用いてAtg17と,直接に結合する8).
Atg17,Atg29,Atg31は栄養条件にかかわらず恒常的に安定な複合体を形成し,in vitroにおいて2分子ずつのストイキオメトリーであることが示されている15).Atg17-Atg29-Atg31複合体の構造はX線結晶解析および電子顕微鏡を用いた単粒子解析により決定された9,10,16)(図2c).Atg17のプロトマーは4本のαへリックスからなる特徴的な三日月状の構造をとり,C末端側でホモ二量体化することにより全体としてS字状の構造をとる.Atg31は8本のストランドからなるβサンドイッチ構造をもつが,そのうちの1本はAtg29に由来することから,Atg31がその立体構造を保持するにはAtg29が必須と考えられる.βサンドイッチ構造にくわえ,Atg31はC末端に1本のαへリックスをもち,それを介してAtg17の3本のαへリックスと4へリックスバンドルを形成することによりAtg17の凹面に強固に結合する.Atg29はN末端側にAtg31のβサンドイッチに組み込まれるストランド,つづいて,3へリックスバンドル構造をもち,さらに,C末端側には天然変性領域をもつ.Atg29の天然変性領域もまたSerおよびThrに富み,それらのうちいくつかは飢餓に依存してリン酸化される9).Atg29の天然変性領域はオートファジーの活性には不要であり,むしろ,オートファジーを抑制する活性をもつ9).天然変性領域のリン酸化によりAtg29の阻害効果が解除されオートファジーが促進されるというモデルが提示されているが9),その分子機構の詳細は不明である.また,Atg29の天然変性領域はAtg11と直接的に結合するが9),今後,その意義についても明らかにしていく必要がある.
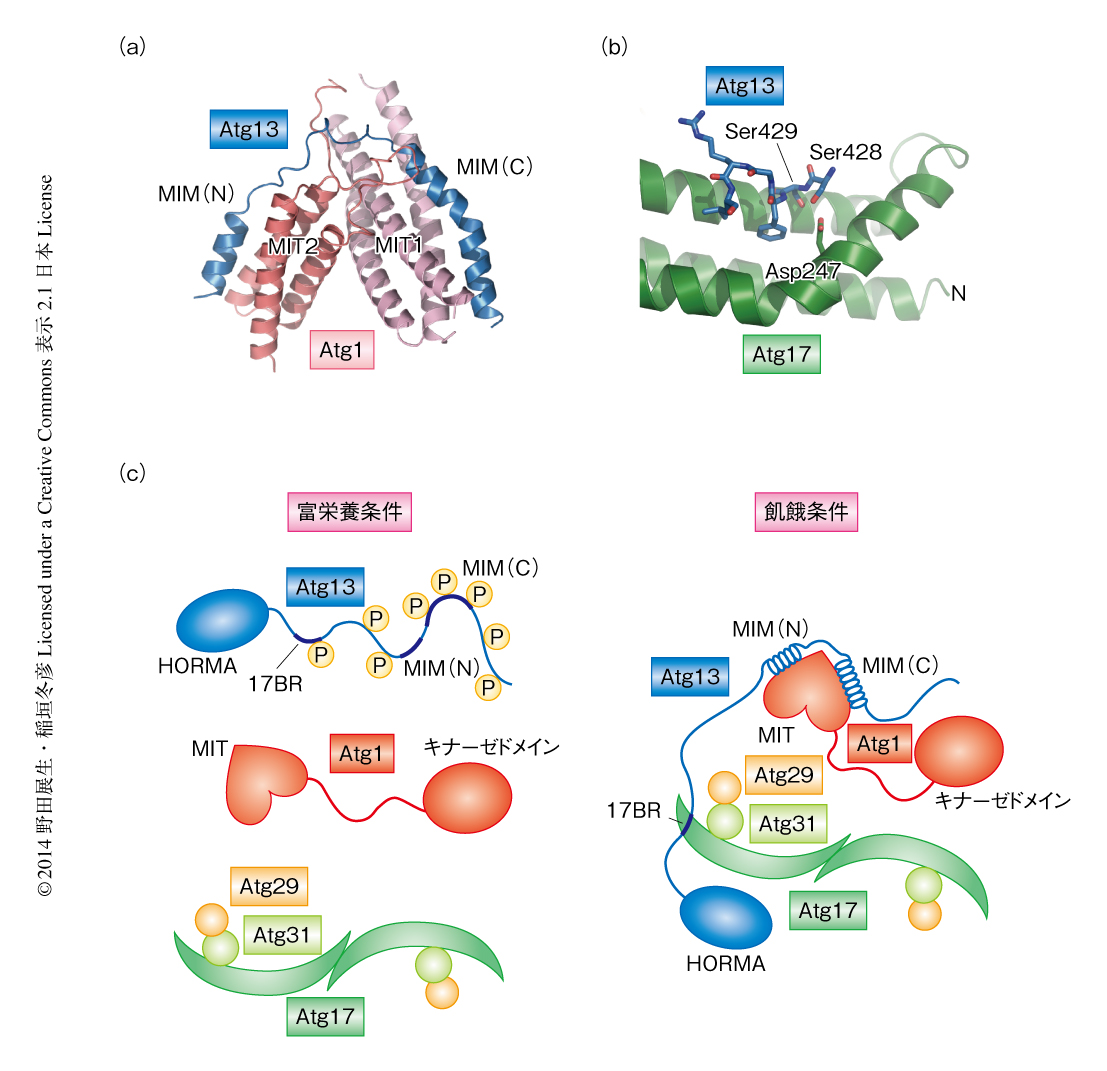
Atg13の天然変性領域のうち,Atg1およびAtg17との結合に必要十分な短い領域をそれぞれ用いることにより,Atg1-Atg13複合体およびAtg13-Atg17-Atg29-Atg31複合体の結晶構造が決定された8)(図3 a,b).Atg13のAtg1結合領域は天然変性領域に存在し,Atg1の非存在下においては特定の立体構造をもたない8).Atg1との結合に際し,Atg1結合領域はヘリックス-ループ-へリックス構造をとり,N末端側のへリックスを用いてAtg1のMIT2と,C末端側のへリックスを用いてAtg1のMIT1と結合する(図3a).これらの相互作用は一般的なMITドメインとMIT結合モチーフ(MIT-interacting motif:MIM)とのあいだにみられる相互作用と類似していたことから,N末端側のへリックス領域はMIM(N),C末端側のへリックス領域はMIM(C) と命名された8).Atg1とAtg13とのあいだの親和性はおもにMIM(N) とMIT2とのあいだの相互作用が担い,MIM(C) とMIT1とのあいだの相互作用はそれを補強する役割を担う.Atg13のAtg17結合領域は天然変性領域の13残基にまでしぼられており,この短い領域を介してAtg17のN末端の付近にある溝と結合する(図3b).疎水的な相互作用にくわえ,Atg13のSer428およびSer429がAtg17のAsp247と水素結合を形成しており(以下,残基番号は基本的に出芽酵母の場合),これらの水素結合はAtg13とAtg17とのあいだの相互作用に必須である8).
オートファジーは飢餓により強く誘導されるが,その制御には栄養センサーであるTOR複合体1がかかわっている17).富栄養条件において,TOR複合体1はAtg13の天然変性領域に含まれる多数のSerおよびThrを直接的にリン酸化する18).リン酸化されるSerはAtg1との結合にもっとも重要なMIM(N) には含まれず,相互作用を補強する役割をもつMIM(C) に複数が存在し,その結果,リン酸化はAtg1とAtg13とのあいだの親和性をある程度まで減弱させる8).一方,Atg13のAtg17結合領域には,リン酸化をうけるSerとしてAtg17のAsp247との水素結合にかかわるSer428およびSer429が含まれる.これらがリン酸化をうけることにより水素結合が破壊され,さらに負電荷の反発も生じることにより,Atg13とAtg17との結合は完全に失われる8).飢餓になるとTOR複合体1の活性が低下することによりAtg13はすみやかに脱リン酸化され,Atg1およびAtg17に対する高い親和性を獲得することによりAtg1複合体を形成しオートファジーの始動にはたらく7,8)(図3c).富栄養条件のもとでAtg1とAtg13とのあいだの相互作用がある程度まで保持されるのは,選択的オートファジーの一種であるCvt経路19) が進むために重要であると考えられる.
主要なAtgタンパク質18種類のうち,その半数に近い8種類がユビキチン様の結合反応系であるAtg8結合系およびAtg12結合系を形成する20)(図1).ユビキチン様タンパク質Atg12はユビキチン活性化酵素様の酵素Atg7およびユビキチン結合酵素様の酵素Atg10のはたらきによりAtg5のLys149側鎖とイソペプチド結合を形成する21).Atg12結合系はユビキチン転移酵素様の酵素を必要とせず,また,Atg12とAtg5との結合を切断する酵素も存在しない.Atg12-Atg5結合体はAtg16と複合体を形成し,つねにAtg12-Atg5-Atg16複合体として機能する22).もうひとつのユビキチン様タンパク質Atg8はユビキチン活性化酵素様の酵素Atg7およびユビキチン結合酵素様の酵素Atg3のはたらきによりリン脂質であるホスファチジルエタノールアミンとアミド結合を形成する23).Atg12-Atg5結合体とは異なり,Atg8-ホスファチジルエタノールアミン結合体は特異的なプロテアーゼAtg4による脱結合反応をうける24).Atg12-Atg5-Atg16複合体はAtg8とホスファチジルエタノールアミンとのあいだの結合を促進することから25),Atg8結合系におけるユビキチン転移酵素様の酵素と考えられている.Atg8結合系およびAtg12結合系のAtgタンパク質もPASに局在して機能するが,Atg1複合体がPASの中核として機能するのに対し,Atg8結合系およびAtg12結合系のAtgタンパク質は主要なAtgタンパク質のなかで最後にPASにリクルートされること6)(図1),さらに,伸長の途中の隔離膜にも局在することから26),オートファゴソーム形成の実働部隊と考えられる.
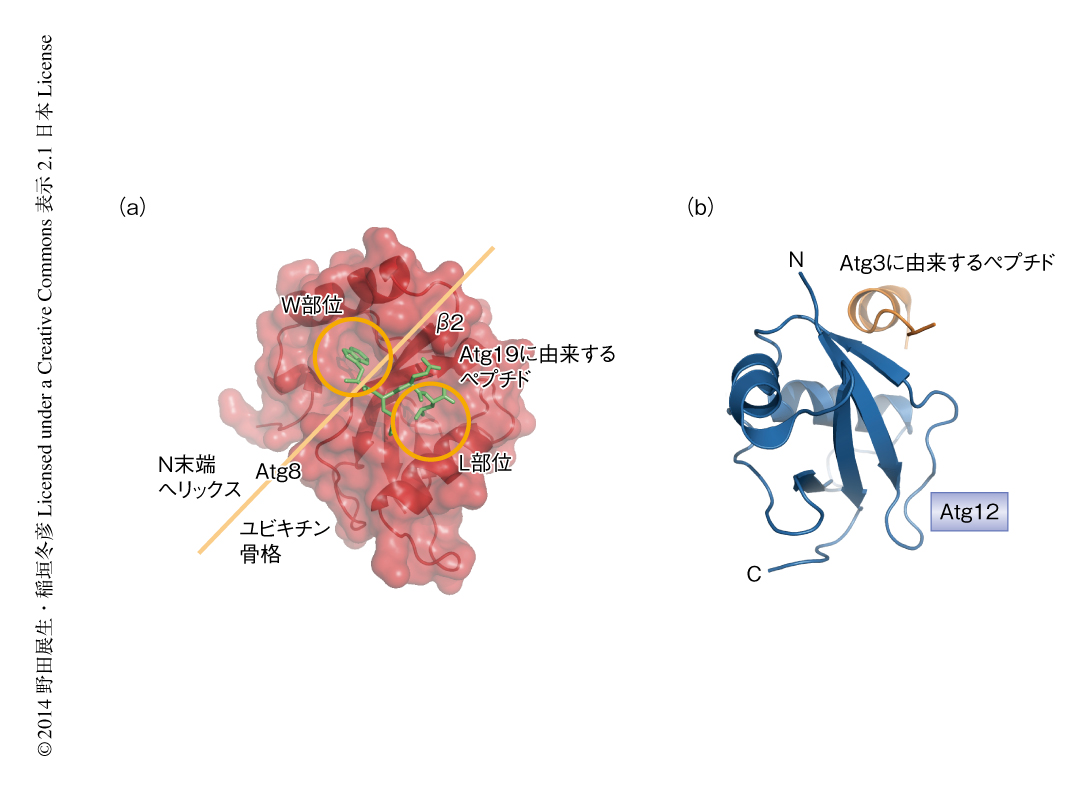
Atg8はさまざまな種に由来するものの構造が決定されてきたが,どれも共通して,ユビキチン骨格とそのN末端側に特徴的な2つのαへリックスからなる構造をとっている27,28)(図4a).特徴的なαへリックスの存在は,Atg8に固有の2つの機能,すなわち,膜の凝集能およびAtg8結合モチーフに対する特異的な認識能をあたえている.膜の凝集能の詳細な分子機構はまだわかっていないが,異なる膜に存在するAtg8-ホスファチジルエタノールアミン結合体どうしが相互作用することにより膜がつなぎとめられるというモデルが提示されている29).Atg8結合モチーフに対する特異的な認識能は,特徴的なαへリックスとユビキチン骨格とのあいだに形成された疎水性のポケットであるW部位,ユビキチン骨格に形成された疎水性のポケットであるL部位,2番目のβストランドにより担われており,Atg8結合モチーフの典型的な配列であるTrp-X-X-Leu配列(Xは任意のアミノ酸残基)のTrpをW部位において,LeuをL部位において,また,主鎖を2番目のβストランドが分子間βシートを形成することにより認識する27,30,31)(図4a).Atg8結合モチーフの典型的な配列のうち,TrpはTyrあるいはPhe,LeuはIleあるいはValにも代えられることがわかっている.Atg8結合モチーフは哺乳類のオートファジー関連タンパク質においても多く見い出されており,それらはLC3結合領域とよばれている27,32).Atg8結合モチーフあるいはLC3結合領域は選択的オートファジーにおいて基質の認識を担う多くの受容体に見い出されており,Atg8-ホスファチジルエタノールアミン結合体は基質の認識における膜での足場として機能する27).一方,Atg12についてはAtg8と比べその機能の解明は遅れている.Atg12において,ユビキチン骨格の構造は保存されているが33),そのN末端側には種によりさまざまな長さの配列が付加され,その役割はわかっていない.唯一,示唆されている機能はユビキチン結合酵素様の酵素Atg3の認識であり,Atg12-Atg5-Atg16複合体がユビキチン転移酵素様の酵素として機能するためにはたらくと考えられている34).哺乳類のAtg12はAtg3に含まれる特定の配列をユビキチン骨格により認識するが35)(図4b),この相互作用は酵母においては保存されておらず,さらなる解析が必要である.
Atg4およびAtg4-Atg8複合体の構造生物学的な研究は,哺乳類のAtg4オーソログであるAtg4BおよびAtg8オーソログであるLC3を用いて行われた36-38).Atg4は翻訳ののちのAtg8のプロセシングとAtg8-ホスファチジルエタノールアミン結合体の脱結合の両方を担うシステインプロテアーゼであり,パパインと類似した構造をもつ.単体のAtg4は活性部位を制御ループ,Trp(ヒトのAtg4Bの場合,Trp142),N末端テイルにより自己阻害する構造をとる.Atg8が結合するとそれらに大規模な構造変化が起こり,Atg8のC末端のGlyはAtg4の活性Cysの近傍に結合する(図5).Atg8ファミリーはC末端に芳香族アミノ酸残基-Gly配列を保存しており,この配列がAtg4の自己阻害の構造を解除するために必要である36).すなわち,自己阻害の構造はAtg4の基質特異性を担保する機構のひとつと考えられる.Atg4はオートファゴソームの形成に機能しているAtg8-ホスファチジルエタノールアミン結合体は切断しないことが示唆されているが,その活性の制御機構についてはわかっていない.
Atg8およびAtg12に共通するユビキチン活性化酵素様の酵素Atg7は,ATPのエネルギーを使うことにより両者のC末端のGlyを活性化し,それらとAtg7自体の活性Cysとのあいだでチオエステル結合を形成したのち,Atg8とそのユビキチン結合酵素様の酵素であるAtg3,あるいは,Atg12とそのユビキチン結合酵素様の酵素であるAtg10とのあいだにチオエステル結合を形成する反応を担う.Atg7は2つの球状ドメインであるN末端ドメインおよびC末端ドメインが短いリンカーでつながれた構造をもつ39-41).C末端ドメインはさらにアデニル化ドメインおよび最C末端ドメインに分けられ,アデニル化ドメインを介してホモ二量体を形成する(図6a).したがって,Atg7はすべてのドメインおよび活性部位を2つずつもつ.一般的なユビキチン活性化酵素はホモ二量体を形成せず,活性部位や同一のドメインは1つずつしか保持しないこととは対照的である.アデニル化ドメインはすべてのユビキチン活性化酵素において保存されているのに対し,N末端ドメインおよび最C末端ドメインはAtg7に固有のドメインである.一方,一般的なユビキチン活性化酵素に保存された活性Cysドメインおよびユビキチン様ドメインはAtg7には存在しない.Atg7の活性Cysはアデニル化ドメインに挿入されたループに存在する.
Atg8のユビキチン結合酵素様の酵素であるAtg3およびAtg12のユビキチン結合酵素様の酵素であるAtg10は,互いに類似したユビキチン結合酵素のコア構造をもち,一般的なユビキチン結合酵素とはやや異なりC末端側のαへリックス2つを欠いている42-44)(図6 b,c).それにくわえ,Atg10はコア構造から飛び出した特徴的なβヘアピン構造をもち,それを用いてAtg12の結合相手であるAtg5を直接的に認識する43)(図6b).一方,Atg3は2つのユニークな挿入領域であるハンドル領域およびフレキシブル領域をもつ42)(図6c).ハンドル領域はコア構造から飛び出した1本の長いαへリックスおよびループ領域からなり,ループ領域にあるAtg8結合モチーフによりAtg8と直接的に結合する45).フレキシブル領域は約80アミノ酸残基からなり,そのほとんどが天然変性状態をとるが,唯一,短いαへリックスをとる領域がAtg7のN末端ドメインと結合する40,42).Atg3のN末端の約20残基は結晶において電子密度が観測されなかったが,アミノ酸配列から両親媒性へリックス構造をとることが予測されており,曲率の大きな膜の認識に関与することが報告されている46,47).
Atg8のユビキチン転移酵素様の酵素であるAtg12-Atg5-Atg16複合体は,Atg16がホモ二量体を形成することによりおのおののタンパク質を2つずつ含む構造をとる48).Atg5は2つのユビキチン様ドメインとLys149を含むヘリックスに富むドメインからなり,これら3つのドメインが互いに相互作用することにより1つの球状の構造をとる49)(図6 b,d).Atg16は平行するホモ二量体を形成するコイルドコイルドメインと,Atg5との結合にかかわるαへリックスとが,フレキシブルなリンカーによりつながれた構造をとる48)(図6d).Atg12はさきに述べたとおりユビキチン骨格からなり,Atg5とイソペプチド結合によりつながれた構造においては,非共有結合性の相互作用も形成することによりAtg5の骨格に固定される50,51)(図4b,図6d).その結果,Atg12-Atg5-Atg16複合体の全体構造は6つのユビキチン様ドメインがコイルドコイル二量体の一端に集まった構造となり,ほかのユビキチン転移酵素の構造とはいちじるしく異なる(図6d).
まず,Atg7が最C末端ドメインにあるフレキシブルな領域でAtg8を補足したのち,Atg8をアデニル化ドメインへと配置換えする41)(図6a,図7).つぎに,Atg8のC末端のGlyがAtg7に結合したATPと反応することでアデニル化され,つづいて,Atg7の活性CysがAtg8のC末端のGlyとチオエステル結合を形成する.この過程において,活性Cysを含むループ領域が構造変化を起こすことが予想されるが,ほかのユビキチン活性化酵素においてみられるようなドメインどうしの大規模な配置転換は必要としない.Atg7はAtg12も類似の様式で認識し活性化すると思われるが,Atg7-Atg12複合体の構造生物学的な研究はこれまでなされていない.Atg7とチオエステル結合を形成したAtg8およびAtg12は,つづいて,それぞれのユビキチン結合酵素様の酵素であるAtg3およびAtg10とチオエステル結合を形成する.この際,どちらのユビキチン結合酵素様の酵素もAtg7のN末端ドメインの15番目のβストランドの付近に類似の様式で結合するが(図6a),ユビキチン様ドメインを用いる一般的なユビキチン活性化酵素とユビキチン結合酵素とのあいだの相互作用とはまったく異なる52,53).Atg7はホモ二量体を形成するため,2分子のユビキチン結合酵素様の酵素が結合することが可能である.したがって,Atg7の活性CysによりつながれたAtg8およびAtg12は,同じAtg7分子に結合したユビキチン結合酵素様の酵素と,ホモ二量体を形成したもう1分子のAtg7に結合したユビキチン結合酵素様の酵素の,どちらの活性Cysにも転移される可能性があるが,実際には,後者にのみ選択的に転移される40,41).Atg7-1の活性CysにつながれたAtg8は,Atg7-2に結合したAtg3へと転移される(図6a,図7).これは,ほかのユビキチン活性化酵素にはみられないAtg7に固有の機構であり,Atg7がホモ二量体を形成する理由のひとつと考えられる.生体においては,Atg8はAtg3のみと,Atg12はAtg10のみとチオエステル結合を形成すると考えられているが,in vitroでは,Atg3はAtg12とも,Atg10はAtg8ともチオエステル結合を形成する52).この特異性の問題は,今後,明らかにしなければならない課題である.
Atg10とチオエステル結合を形成したAtg12は,最後に,Atg5のLys149と結合する.Atg10は固有のβヘアピン構造を用いてAtg5のユビキチン様ドメインにあるβストランドと相互作用することにより,ユビキチン転移酵素様の酵素の助けなしに結合反応を担うと考えられる43)(図6b).一方,Atg3とチオエステル結合を形成したAtg8は,ユビキチン転移酵素様の酵素であるAtg12-Atg5-Atg16複合体の助けを借りることによりホスファチジルエタノールアミンと結合する(図7).Atg3の活性部位の構造は単独では低活性の状態であり,Atg12-Atg5-Atg16複合体と相互作用することにより高活性の状態に変換されることが生化学的な手法により強く示唆されている54).また,Atg12-Atg5-Atg16複合体はAtg3を適切な膜へとリクルートすることにより,Atg8の結合相手であるホスファチジルエタノールアミンとAtg8とを出会わせる機能も担うと考えられている34).しかし,これらの分子機構に関する理解は不十分であり,全容の解明のためにはさらなる構造生物学的な研究が必須である.
オートファゴソームの形成にかかわる主要なAtgタンパク質のうち,Atg1複合体とAtg8結合系およびAtg12結合系については,近年,構造生物学的な研究が急速に進展し,これらがはたす分子機能についてその一端が明らかにされた.しかしながら,たとえば,Atg17のとるS字状の構造は何をしているのかなど,構造がわかることによりさらなる謎もつぎつぎと生じている.また,Atg1複合体とAtg8結合系およびAtg12結合系とを橋渡しするAtgタンパク質の構造生物学的な研究はほとんど進展していないのが現状である.構造生物学的な研究と構造情報を活用した機能の研究を並行して進めることが,オートファジーにおける謎をひとつひとつ解明していくためにきわめて重要である.
略歴:2001年 東京大学大学院薬学系研究科博士課程 修了,同年 北海道大学大学院薬学研究科 博士研究員,同 助教,同講師を経て,2011年より微生物化学研究会微生物化学研究所 主席研究員.
研究テーマ:オートファジーの分子機構.
関心事:オートキャンプ.
稲垣 冬彦(Fuyuhiko Inagaki)
北海道大学大学院先端生命科学研究院 特任教授.
研究テーマ:核磁気共鳴,構造生物学,シグナル伝達,オートファジー.
© 2014 野田展生・稲垣冬彦 Licensed under CC 表示 2.1 日本
(1微生物化学研究会微生物化学研究所 分子構造解析部,2北海道大学大学院先端生命科学研究院 次世代ポストゲノム研究センター)
email:野田展生,稲垣冬彦
領域融合レビュー, 3, e012 (2014) DOI: 10.7875/leading.author.3.e012
Nobuo N. Noda & Fuyuhiko Inagaki: Structure and molecular function of autophagosome-forming factors.
要 約
オートファジーにおける最大のイベントであるオートファゴソームの形成は18種類の主要なAtgタンパク質が担っており,そのうちの5種類が膜の形成の始動にかかわるAtg1複合体を,8種類が膜の伸長において実働部隊として機能するAtg8結合系およびAtg12結合系を構成している.近年,これら主要なAtgタンパク質に関する構造生物学的な研究が飛躍的に進展し,その構造基盤および分子機能の一端が明らかになってきた.このレビューでは,Atg1複合体とAtg8結合系およびAtg12結合系に着目し,これまでの研究により明らかにされた構造基盤および分子機能について最新の知見にもとづき解説するとともに,今後の課題についてふれる.
はじめに
オートファジーは真核生物に保存された細胞における基本的な分解系であり,老廃物の分解やリサイクルをとおして細胞の恒常性の維持に寄与している1).オートファジーにおけるもっとも特徴的かつ重要なイベントは,二重膜からなる構造体であるオートファゴソームの形成である.オートファジーが誘導されると,突然,細胞質に隔離膜とよばれる膜構造が出現し,それが伸長して閉じることによりオートファゴソームが新生されるが,その過程でタンパク質やオルガネラなどの分解の対象がオートファゴソームの内部に隔離される.つづいて,オートファゴソームはリソソーム(酵母や植物では,液胞)と融合し,内容物は加水分解酵素のはたらきにより一網打尽に分解される.オートファジーを制御するAtgタンパク質はこれまでおもに出芽酵母を用いて同定され,その数は現時点で38種類に達している2,3).これらのうち主要なAtgタンパク質としてオートファゴソームの形成に関与するものは18種類であり,以下の6つの機能グループ,すなわち,1)Atg1複合体,2)Atg9,3)オートファジー特異的PI3K複合体(PI3K:phosphatidylinositol 3-kinase,ホスファチジルイノシトール3-キナーゼ),4)Atg2-Atg18複合体,5)Atg12結合系,6)Atg8結合系,に分類される3)(図1).これら6つの機能グループはPAS(pre-autophagosomal structure,プレオートファゴソーム構造体)に局在し,協働して機能することによりオートファゴソームの形成をひき起こす4).これらの主要なAtgタンパク質および機能グループは進化において広く保存されていることから,オートファゴソームの形成の基本的な分子機構もまた進化において保存されていると考えられる.このレビューでは,オートファゴソームの形成にかかわる主要なAtgタンパク質のうち,近年,構造生物学的な研究がいちじるしく進展したAtg1複合体とAtg8結合系およびAtg12結合系について,その構造と分子機能に関する最新の知見をまとめる.
なお,出芽酵母におけるオートファジーの研究については,荒木保弘・大隅良典, 領域融合レビュー, 1, e005, 2012,オートファジーと疾患とのかかわりについては,蔭山 俊・小松雅明, 領域融合レビュー, 3, e006, 2014 を参照されたい.
1.Atg1複合体:オートファゴソーム形成の始動装置
オートファジーは飢餓により強く誘導されるが,その際,まずオートファゴソームの形成の場であるPASが構築される4,5).PASはAtgタンパク質が集積して形成される構造体であるが,PASのもっとも中核はAtg1複合体が担うと考えられている5,6).すなわち,Atg1複合体はPASの構築をとおしてオートファゴソーム形成の始動装置として機能する.Atg1は主要なAtgタンパク質のなかで唯一のキナーゼであり,飢餓の際にAtg13,Atg17,Atg29,Atg31とAtg1複合体を形成する5,7).Atg1複合体はAtg9およびオートファジー特異的PI3K複合体をリクルートし,さらにこれらがAtg2-Atg18複合体,Atg8結合系,Atg12結合系をリクルートすることによりPASが完成する6)(図1).
2.Atg1複合体の構成タンパク質の構造
Atg1は構造的に3つの領域,すなわち,N末端側のキナーゼドメイン,C末端側の球状のドメイン,そして,両者をつなぐ天然変性領域に分けられる(図2a).キナーゼドメインの立体構造は実験的にはまだ決定されていないが,アミノ酸配列の相同性からプロテインキナーゼスーパーファミリーにおいて保存された構造をもつと予想されている.一方,C末端側の球状のドメインの構造は,Atg13のAtg1結合領域との複合体としてX線結晶解析により決定された8)(新着論文レビュー でも掲載).Atg1の球状のドメインは2つの3へリックスバンドルがタンデムにつながり,両者が互いに密接に相互作用することによりひとつの球状構造を形成している.それぞれの3へリックスバンドルはMITドメインと高い構造類似性を示したため,N末端側からMIT1およびMIT2と名づけられた8).MITドメインはMVB経路など膜輸送にかかわるタンパク質にしばしばみられるドメインであり,タンパク質どうしをつなぐ結合モジュールとして機能する.同様に,Atg1のMIT1およびMIT2はAtg13および選択的オートファジーにかかわるAtg11との結合に関与する8,9).また,Atg1のMIT1およびMIT2を含むC末端側の領域が大きな曲率をもつリポソームと結合することも報告されている10).しかし,その構造には膜の曲率を認識するような構造的な特徴はみられず,膜との結合の機構に関する詳細は不明である.Atg1の天然変性領域は約300残基からなり,アミノ酸配列から特定の立体構造をとらないことが予測される.Serに富み,それらのうちいくつかはリン酸化をうけることがわかっているが,その意義は不明である.Atg1の天然変性領域にはAtg8結合モチーフが含まれ,これを介してAtg8と直接的に結合する11,12).Atg8結合モチーフを介したAtg1とAtg8とのあいだの相互作用は両者のPASへの局在には不要であるが,Atg1が隔離膜に局在するために必要である.Atg1の隔離膜への局在は効率的なオートファジーに必要であることがわかっているが,Atg1が隔離膜において担う具体的な分子機能についてはわかっていない.
Atg13は構造的に2つの領域,すなわち,N末端側の球状のドメインとC末端側の天然変性領域とに分けられる(図2b).Atg13の球状ドメインの構造はX線結晶解析により決定された13).骨格の部分は5本のストランドからなるβシートとその片面にならんだ4本のαへリックスからなり,そのC末端側に3本のストランドからなるβシートが付加された構造をとる.骨格の部分の構造はHORMAドメインと同じトポロジーをもつことから,Atg13の球状のドメインはHORMAと命名された13).Atg13のHORMAはオートファジー特異的PI3K複合体がPASに局在するため必要であるが,両者のあいだの直接的な結合は確認されていない13).HORMAドメインをもつMad2の構造生物学的な研究により,Mad2は開いた構造と閉じた構造の2つの主要なコンホメーションの平衡状態にあり,リガンドの結合によりその平衡が閉じた構造へとかたよることがわかっている14).Atg13のHORMAの結晶構造は閉じた構造に類似した構造をとるが,これまで,開いた構造に類似した構造は報告されていない.Atg13のHORMAが同様の構造変化を示すのか,また,直接の結合相手が何であるのかは,今後,明らかにしなければならない課題である.Atg13のHORMA以外の約470残基は天然変性領域であると予測されている.Atg13の天然変性領域はSerおよびThrに富み,それらの多くがリン酸化をうけることがわかっている8).また,天然変性領域の約60残基の領域を用いてAtg1と,13残基からなる短い領域を用いてAtg17と,直接に結合する8).
Atg17,Atg29,Atg31は栄養条件にかかわらず恒常的に安定な複合体を形成し,in vitroにおいて2分子ずつのストイキオメトリーであることが示されている15).Atg17-Atg29-Atg31複合体の構造はX線結晶解析および電子顕微鏡を用いた単粒子解析により決定された9,10,16)(図2c).Atg17のプロトマーは4本のαへリックスからなる特徴的な三日月状の構造をとり,C末端側でホモ二量体化することにより全体としてS字状の構造をとる.Atg31は8本のストランドからなるβサンドイッチ構造をもつが,そのうちの1本はAtg29に由来することから,Atg31がその立体構造を保持するにはAtg29が必須と考えられる.βサンドイッチ構造にくわえ,Atg31はC末端に1本のαへリックスをもち,それを介してAtg17の3本のαへリックスと4へリックスバンドルを形成することによりAtg17の凹面に強固に結合する.Atg29はN末端側にAtg31のβサンドイッチに組み込まれるストランド,つづいて,3へリックスバンドル構造をもち,さらに,C末端側には天然変性領域をもつ.Atg29の天然変性領域もまたSerおよびThrに富み,それらのうちいくつかは飢餓に依存してリン酸化される9).Atg29の天然変性領域はオートファジーの活性には不要であり,むしろ,オートファジーを抑制する活性をもつ9).天然変性領域のリン酸化によりAtg29の阻害効果が解除されオートファジーが促進されるというモデルが提示されているが9),その分子機構の詳細は不明である.また,Atg29の天然変性領域はAtg11と直接的に結合するが9),今後,その意義についても明らかにしていく必要がある.
3.Atg13を介したAtg1複合体の構築の基盤
Atg13の天然変性領域のうち,Atg1およびAtg17との結合に必要十分な短い領域をそれぞれ用いることにより,Atg1-Atg13複合体およびAtg13-Atg17-Atg29-Atg31複合体の結晶構造が決定された8)(図3 a,b).Atg13のAtg1結合領域は天然変性領域に存在し,Atg1の非存在下においては特定の立体構造をもたない8).Atg1との結合に際し,Atg1結合領域はヘリックス-ループ-へリックス構造をとり,N末端側のへリックスを用いてAtg1のMIT2と,C末端側のへリックスを用いてAtg1のMIT1と結合する(図3a).これらの相互作用は一般的なMITドメインとMIT結合モチーフ(MIT-interacting motif:MIM)とのあいだにみられる相互作用と類似していたことから,N末端側のへリックス領域はMIM(N),C末端側のへリックス領域はMIM(C) と命名された8).Atg1とAtg13とのあいだの親和性はおもにMIM(N) とMIT2とのあいだの相互作用が担い,MIM(C) とMIT1とのあいだの相互作用はそれを補強する役割を担う.Atg13のAtg17結合領域は天然変性領域の13残基にまでしぼられており,この短い領域を介してAtg17のN末端の付近にある溝と結合する(図3b).疎水的な相互作用にくわえ,Atg13のSer428およびSer429がAtg17のAsp247と水素結合を形成しており(以下,残基番号は基本的に出芽酵母の場合),これらの水素結合はAtg13とAtg17とのあいだの相互作用に必須である8).
4.Atg13の飢餓に依存的な脱リン酸化を介したAtg1複合体の形成の機構
オートファジーは飢餓により強く誘導されるが,その制御には栄養センサーであるTOR複合体1がかかわっている17).富栄養条件において,TOR複合体1はAtg13の天然変性領域に含まれる多数のSerおよびThrを直接的にリン酸化する18).リン酸化されるSerはAtg1との結合にもっとも重要なMIM(N) には含まれず,相互作用を補強する役割をもつMIM(C) に複数が存在し,その結果,リン酸化はAtg1とAtg13とのあいだの親和性をある程度まで減弱させる8).一方,Atg13のAtg17結合領域には,リン酸化をうけるSerとしてAtg17のAsp247との水素結合にかかわるSer428およびSer429が含まれる.これらがリン酸化をうけることにより水素結合が破壊され,さらに負電荷の反発も生じることにより,Atg13とAtg17との結合は完全に失われる8).飢餓になるとTOR複合体1の活性が低下することによりAtg13はすみやかに脱リン酸化され,Atg1およびAtg17に対する高い親和性を獲得することによりAtg1複合体を形成しオートファジーの始動にはたらく7,8)(図3c).富栄養条件のもとでAtg1とAtg13とのあいだの相互作用がある程度まで保持されるのは,選択的オートファジーの一種であるCvt経路19) が進むために重要であると考えられる.
5.Atg8結合系およびAtg12結合系:オートファゴソーム形成の実働部隊
主要なAtgタンパク質18種類のうち,その半数に近い8種類がユビキチン様の結合反応系であるAtg8結合系およびAtg12結合系を形成する20)(図1).ユビキチン様タンパク質Atg12はユビキチン活性化酵素様の酵素Atg7およびユビキチン結合酵素様の酵素Atg10のはたらきによりAtg5のLys149側鎖とイソペプチド結合を形成する21).Atg12結合系はユビキチン転移酵素様の酵素を必要とせず,また,Atg12とAtg5との結合を切断する酵素も存在しない.Atg12-Atg5結合体はAtg16と複合体を形成し,つねにAtg12-Atg5-Atg16複合体として機能する22).もうひとつのユビキチン様タンパク質Atg8はユビキチン活性化酵素様の酵素Atg7およびユビキチン結合酵素様の酵素Atg3のはたらきによりリン脂質であるホスファチジルエタノールアミンとアミド結合を形成する23).Atg12-Atg5結合体とは異なり,Atg8-ホスファチジルエタノールアミン結合体は特異的なプロテアーゼAtg4による脱結合反応をうける24).Atg12-Atg5-Atg16複合体はAtg8とホスファチジルエタノールアミンとのあいだの結合を促進することから25),Atg8結合系におけるユビキチン転移酵素様の酵素と考えられている.Atg8結合系およびAtg12結合系のAtgタンパク質もPASに局在して機能するが,Atg1複合体がPASの中核として機能するのに対し,Atg8結合系およびAtg12結合系のAtgタンパク質は主要なAtgタンパク質のなかで最後にPASにリクルートされること6)(図1),さらに,伸長の途中の隔離膜にも局在することから26),オートファゴソーム形成の実働部隊と考えられる.
6.オートファジーに特化したユビキチン様タンパク質Atg8およびAtg12
Atg8はさまざまな種に由来するものの構造が決定されてきたが,どれも共通して,ユビキチン骨格とそのN末端側に特徴的な2つのαへリックスからなる構造をとっている27,28)(図4a).特徴的なαへリックスの存在は,Atg8に固有の2つの機能,すなわち,膜の凝集能およびAtg8結合モチーフに対する特異的な認識能をあたえている.膜の凝集能の詳細な分子機構はまだわかっていないが,異なる膜に存在するAtg8-ホスファチジルエタノールアミン結合体どうしが相互作用することにより膜がつなぎとめられるというモデルが提示されている29).Atg8結合モチーフに対する特異的な認識能は,特徴的なαへリックスとユビキチン骨格とのあいだに形成された疎水性のポケットであるW部位,ユビキチン骨格に形成された疎水性のポケットであるL部位,2番目のβストランドにより担われており,Atg8結合モチーフの典型的な配列であるTrp-X-X-Leu配列(Xは任意のアミノ酸残基)のTrpをW部位において,LeuをL部位において,また,主鎖を2番目のβストランドが分子間βシートを形成することにより認識する27,30,31)(図4a).Atg8結合モチーフの典型的な配列のうち,TrpはTyrあるいはPhe,LeuはIleあるいはValにも代えられることがわかっている.Atg8結合モチーフは哺乳類のオートファジー関連タンパク質においても多く見い出されており,それらはLC3結合領域とよばれている27,32).Atg8結合モチーフあるいはLC3結合領域は選択的オートファジーにおいて基質の認識を担う多くの受容体に見い出されており,Atg8-ホスファチジルエタノールアミン結合体は基質の認識における膜での足場として機能する27).一方,Atg12についてはAtg8と比べその機能の解明は遅れている.Atg12において,ユビキチン骨格の構造は保存されているが33),そのN末端側には種によりさまざまな長さの配列が付加され,その役割はわかっていない.唯一,示唆されている機能はユビキチン結合酵素様の酵素Atg3の認識であり,Atg12-Atg5-Atg16複合体がユビキチン転移酵素様の酵素として機能するためにはたらくと考えられている34).哺乳類のAtg12はAtg3に含まれる特定の配列をユビキチン骨格により認識するが35)(図4b),この相互作用は酵母においては保存されておらず,さらなる解析が必要である.
7.脱結合酵素Atg4の構造と活性の発現の機構
Atg4およびAtg4-Atg8複合体の構造生物学的な研究は,哺乳類のAtg4オーソログであるAtg4BおよびAtg8オーソログであるLC3を用いて行われた36-38).Atg4は翻訳ののちのAtg8のプロセシングとAtg8-ホスファチジルエタノールアミン結合体の脱結合の両方を担うシステインプロテアーゼであり,パパインと類似した構造をもつ.単体のAtg4は活性部位を制御ループ,Trp(ヒトのAtg4Bの場合,Trp142),N末端テイルにより自己阻害する構造をとる.Atg8が結合するとそれらに大規模な構造変化が起こり,Atg8のC末端のGlyはAtg4の活性Cysの近傍に結合する(図5).Atg8ファミリーはC末端に芳香族アミノ酸残基-Gly配列を保存しており,この配列がAtg4の自己阻害の構造を解除するために必要である36).すなわち,自己阻害の構造はAtg4の基質特異性を担保する機構のひとつと考えられる.Atg4はオートファゴソームの形成に機能しているAtg8-ホスファチジルエタノールアミン結合体は切断しないことが示唆されているが,その活性の制御機構についてはわかっていない.
8.ユビキチン様の結合反応系をつかさどる酵素の構造
Atg8およびAtg12に共通するユビキチン活性化酵素様の酵素Atg7は,ATPのエネルギーを使うことにより両者のC末端のGlyを活性化し,それらとAtg7自体の活性Cysとのあいだでチオエステル結合を形成したのち,Atg8とそのユビキチン結合酵素様の酵素であるAtg3,あるいは,Atg12とそのユビキチン結合酵素様の酵素であるAtg10とのあいだにチオエステル結合を形成する反応を担う.Atg7は2つの球状ドメインであるN末端ドメインおよびC末端ドメインが短いリンカーでつながれた構造をもつ39-41).C末端ドメインはさらにアデニル化ドメインおよび最C末端ドメインに分けられ,アデニル化ドメインを介してホモ二量体を形成する(図6a).したがって,Atg7はすべてのドメインおよび活性部位を2つずつもつ.一般的なユビキチン活性化酵素はホモ二量体を形成せず,活性部位や同一のドメインは1つずつしか保持しないこととは対照的である.アデニル化ドメインはすべてのユビキチン活性化酵素において保存されているのに対し,N末端ドメインおよび最C末端ドメインはAtg7に固有のドメインである.一方,一般的なユビキチン活性化酵素に保存された活性Cysドメインおよびユビキチン様ドメインはAtg7には存在しない.Atg7の活性Cysはアデニル化ドメインに挿入されたループに存在する.
Atg8のユビキチン結合酵素様の酵素であるAtg3およびAtg12のユビキチン結合酵素様の酵素であるAtg10は,互いに類似したユビキチン結合酵素のコア構造をもち,一般的なユビキチン結合酵素とはやや異なりC末端側のαへリックス2つを欠いている42-44)(図6 b,c).それにくわえ,Atg10はコア構造から飛び出した特徴的なβヘアピン構造をもち,それを用いてAtg12の結合相手であるAtg5を直接的に認識する43)(図6b).一方,Atg3は2つのユニークな挿入領域であるハンドル領域およびフレキシブル領域をもつ42)(図6c).ハンドル領域はコア構造から飛び出した1本の長いαへリックスおよびループ領域からなり,ループ領域にあるAtg8結合モチーフによりAtg8と直接的に結合する45).フレキシブル領域は約80アミノ酸残基からなり,そのほとんどが天然変性状態をとるが,唯一,短いαへリックスをとる領域がAtg7のN末端ドメインと結合する40,42).Atg3のN末端の約20残基は結晶において電子密度が観測されなかったが,アミノ酸配列から両親媒性へリックス構造をとることが予測されており,曲率の大きな膜の認識に関与することが報告されている46,47).
Atg8のユビキチン転移酵素様の酵素であるAtg12-Atg5-Atg16複合体は,Atg16がホモ二量体を形成することによりおのおののタンパク質を2つずつ含む構造をとる48).Atg5は2つのユビキチン様ドメインとLys149を含むヘリックスに富むドメインからなり,これら3つのドメインが互いに相互作用することにより1つの球状の構造をとる49)(図6 b,d).Atg16は平行するホモ二量体を形成するコイルドコイルドメインと,Atg5との結合にかかわるαへリックスとが,フレキシブルなリンカーによりつながれた構造をとる48)(図6d).Atg12はさきに述べたとおりユビキチン骨格からなり,Atg5とイソペプチド結合によりつながれた構造においては,非共有結合性の相互作用も形成することによりAtg5の骨格に固定される50,51)(図4b,図6d).その結果,Atg12-Atg5-Atg16複合体の全体構造は6つのユビキチン様ドメインがコイルドコイル二量体の一端に集まった構造となり,ほかのユビキチン転移酵素の構造とはいちじるしく異なる(図6d).
9.Atgタンパク質の結合反応の分子機構
まず,Atg7が最C末端ドメインにあるフレキシブルな領域でAtg8を補足したのち,Atg8をアデニル化ドメインへと配置換えする41)(図6a,図7).つぎに,Atg8のC末端のGlyがAtg7に結合したATPと反応することでアデニル化され,つづいて,Atg7の活性CysがAtg8のC末端のGlyとチオエステル結合を形成する.この過程において,活性Cysを含むループ領域が構造変化を起こすことが予想されるが,ほかのユビキチン活性化酵素においてみられるようなドメインどうしの大規模な配置転換は必要としない.Atg7はAtg12も類似の様式で認識し活性化すると思われるが,Atg7-Atg12複合体の構造生物学的な研究はこれまでなされていない.Atg7とチオエステル結合を形成したAtg8およびAtg12は,つづいて,それぞれのユビキチン結合酵素様の酵素であるAtg3およびAtg10とチオエステル結合を形成する.この際,どちらのユビキチン結合酵素様の酵素もAtg7のN末端ドメインの15番目のβストランドの付近に類似の様式で結合するが(図6a),ユビキチン様ドメインを用いる一般的なユビキチン活性化酵素とユビキチン結合酵素とのあいだの相互作用とはまったく異なる52,53).Atg7はホモ二量体を形成するため,2分子のユビキチン結合酵素様の酵素が結合することが可能である.したがって,Atg7の活性CysによりつながれたAtg8およびAtg12は,同じAtg7分子に結合したユビキチン結合酵素様の酵素と,ホモ二量体を形成したもう1分子のAtg7に結合したユビキチン結合酵素様の酵素の,どちらの活性Cysにも転移される可能性があるが,実際には,後者にのみ選択的に転移される40,41).Atg7-1の活性CysにつながれたAtg8は,Atg7-2に結合したAtg3へと転移される(図6a,図7).これは,ほかのユビキチン活性化酵素にはみられないAtg7に固有の機構であり,Atg7がホモ二量体を形成する理由のひとつと考えられる.生体においては,Atg8はAtg3のみと,Atg12はAtg10のみとチオエステル結合を形成すると考えられているが,in vitroでは,Atg3はAtg12とも,Atg10はAtg8ともチオエステル結合を形成する52).この特異性の問題は,今後,明らかにしなければならない課題である.
Atg10とチオエステル結合を形成したAtg12は,最後に,Atg5のLys149と結合する.Atg10は固有のβヘアピン構造を用いてAtg5のユビキチン様ドメインにあるβストランドと相互作用することにより,ユビキチン転移酵素様の酵素の助けなしに結合反応を担うと考えられる43)(図6b).一方,Atg3とチオエステル結合を形成したAtg8は,ユビキチン転移酵素様の酵素であるAtg12-Atg5-Atg16複合体の助けを借りることによりホスファチジルエタノールアミンと結合する(図7).Atg3の活性部位の構造は単独では低活性の状態であり,Atg12-Atg5-Atg16複合体と相互作用することにより高活性の状態に変換されることが生化学的な手法により強く示唆されている54).また,Atg12-Atg5-Atg16複合体はAtg3を適切な膜へとリクルートすることにより,Atg8の結合相手であるホスファチジルエタノールアミンとAtg8とを出会わせる機能も担うと考えられている34).しかし,これらの分子機構に関する理解は不十分であり,全容の解明のためにはさらなる構造生物学的な研究が必須である.
おわりに
オートファゴソームの形成にかかわる主要なAtgタンパク質のうち,Atg1複合体とAtg8結合系およびAtg12結合系については,近年,構造生物学的な研究が急速に進展し,これらがはたす分子機能についてその一端が明らかにされた.しかしながら,たとえば,Atg17のとるS字状の構造は何をしているのかなど,構造がわかることによりさらなる謎もつぎつぎと生じている.また,Atg1複合体とAtg8結合系およびAtg12結合系とを橋渡しするAtgタンパク質の構造生物学的な研究はほとんど進展していないのが現状である.構造生物学的な研究と構造情報を活用した機能の研究を並行して進めることが,オートファジーにおける謎をひとつひとつ解明していくためにきわめて重要である.
文 献
- Mizushima, N. & Komatsu, M.: Autophagy: renovation of cells and tissues. Cell, 147, 728-741 (2011)[PubMed]
- Araki, Y., Ku, W. C., Akioka, M. et al.: Atg38 is required for autophagy-specific phosphatidylinositol 3-kinase complex integrity. J. Cell Biol., 203, 299-313 (2013)[PubMed]
- Mizushima, N., Yoshimori, T. & Ohsumi, Y.: The role of Atg proteins in autophagosome formation. Annu. Rev. Cell Dev. Biol., 27, 107-132 (2011)[PubMed]
- Suzuki, K., Kirisako, T., Kamada, Y. et al.: The pre-autophagosomal structure organized by concerted functions of APG genes is essential for autophagosome formation. EMBO J., 20, 5971-5981 (2001)[PubMed]
- Kawamata, T., Kamada, Y., Kabeya, Y. et al.: Organization of the pre-autophagosomal structure responsible for autophagosome formation. Mol. Biol. Cell, 19, 2039-2050 (2008)[PubMed]
- Suzuki, K., Kubota, Y., Sekito, T. et al.: Hierarchy of Atg proteins in pre-autophagosomal structure organization. Genes Cells, 12, 209-218 (2007)[PubMed]
- Kamada, Y., Funakoshi, T., Shintani, T. et al.: Tor-mediated induction of autophagy via an Apg1 protein kinase complex. J. Cell Biol., 150, 1507-1513 (2000)[PubMed]
- Fujioka, Y., Suzuki, S. W., Yamamoto, H. et al.: Structural basis of starvation-induced assembly of the autophagy initiation complex. Nat. Struct. Mol. Biol., 21, 513-521 (2014)[PubMed] [新着論文レビュー]
- Mao, K., Chew, L. H., Inoue-Aono, Y. et al.: Atg29 phosphorylation regulates coordination of the Atg17-Atg31-Atg29 complex with the Atg11 scaffold during autophagy initiation. Proc. Natl. Acad. Sci. USA, 110, E2875-E2884 (2013)[PubMed]
- Ragusa, M. J., Stanley, R. E. & Hurley, J. H.: Architecture of the Atg17 complex as a scaffold for autophagosome biogenesis. Cell, 151, 1501-1512 (2012)[PubMed]
- Kraft, C., Kijanska, M., Kalie, E. et al.: Binding of the Atg1/ULK1 kinase to the ubiquitin-like protein Atg8 regulates autophagy. EMBO J., 31, 3691-3703 (2012)[PubMed]
- Nakatogawa, H., Ohbayashi, S., Sakoh-Nakatogawa, M. et al.: The autophagy-related protein kinase Atg1 interacts with the ubiquitin-like protein Atg8 via the Atg8 family interacting motif to facilitate autophagosome formation. J. Biol. Chem., 287, 28503-28507 (2012)[PubMed]
- Jao, C. C., Ragusa, M. J., Stanley, R. E. et al.: A HORMA domain in Atg13 mediates PI 3-kinase recruitment in autophagy. Proc. Natl. Acad. Sci. USA, 110, 5486-5491 (2013)[PubMed]
- Luo, X. & Yu, H.: Protein metamorphosis: the two-state behavior of Mad2. Structure, 16, 1616-1625 (2008)[PubMed]
- Kabeya, Y., Noda, N. N., Fujioka, Y. et al.: Characterization of the Atg17-Atg29-Atg31 complex specifically required for starvation-induced autophagy in Saccharomyces cerevisiae. Biochem. Biophys. Res. Commun., 389, 612-615 (2009)[PubMed]
- Chew, L. H., Setiaputra, D., Klionsky, D. J. et al.: Structural characterization of the Saccharomyces cerevisiae autophagy regulatory complex Atg17-Atg31-Atg29. Autophagy, 9, 1467-1474 (2013)[PubMed]
- Noda, T. & Ohsumi, Y.: Tor, a phosphatidylinositol kinase homologue, controls autophagy in yeast. J. Biol. Chem., 273, 3963-3966 (1998)[PubMed]
- Kamada, Y., Yoshino, K., Kondo, C. et al.: Tor directly controls the Atg1 kinase complex to regulate autophagy. Mol. Cell. Biol., 30, 1049-1058 (2010)[PubMed]
- Lynch-Day, M. A. & Klionsky, D. J.: The Cvt pathway as a model for selective autophagy. FEBS Lett., 584, 1359-1366 (2010)[PubMed]
- Ohsumi, Y.: Molecular dissection of autophagy: two ubiquitin-like systems. Nat. Rev. Mol. Cell Biol., 2, 211-216 (2001)[PubMed]
- Mizushima, N., Noda, T., Yoshimori, T. et al.: A protein conjugation system essential for autophagy. Nature, 395, 395-398 (1998)[PubMed]
- Mizushima, N., Noda, T. & Ohsumi, Y.: Apg16p is required for the function of the Apg12p-Apg5p conjugate in the yeast autophagy pathway. EMBO J., 18, 3888-3896 (1999)[PubMed]
- Ichimura, Y., Kirisako, T., Takao, T. et al.: A ubiquitin-like system mediates protein lipidation. Nature, 408, 488-492 (2000)[PubMed]
- Kirisako, T., Ichimura, Y., Okada, H. et al.: The reversible modification regulates the membrane-binding state of Apg8/Aut7 essential for autophagy and the cytoplasm to vacuole targeting pathway. J. Cell Biol., 151, 263-276 (2000)[PubMed]
- Hanada, T., Noda, N. N., Satomi, Y. et al.: The Atg12-Atg5 conjugate has a novel E3-like activity for protein lipidation in autophagy. J. Biol. Chem., 282, 37298-37302 (2007)[PubMed]
- Suzuki, K., Akioka, M., Kondo-Kakuta, C. et al.: Fine mapping of autophagy-related proteins during autophagosome formation in Saccharomyces cerevisiae. J. Cell Sci., 126, 2534-2544 (2013)[PubMed]
- Noda, N. N., Ohsumi, Y. & Inagaki, F.: Atg8-family interacting motif crucial for selective autophagy. FEBS Lett., 584, 1379-1385 (2010)[PubMed]
- Sugawara, K., Suzuki, N. N., Fujioka, Y. et al.: The crystal structure of microtubule-associated protein light chain 3, a mammalian homologue of Saccharomyces cerevisiae Atg8. Genes Cells, 9, 611-618 (2004)[PubMed]
- Nakatogawa, H., Ichimura, Y. & Ohsumi, Y.: Atg8, a ubiquitin-like protein required for autophagosome formation, mediates membrane tethering and hemifusion. Cell, 130, 165-178 (2007)[PubMed]
- Noda, N. N., Kumeta, H., Nakatogawa, H. et al.: Structural basis of target recognition by Atg8/LC3 during selective autophagy. Genes Cells, 13, 1211-1218 (2008)[PubMed]
- Ichimura, Y., Kumanomidou, T., Sou, Y. S. et al.: Structural basis for sorting mechanism of p62 in selective autophagy. J. Biol. Chem., 283, 22847-22857 (2008)[PubMed]
- Pankiv, S., Clausen, T. H., Lamark, T. et al.: p62/SQSTM1 binds directly to Atg8/LC3 to facilitate degradation of ubiquitinated protein aggregates by autophagy. J. Biol. Chem., 282, 24131-24145 (2007)[PubMed]
- Suzuki, N. N., Yoshimoto, K., Fujioka, Y. et al.: The crystal structure of plant ATG12 and its biological implication in autophagy. Autophagy, 1, 119-126 (2005)[PubMed]
- Fujita, N., Itoh, T., Omori, H. et al.: The Atg16L complex specifies the site of LC3 lipidation for membrane biogenesis in autophagy. Mol. Biol. Cell, 19, 2092-2100 (2008)[PubMed]
- Metlagel, Z., Otomo, C., Takaesu, G. et al.: Structural basis of ATG3 recognition by the autophagic ubiquitin-like protein ATG12. Proc. Natl. Acad. Sci. USA, 110, 18844-18849 (2013)[PubMed]
- Satoo, K., Noda, N. N., Kumeta, H. et al.: The structure of Atg4B-LC3 complex reveals the mechanism of LC3 processing and delipidation during autophagy. EMBO J., 28, 1341-1350 (2009)[PubMed]
- Kumanomidou, T., Mizushima, T., Komatsu, M. et al.: The crystal structure of human Atg4b, a processing and de-conjugating enzyme for autophagosome-forming modifiers. J. Mol. Biol., 355, 612-618 (2006)[PubMed]
- Sugawara, K., Suzuki, N. N., Fujioka, Y. et al.: Structural basis for the specificity and catalysis of human Atg4B responsible for mammalian autophagy. J. Biol. Chem., 280, 40058-40065 (2005)[PubMed]
- Hong, S. B., Kim, B. W., Lee, K. E. et al.: Insights into noncanonical E1 enzyme activation from the structure of autophagic E1 Atg7 with Atg8. Nat. Struct. Mol. Biol., 18, 1323-1330 (2011)[PubMed]
- Taherbhoy, A. M., Tait, S. W., Kaiser, S. E. et al.: Atg8 transfer from Atg7 to Atg3: a distinctive E1-E2 architecture and mechanism in the autophagy pathway. Mol. Cell, 44, 451-461 (2011)[PubMed]
- Noda, N. N., Satoo, K., Fujioka, Y. et al.: Structural basis of Atg8 activation by a homodimeric E1, Atg7. Mol. Cell, 44, 462-475 (2011)[PubMed] [新着論文レビュー]
- Yamada, Y., Suzuki, N. N., Hanada, T. et al.: The crystal structure of Atg3, an autophagy-related ubiquitin carrier protein (E2) enzyme that mediates Atg8 lipidation. J. Biol. Chem., 282, 8036-8043 (2007)[PubMed]
- Yamaguchi, M., Noda, N. N., Yamamoto, H. et al.: Structural insights into Atg10-mediated formation of the autophagy-essential Atg12-Atg5 conjugate. Structure, 20, 1244-1254 (2012)[PubMed]
- Hong, S. B., Kim, B. W., Kim, J. H. et al.: Structure of the autophagic E2 enzyme Atg10. Acta Crystallogr. D. Biol. Crystallogr., 68, 1409-1417 (2012)[PubMed]
- Yamaguchi, M., Noda, N. N., Nakatogawa, H. et al.: Autophagy-related protein 8 (Atg8) family interacting motif in Atg3 mediates the Atg3-Atg8 interaction and is crucial for the cytoplasm-to-vacuole targeting pathway. J. Biol. Chem., 285, 29599-29607 (2010)[PubMed]
- Nath, S., Dancourt, J., Shteyn, V. et al.: Lipidation of the LC3/GABARAP family of autophagy proteins relies on a membrane-curvature-sensing domain in Atg3. Nat. Cell Biol., 16, 415-424 (2014)[PubMed]
- Hanada, T., Satomi, Y., Takao, T. et al.: The amino-terminal region of Atg3 is essential for association with phosphatidylethanolamine in Atg8 lipidation. FEBS Lett., 583, 1078-1083 (2009)[PubMed]
- Fujioka, Y., Noda, N. N., Nakatogawa, H. et al.: Dimeric coiled-coil structure of Saccharomyces cerevisiae Atg16 and its functional significance in autophagy. J. Biol. Chem., 285, 1508-1515 (2010)[PubMed]
- Matsushita, M., Suzuki, N. N., Obara, K. et al.: Structure of Atg5•Atg16, a complex essential for autophagy. J. Biol. Chem., 282, 6763-6772 (2007)[PubMed]
- Noda, N. N., Fujioka, Y., Hanada, T. et al.: Structure of the Atg12-Atg5 conjugate reveals a platform for stimulating Atg8-PE conjugation. EMBO Rep., 14, 206-211 (2013)[PubMed]
- Otomo, C., Metlagel, Z., Takaesu, G. et al.: Structure of the human ATG12~ATG5 conjugate required for LC3 lipidation in autophagy. Nat. Struct. Mol. Biol., 20, 59-66 (2013)[PubMed]
- Yamaguchi, M., Matoba, K., Sawada, R. et al.: Noncanonical recognition and UBL loading of distinct E2s by autophagy-essential Atg7. Nat. Struct. Mol. Biol., 19, 1250-1256 (2012)[PubMed] [新着論文レビュー]
- Kaiser, S. E., Mao, K., Taherbhoy, A. M. et al.: Noncanonical E2 recruitment by the autophagy E1 revealed by Atg7-Atg3 and Atg7-Atg10 structures. Nat. Struct. Mol. Biol., 19, 1242-1249 (2012)[PubMed]
- Sakoh-Nakatogawa, M., Matoba, K., Asai, E. et al.: Atg12-Atg5 conjugate enhances E2 activity of Atg3 by rearranging its catalytic site. Nat. Struct. Mol. Biol., 20, 433-439 (2013)[PubMed] [新着論文レビュー]
著者プロフィール
略歴:2001年 東京大学大学院薬学系研究科博士課程 修了,同年 北海道大学大学院薬学研究科 博士研究員,同 助教,同講師を経て,2011年より微生物化学研究会微生物化学研究所 主席研究員.
研究テーマ:オートファジーの分子機構.
関心事:オートキャンプ.
稲垣 冬彦(Fuyuhiko Inagaki)
北海道大学大学院先端生命科学研究院 特任教授.
研究テーマ:核磁気共鳴,構造生物学,シグナル伝達,オートファジー.
© 2014 野田展生・稲垣冬彦 Licensed under CC 表示 2.1 日本