オートファジーを長き眠りからめざめさせた酵母
2012/09/19
荒木保弘・大隅良典
(東京工業大学フロンティア研究機構)
email:荒木保弘,大隅良典
領域融合レビュー, 1, e005 (2012) DOI: 10.7875/leading.author.1.e005
Yasuhiro Araki & Yoshinori Ohsumi: Awakening the hibernation of autophagy research using yeast.

オートファジーは,タンパク質など細胞質成分のみならずオルガネラのような巨大な構造体を丸ごと分解する,真核生物に広く保存されたバルク分解系である.オートファジーの概念は最初に哺乳動物の系から提唱されたが,その分子実体が明らかになるのに40年もの年月を要した.このブレイクスルーは,もっともシンプルな真核生物のモデル系である出芽酵母によるものであった.ここでは,出芽酵母がどのようにオートファジー研究に飛躍をもたらしたかを,これまで筆者らが得たオートファジーの分子機構に関する知見を中心に解説する.
オートファジーとは,細胞の自己成分を細胞内の消化器官であるリソソームあるいは液胞に輸送し分解する,酵母からヒトまで真核生物に広く保存された一連の過程をさす.当初,想定された栄養飢餓の際の生体物質のリサイクル系としての機能だけでなく,最近では,発生と分化,がんや神経変性疾患の抑制,老化,免疫応答,抗原提示,細胞死,病原体の排除など,オートファジーのかかわる生命現象は枚挙にいとまがない1).このように,オートファジーのテリトリーが急速に拡張したのはここ10年のことであることから,オートファジーを新しい学問領域だと思っている人は多いのではないだろうか.オートファジーの概念が提唱されたのはいまからほぼ50年前のことであり,これは,もうひとつの細胞内タンパク質分解系であるユビキチン-プロテアソーム系の発見より25年も前になる.しかしながら,オートファジーは誕生からほどなく表舞台から消えていった2).最近になり,ふたたび脚光をあびるようになったのは,出芽酵母Saccharomyces cerevisiaeを用いた研究を端緒とするものである.ここでは,出芽酵母がどのようにオートファジー研究の推進に寄与したのかを,オートファジー研究の歴史的な推移,また,オートファジーの分子機構に関する知見をまじえ紹介する.これまで,3種類のオートファジー,ミクロオートファジー,マクロオートファジー,シャペロン介在オートファジーが知られているが,ここではそのうち,主要な経路でありもっとも知見の蓄積しているマクロオートファジーをとりあげ,以降,これを単にオートファジーとよぶ.
近年,“動的平衡”という言葉をよく耳にすることもあり,現代のわれわれは自分のからだを構成している物質は合成と分解との平衡状態にあることを知っている.生体構成物質,とくにタンパク質のターンオーバーの概念は,ちょうど70年前の1942年,生物に対しはじめて同位体標識を試みたSchoenheimerによりもたらされた3).しかし,タンパク質の合成に関する知見の膨大な蓄積に対し,もう一方の分解についての知見は皆無なままであった.
タンパク質分解の概念が具現化したのは,1950年代から1960年代,米国Rockefeller大学においてであった.当時,電子顕微鏡により細胞の微細構造が観察されるようになり,その微細構造体を細胞分画により分離して生化学的な方法で機能を明らかにするという細胞生物学がまさにここで花開きつつあった.こうしたなか,1953年から1955年にかけて,de Duveにより多様な加水分解酵素を含むオルガネラとしてリソソームが発見された4,5).de Duveは,この発見により1974年,Rockefeller大学の同僚であったPalade,Claudeとともにノーベル医学生理学賞を受賞している.加水分解酵素が細胞質に散在することは生物にとり危険きわまりないことは容易に想像され,これらをリソソームという膜コンパートメントに隔離することは非常に理にかなっていたが,同時に,分解されるべき基質をどのようにこのオルガネラに輸送しているのかが問題になった.1960年前後,ミトコンドリアや小胞体といったオルガネラやリボソームなど細胞質成分を含む小胞が電子顕微鏡により観察された.さらに,内容物がリソソームのもつ加水分解酵素により消化されつつある小胞の像が得られた6).de Duveは,細胞質成分が小胞としてリソソームと融合し分解される一連の過程をオートファジー,そして,その小胞をオートファゴソームとはじめて命名した.いまからほぼ50年前の1963年のことである7).その翌年には,栄養飢餓によりオートファジーの亢進されることが観察され,オートファジーは自己の細胞質成分をリソソームにおいて分解し,アミノ酸などの生体内物質をリサイクルする機構であると考えられた.しかしながら,この時点ではオートファジーは細胞におけるタンパク質分解マシナリーとしての地位をゆるぎないものとするまではいたらなかった.おのおののタンパク質が固有の半減期をもつという事実と,基質をバルクとしてリソソームにおいて非選択的に分解するオートファジーは概念的にあいいれなかった.さらに,反証する結果がde DuveのいたRockefeller大学のPooleらによりもたらされた8).クロロキンやイオノホアX537Aの処理によりリソソームのもつ加水分解酵素を阻害すると,ファゴサイトーシスにより細胞の外から取り込まれたタンパク質の分解は抑制されたものの,恒常的に起こっている生体でのタンパク質分解にはほとんど影響がみられなかったのである.
この発表がなされた1977年には,ユビキチン-プロテアソーム系の扉が開かれた.1964年,Rabinovitzにより,分化の途上でリソソームを消失する網状赤血球においても,タンパク質分解は促進されていることが判明していた9).網状赤血球で観察されたリソソームに依存しないタンパク質分解系,これこそが,ユビキチン-プロテアソーム系にほかならなかった.ユビキチン-プロテアソーム系の発見はリソソームおよびオートファジーの研究の副次的な産物であったことになる.1977年,Goldbergが網状赤血球抽出液を用いてATPに依存性のタンパク質分解無細胞系を確立し10),1978年,Herskoらは,さらに分画に着手した11).ユビキチン-プロテアソーム系の誕生である.1990年代に入るころにはユビキチン化反応の全貌が明らかにされ,さらに,ユビキチン化されたタンパク質を分解する本体であるプロテアソームが同定されるなど,ユビキチンはタンパク質分解シグナルとして機能し,ユビキチン化されたタンパク質はプロテアソームにより選択的に分解される,というスキームが確立した.さらに,ユビキチン-プロテアソーム系は,細胞周期やDNA損傷応答,転写,シグナル伝達など,生命現象の実に多様な局面にかかわっていることが明らかになり,細胞におけるタンパク質分解系としてユビキチン-プロテアソーム系が席巻した.ユビキチン-プロテアソーム系は無細胞系から研究がはじまり,ほどなくin vitroにおける完全再構成系が構築されるなど,生化学的な手法によりその分子機構にせまることができ,また,その過程で分子実体が同定されると,その遺伝子の同定,逆遺伝学による機能解析など,分子生物学的な手法によるアプローチがスムーズに遂行された.一方,オートファジーにかかわる遺伝子やタンパク質についてほとんど研究に進展がみられなかったのは,電子顕微鏡による観察がオートファゴソームを検出する唯一の手段であったことが大きな要因であった.オートファジーに特異的に関与する遺伝子がはじめてクローニングされたのは1996年であったが,その研究対象となったのは,それまでオートファジーの研究に使われてきた哺乳動物ではなく,誰にもかえりみられることのなかったもっともシンプルな真核生物のモデル系,出芽酵母であった12).
1992年,大隅良典らは,出芽酵母においてオートファジーをはじめて観察した13).液胞は真菌から高等植物まで存在するオルガネラである.細胞において最大のコンパートメントであり,出芽酵母では細胞の全体積の25%以上をしめる.液胞には多数の加水分解酵素が内在していることから,古くから細胞における分解コンパートメントであると考えられてきた.液胞に存在する加水分解酵素の活性は栄養状態により変動し,とくに栄養飢餓により誘導される.また,出芽酵母において液胞の加水分解酵素を欠損した株は富栄養状態においては顕著な表現型を示さないが,栄養飢餓により誘導される胞子形成が不全になる.このことから,液胞は栄養飢餓状態に対し重要な生理機能をもつことが示唆されていた.栄養飢餓培地に移したタンパク質分解酵素欠損株の形態変化を光学顕微鏡で観察することにより,液胞に球形の構造体が認められた.野生株では希有なこの構造体は,液胞に取り込まれたものの,タンパク質分解酵素の欠損のため分解されずに蓄積した小胞であった.電子顕微鏡でのさらなる観察により,これは細胞質成分をそのまま含む一重膜の構造体であることが示され,オートファジックボディーと名づけられた.また,細胞質成分をとりかこんださまざまな二重膜構造を細胞質において観察するにいたり,以下のオートファゴソーム形成の構図がうかびあがった(図1).栄養飢餓に応答して細胞質において隔離膜と名づけられた嚢状の膜構造が出現し,膜の伸張とともに細胞質成分をとりかこむ.そののち,最終形態であるオートファゴソームの外膜が液胞膜と融合することにより,内膜にとりかこまれた内容物は液胞の内部へと放出される.液胞がリソソームに比べはるかに大きいことを除けば,出芽酵母でみられたこの一連の膜動態は,まさにde Duveの提唱したオートファジーそのものであった.
この出芽酵母におけるオートファジーの発見が,オートファゴソームの形成をつかさどる分子実体の解明を一気に押し進めることになった.この推進の要因は2点に集約される.1つ目に,この時期の出芽酵母の研究は,従来の変異体の分離,遺伝解析といった古典的な遺伝学にくわえ,遺伝子クローニング,逆遺伝学といった分子生物学的な解析の技術基盤が確立していただけでなく,真核生物においてはじめてのゲノムプロジェクトとしてゲノム全塩基配列の解読が精力的になされていて,ゲノム情報の利用が可能になっていた.出芽酵母ゲノムプロジェクトの完了は1997年である.2つ目として,動物細胞におけるオートファジーは電子顕微鏡でしかとらえられなかったのに対し,出芽酵母のオートファジーは液胞酵素の不活性株を用いることで位相差顕微鏡によりオートファジックボディーの蓄積として容易にとらえることができた.
ひきつづいて1993年,大隅らは出芽酵母のオートファジー不全変異体を分離することに成功した14).まず,液胞酵素欠損株にみられるはずのオートファジックボディーが液胞に蓄積しない変異株を光学顕微鏡での観察により単離した.このとき,唯一,得られたのがapg1変異株であった.このAPG1遺伝子は現在ではATG1遺伝子にあたる(後述).このapg1変異株は栄養飢餓状態では急速に生存率の低下することがわかり,栄養飢餓状態での生存率の低下とオートファジックボディーの形成不全の2つを指標としたスクリーニングにより,apg1変異株にくわえて,さらに13の相補群に分類されるオートファジー不全変異体を分離した.そして,これらの変異体からオートファジーに必須の14個の遺伝子が同定された.これらは1つを除きすべてが新規の遺伝子であり,オートファジーがこれまでにない分子機構で駆動されていることが予想された.オートファジー不全変異体は,栄養飢餓がシグナルへと変換し伝達される段階,オートファゴソーム形成の段階,オートファゴソームが液胞と融合し内容物が分解される段階,のいずれかに異常を生じていると考えられた.同定されたすべての遺伝子を欠損した株では電子顕微鏡ではオートファゴソームがまったく観察されなかったことから,これらの遺伝子はオートファゴソーム形成の段階,もしくは,それ以前に関与しているものと考えられた.さらに2001年には,GFPとの融合タンパク質を用いることで,これら遺伝子のほぼすべての産物は栄養飢餓状態に応答して液胞の近傍の一点に局在することが明らかになった15).この構造体は蛍光顕微鏡でのみ観察され電子顕微鏡ではいまだとらえられていないなど,その実体の詳細は不明であるもののオートファゴソームの形成の場であると考えられ,PAS(pre-autophagosomal structure,プレオートオートファゴソーム構造体)とよばれる.
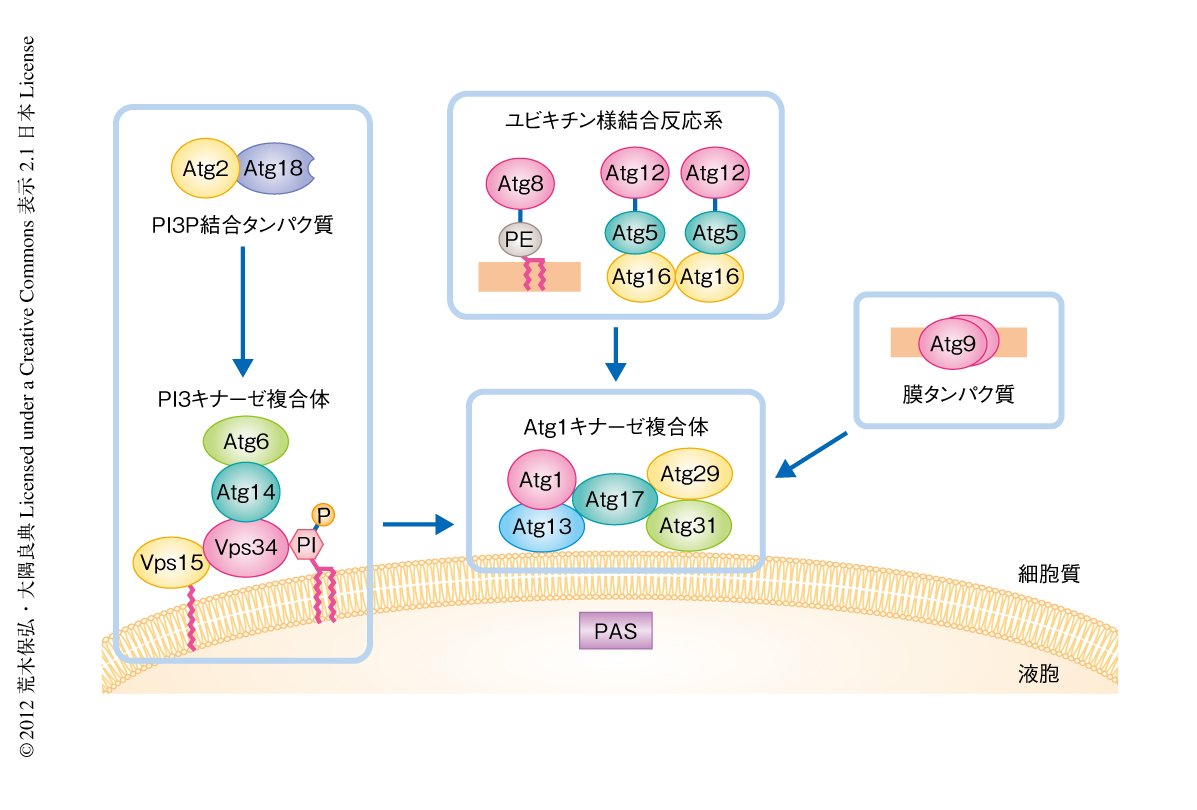
2004年には,おもにThummらにより分離されたaut変異株や16),Klionskyらにより分離されたcvt変異株17),さらに,そのほかの出芽酵母オートファジー関連遺伝子をあわせて,名称がATG(autophagy-related)遺伝子に統一された18).現在では30をこえるATG遺伝子が見い出されている.そのうち,最初に発見された14個にくわえ,合計18個(Atg1~Atg10,Atg12~Atg14,Atg16~Atg18,Atg29,Atg31)がオートファゴソームの形成に必須の遺伝子とされている.これまでの解析により,これら遺伝子の産物であるオートファゴソームの形成に必須な18個のタンパク質(以下,“Atgタンパク質”とよぶ)は,いくつかの機能単位を構成していることが明らかになっている19)(図2).以下,この機能単位ごとに,オートファゴソーム形成の分子機構を,筆者らのこれまで得た知見を中心に解説する.
1)Atg1キナーゼ複合体
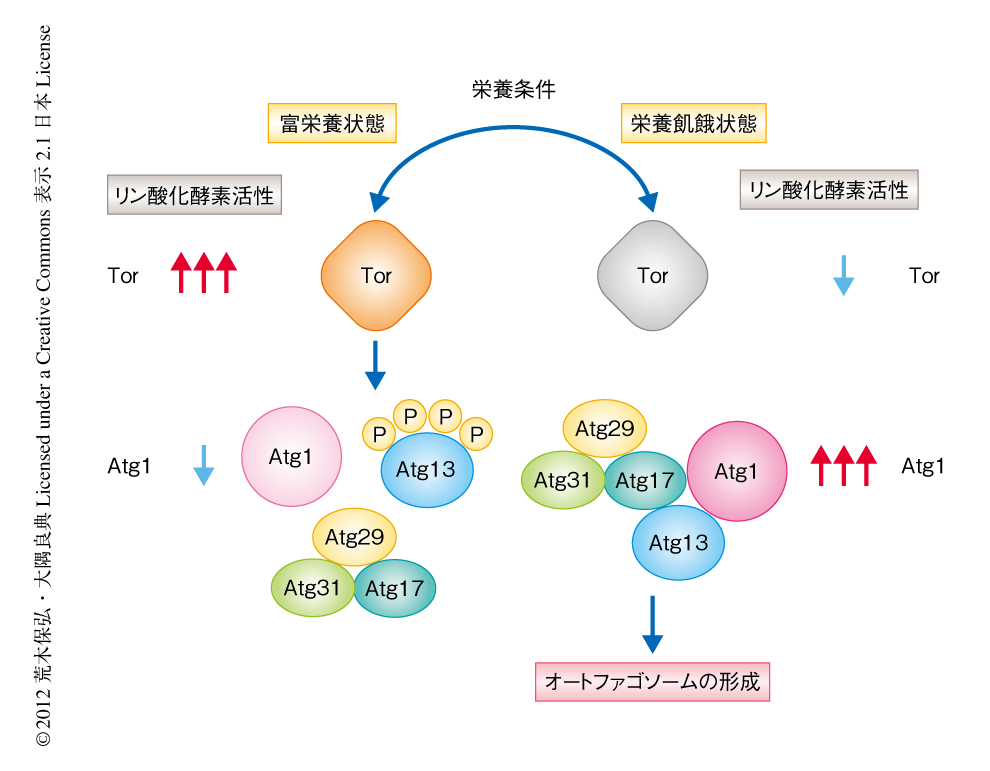
Atgタンパク質について,その1次構造から得られる情報はほとんど皆無であった.例外のひとつがAtg1である.ATG1遺伝子は1997年にクローニングされ,その1次構造からAtg1はリン酸化酵素(キナーゼ)であることがわかった.この酵素の過剰発現により出芽酵母のatg13変異株のオートファジー活性が回復したことからAtg1とAtg13の機能的な関連性が示唆されたが,その機構は不明のままであった.ここに,“生物が細胞内外の栄養状態をどのように感知しているか”という生物学において大きな問題がかかわってきた.1991年,免疫抑制剤ラパマイシンの生体における標的タンパク質としてTorが同定され,ラパマイシンはTorの特異的な阻害剤として作用することが実証された.Torは脂質リン酸化酵素に相同性をもっていたがその基質は不明であった.ラパマイシン処理またはTorへの変異導入によりTorを不活性化すると,G1期の初期において細胞周期が停止するなど栄養飢餓のときと同じ挙動が観察され,Torは栄養状態に応じた細胞の増殖制御を担っていると考えられた.1998年,Torの不活性化にともない富栄養状態においてもオートファジーの誘導されることが明らかになり,富栄養状態ではTorがオートファジーを抑制していることがわかったが,Torによりリン酸化される基質は不明なままであった20).2000年,これらの疑問が解決された.富栄養状態では高度にリン酸化されているAtg13がラパマイシン処理により脱リン酸化型に変換され,Atg1と複合体を形成すること,それにともないAtg1のリン酸化酵素活性の亢進することが示されたのである21).Atg13がTorに依存してリン酸化されることでオートファジーが抑制され,栄養飢餓のときには脱リン酸化型Atg13とAtg1からなるAtg1キナーゼ複合体が形成される,そして,Atg1のもつリン酸化酵素活性の亢進がオートファジー誘導のもっとも初期段階である,というスキームができた(図3).2010年には,Torによるリン酸化をうけない変異型のAtg13をもつ出芽酵母ではオートファジーが恒常的に活性化されていたことから,オートファジーの抑制の際のTorのおもな基質はAtg13であることが確認された22).Atg1のリン酸化酵素活性はオートファジーに必須であるが,その基質はまだ明らかになっていない.近年,栄養飢餓状態において,Atg1キナーゼ複合体にはAtg13にくわえ,Atg17,Atg29,Atg31が含まれ,これらを欠損した株ではAtgタンパク質のPASへの局在が失われたことから,Atg1キナーゼ複合体はPASのもっとも基部になっていると考えられる23).
2)PI3キナーゼ複合体とそのエフェクタータンパク質
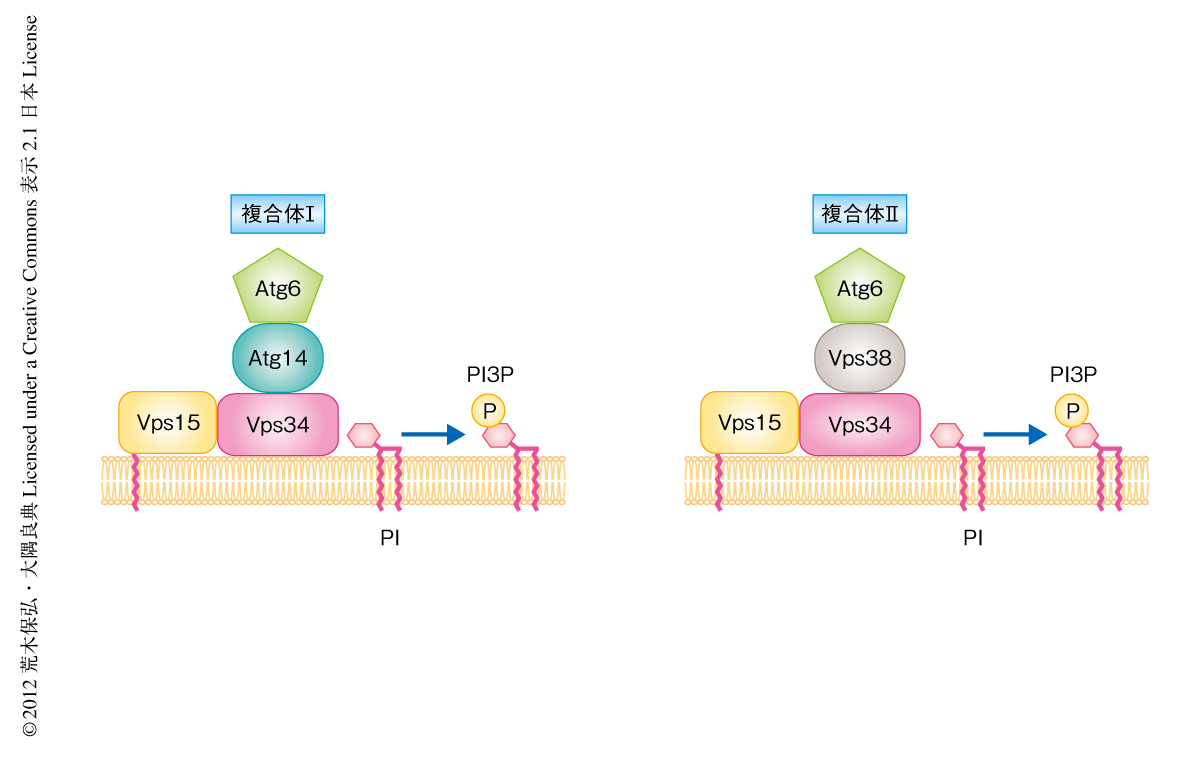
1998年にクローニングされたATG6遺伝子は,オートファゴソームの形成に必須な18個の遺伝子のなかで唯一,既知の遺伝子であり,Emrらにより,カルボキシペプチダーゼYが本来の液胞に局在せず細胞外に分泌されるvps(vacuolar protein sorting)変異体の責任遺伝子のひとつとして分離されVPS30遺伝子と名づけられていた.出芽酵母のatg6変異株はオートファジーと液胞酵素の輸送に異常を生じた.しかし,同時に遺伝子クローニングの報告されたAtg14は,栄養状態に関係なくつねにAtg6と複合体を形成するにもかかわらず,Atg14の過剰発現ではatg6変異株のオートファジー不全のみが抑圧され,かつ,atg14変異体はオートファジーにのみ異常を生じた24).これは,同じ複合体の構成タンパク質であるにもかかわらず,変異体の表現型は異なるという不可解な結果であった.2001年,Atg6と相互作用するタンパク質として新たにVps15,Vps34,Vps38が同定された.さらなる生化学的な解析から,Atg6は2つの複合体,すなわち,Vps15,Vps34,Atg6,Atg14からなる複合体Iと,Vps15,Vps34,Atg6,Vps38からなる複合体IIを形成しており,複合体Iがオートファジーに,複合体IIが液胞酵素の輸送に寄与していることが判明した25)(図4).この2つの複合体に共通する構成タンパク質であるAtg6の変異体が2つの異常を示すのは当然であった.Vps34は生体膜リン脂質の一種,ホスファチジルイノシトール3-リン酸を合成するPI3キナーゼであった.その活性はオートファジーに必須であり,産生されるホスファチジルイノシトール3-リン酸は下流のエフェクタータンパク質であるAtg2を,ホスファチジルイノシトール3-リン酸との結合能をもつAtg18との複合体としてリクルートした26).しかし,Atg18のPASへの局在はAtg2に依存していたこと,atg18欠損株においてAtg2を別のホスファチジルイノシトール3-リン酸結合ドメインとの融合タンパク質により人工的にPASへと局在させてもオートファジーに異常を示したことから,Atg18のホスファチジルイノシトール3-リン酸との結合能とは別の分子機能がオートファゴソームの形成に必須であると考えられた.Atg2は約1600アミノ酸残基からなる巨大なタンパク質だが,既知の機能ドメインがなくその機能の詳細は不明である.PASへの局在におけるほかのAtgタンパク質への依存性から考えてオートファゴソーム形成の最終段階に機能することが示唆され,Atg2の機能,すなわち,Atg2の未知の酵素活性やさらなる下流タンパク質を探ることが,隔離膜の脂質はどこからどのように供給されるのか,膜の伸張や曲がりはどのようになされるのか,といったオートファゴソーム形成の本質的な疑問の解決に直結すると考えられる.
3)膜タンパク質Atg9
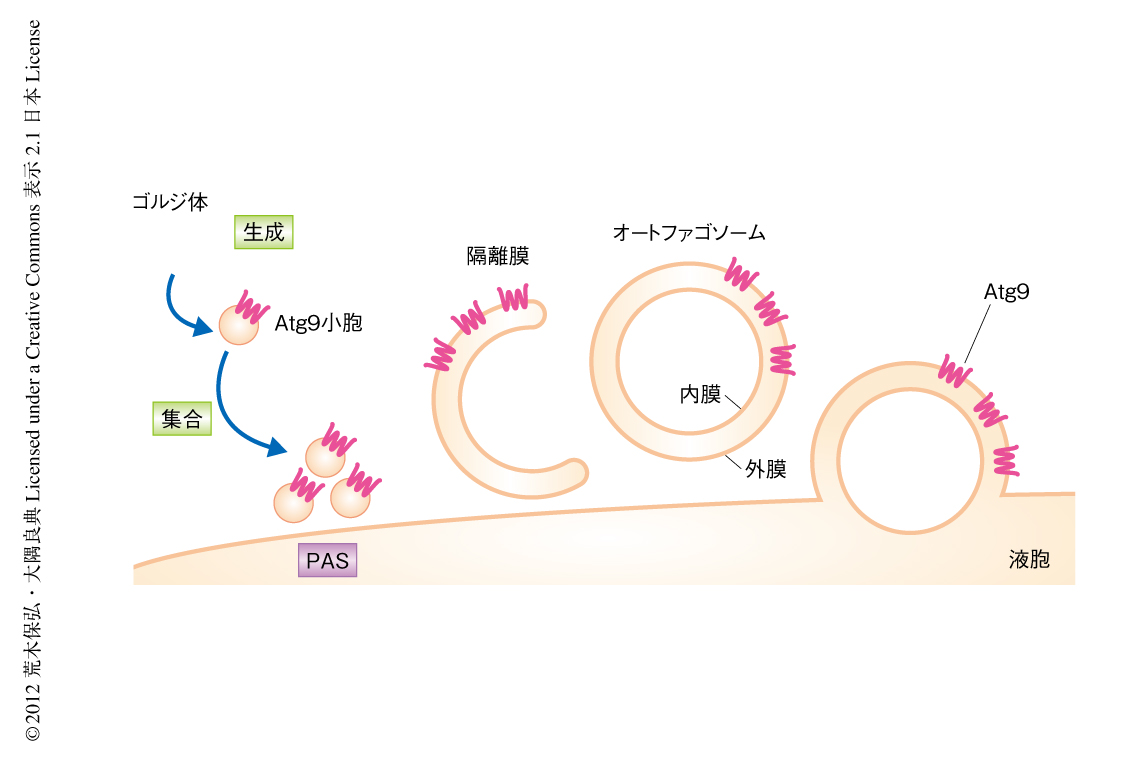
Atg9はAtgタンパク質のなかで唯一,膜貫通領域をもつ膜タンパク質であり,長いあいだ,オートファゴソームの形成において脂質の供給源であると考えられてきた.ごく最近,細胞質を激しくうごめく約30分子のAtg9が膜に存在する30~60 nmの小胞の存在がとらえられた27).蛍光顕微鏡による定量解析から,栄養飢餓に応じPASには平均3個のAtg9小胞しかリクルートされず,それ以上のAtg9小胞の流入はみられないことから,Atg9は脂質の供給源として膜の伸張に関与するより,オートファゴソーム形成の核として初期段階に機能しているようである(図5).Atg9は最終的にオートファゴソームの外膜に局在する.
4)オートファジーに内包される2つのユビキチン様結合反応系
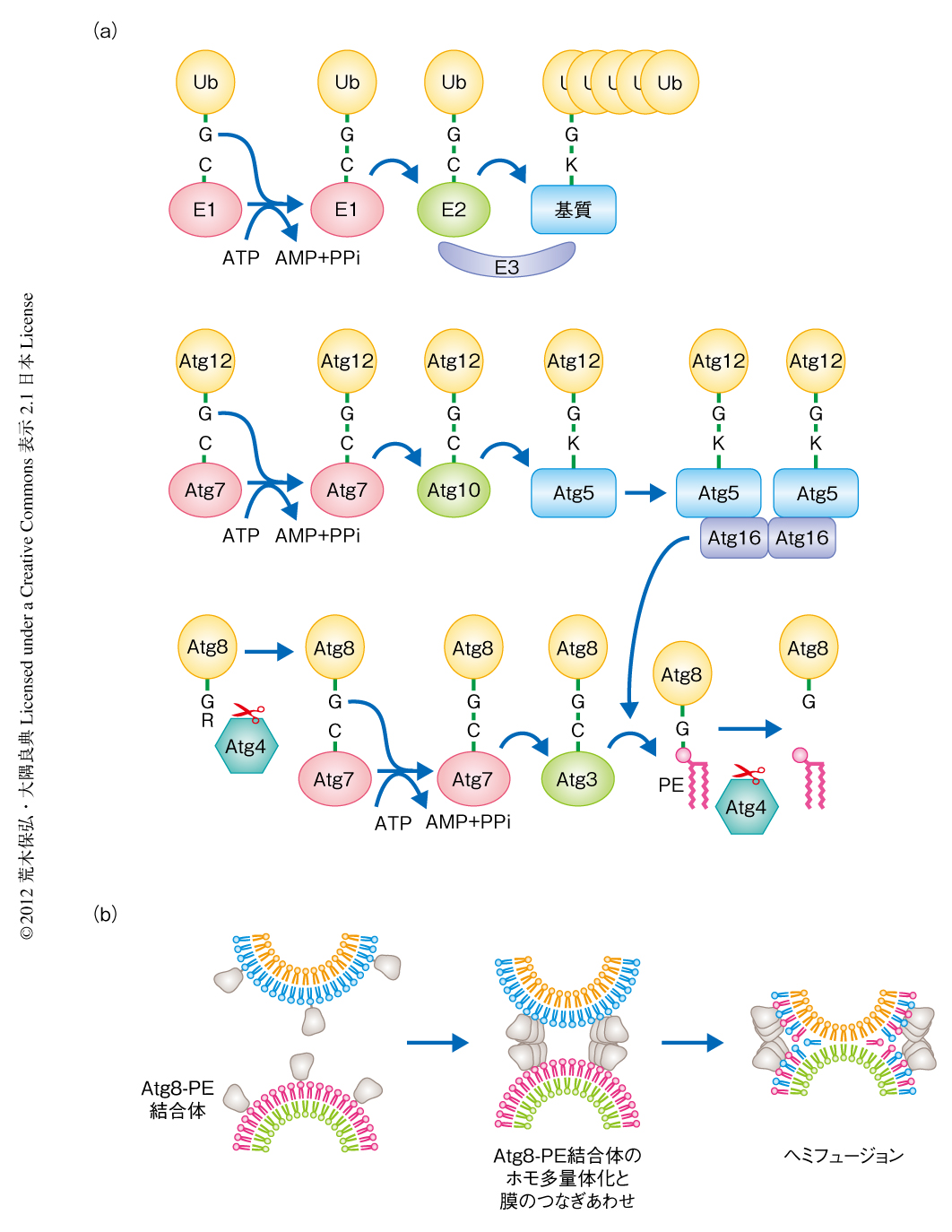
オートファジー研究においてもっとも驚きであったのは,Atgタンパク質にユビキチン様結合反応系が2つも含まれることであったといっても過言ではない.ユビキチン結合反応系とは,ユビキチン活性化酵素(E1),ユビキチン結合酵素(E2),ユビキチンリガーゼ(E3)の3種の酵素のはたらきにより,ATPに依存的に,ユビキチンのC末端のグリシン残基と標的タンパク質のリジン残基のεアミノ基とのイソペプチド結合が形成される系である(図6a).
1998年,Atg12結合反応系が明らかになった28).186アミノ酸残基からなるAtg12は,ウェスタンブロットによりAtg12自体のほかに,40 kDほど大きいバンドが認められた.Atg5もウェスタンブロットにより同様に2本のバンドが認められ,高分子量のバンドはAtg12のウェスタンブロットにより得られたバンドと一致した.最終的に,この高分子量のバンドは,Atg12のC末端のアミノ酸残基がAtg5の149番目のリジン残基と共有結合したものであることが判明した.Atg12のC末端のグリシン残基の欠失,あるいは,Atg5の149番目のリジン残基の変異により,この結合体の形成不全とオートファジー不全が示されたことから,Atg5を標的としたAtg12によるユビキチン様結合反応系がオートファジーに必須であると考えられた.ついで,E1,E2,E3の実体が問題となった.出芽酵母のatg7変異株およびatg10変異株ではAtg12-Atg5結合体は消失した.ユビキチン様結合反応系の特徴であるチオエステル結合がAtg12とAtg7のあいだ,および,Atg12とAtg10のあいだでみられたこと,Atg7がユビキチン活性化酵素Uba1と相同性をもっていたこと,Atg12-Atg7結合体はATG10遺伝子に依存しないがAtg12-Atg10結合体はATG7遺伝子に依存したことから,Atg7がE1,Atg10がE2であることが判明した(図6a).E3は未同定であるが,in vitroにおける再構成系においてAtg7とAtg10のみでAtg12-Atg5結合体が高効率で生成されたことから,E3はこの系には必要ないと考えられている.Atg12結合反応系には逆反応,すなわち,脱Atg12化酵素の存在しないことも,ほかのユビキチン様結合反応系とは異なる点であった.Atg12はC末端がグリシン残基である成熟型として遺伝子にコードされていた.また,Atg12-Atg5結合体はAtg16と相互作用し,Atg16との結合を介し二量体として存在した.この二量体の形成はPASへの局在に必須であった.
Atg8がもうひとつのユビキチン様結合反応系にかかわることは,2000年に判明した.Atg8のC末端側にタグを導入すると,これは生体においてATG4遺伝子に依存的に除去された.精製標品を用いた実験から,Atg4はシステインプロテアーゼであり,Atg4によりAtg8のC末端のアルギニン残基が除去されグリシン残基の露出されることが明らかになった29).出芽酵母のatg4変異株におけるオートファジー不全が,C末端のアルギニン残基を欠きグリシン残基となったAtg8の変異体により一部抑圧されたことから,Atg8のC末端の露出したグリシン残基が重要であること,また,Atg12との1次構造における相同性からAtg8はユビキチン様タンパク質であることが予想された.免疫電子顕微鏡法によりAtg8のオートファゴソームへの局在が観察されたが,これに一致するように,生化学的な解析においてもAtg8は膜画分に存在した.atg4変異株やC末端のグリシン残基を欠損したAtg8の変異体ではAtg8は膜と相互作用できなかったことから,Atg8の生体における標的物質は膜に強く結合するものであると考えられ,精力的に探索された.その結果,高濃度の尿素の存在のもとでのウェスタンブロットによりごくわずかな移動度の差を示す2本のバンドが認められたが,この時点では標的物質は不明であった.同じ年,移動度の大きなバンドはホスファチジルエタノールアミンの頭部のアミノ基とAtg8のグリシン残基とがアミド結合を形成したものであることが明らかになった30).これは,現在までに知られているユビキチン様タンパク質のうち,タンパク質ではなく脂質を標的とする唯一の例である.Atg8-ホスファチジルエタノールアミン結合体はatg7変異体およびatg3変異体において消失し,Atg8とAtg7のあいだ,および,Atg8とAtg3のあいだにチオエステル結合がみられたこと,Atg3は活性化システイン残基の近傍においてAtg10と相同であったことから,Atg7がE1,Atg3がE2であることが明らかになった(図6a).Atg8とAtg12はおのおの異なるE2をもつにもかかわらず,共通のE1としてAtg7を用いるという特異な反応機構をもっていた.精製したAtg12-Atg5結合体をAtg8の再構成反応系に添加するとAtg8のホスファチジルエタノールアミン化が亢進した.2007年には,Atg8のオートファゴソーム形成における機能の一端が明らかになった31).in vitroにおける再構成反応系において,Atg8のホスファチジルエタノールアミン化にともない,ホスファチジルエタノールアミンを含むリポソームが凝集体を形成した.Atg8-ホスファチジルエタノールアミン結合体はホモ多量体を形成していたことから,この多量体化により膜どうしをつなぎあわせる機能をもつものと考えられた.さらに,この凝集した小胞の膜が,脂質二重層のうち外側の一層のみが融合するヘミフュージョンを誘起していることがわかった(図6b).in vitroにおいてリポソームの凝集やヘミフュージョンの起こらないあるいは低下しているAtg8の変異体を分離したところ,これらの変異体をもつ出芽酵母ではそのin vitroにおける活性に相関して,オートファジックボディーができないか,できても小型化しオートファジーに異常を生じたことから,Atg8は細胞においてもオートファゴソームの形成の際の膜の伸張や最終段階における隔離膜の末端の融合に寄与していると考えられた.
哺乳動物においてはじまったオートファジーの研究は,長い停滞期をへて,出芽酵母においてその分子実体が解明された.Atgタンパク質は生物種をこえて進化的に広く保存されており,そのホモログをノックアウトまたはノックダウンすることにより,哺乳動物におけるオートファジーが再検証された32).その結果,生体物質のリサイクルという基本的な役割のほかにも実に多様な生命現象への関与が明らかになり,現在,哺乳動物のオートファジー研究は興隆をきわめている.しかし,Atgタンパク質がどのように有機的に連携してオートファゴソームの形成にいたるのかというオートファジーにおけるもっとも重要な問題は,出芽酵母においてもほとんど解明されていない.
その大きな障壁のひとつとして,オートファゴソーム形成の素過程を解明するための中間体が観察されていないことがあげられる.出芽酵母のオートファゴソーム形成必須遺伝子の変異株ではすべて,電子顕微鏡による観察ではオートファゴソームの形成がみられず,オートファゴソーム形成の進行の途中で停止したような膜中間体を検出できていない.この現状を打破するには,in vitroにおけるオートファゴソーム形成の再構成系の構築がさけてとおれないであろう.必要となるタンパク質や脂質などが多岐にわたり非常な困難が予想される一方で,現在までの網羅的なスクリーニングにもかかわらずオートファゴソーム形成必須遺伝子は18から増えていないという事実から,典型的なAtgタンパク質はすべて単離されていると考えられる.既知のAtgタンパク質とその関連タンパク質,および,脂質を混合することで,あんがい簡単にオートファゴソーム様の脂質二重膜からなる小胞の出現が観察されるかもしれない.これには,オートファゴソームの脂質組成の解明が必須であろう.これまで膜の融合においては,SNAREを用いた融合,液胞の融合33),エンドソーム膜の融合など34),in vitro完全再構成系の成功例が散見される.オートファゴソームは二重膜であり,さらに,その形成過程は膜の伸張,変形,融合からなるため,その複雑さはこれらとは比較にならない.しかし,いったんin vitroにおける再構成系が構築されれば,そこから得られる恩恵は計りしれないものになるであろう.
略歴:2001年 東京大学大学院薬学系研究科博士課程 修了,同 助手,英国Paterson Institute for Cancer Research研究員,ドイツHeidelberg大学ZMBH研究員,東京工業大学先進研究機構 先進研究員を経て,2011年より東京工業大学フロンティア研究機構 特任助教.
研究テーマ:オートファジー,とくに,オートファゴソーム形成の分子機構.論理的かつ厳密な出芽酵母ならではの研究によりオートファジーを理解したい.
大隅 良典(Yoshinori Ohsumi)
東京工業大学フロンティア研究機構 特任教授.
研究室URL:http://www.ohsumilab.aro.iri.titech.ac.jp/
© 2012 荒木保弘・大隅良典 Licensed under CC 表示 2.1 日本
(東京工業大学フロンティア研究機構)
email:荒木保弘,大隅良典
領域融合レビュー, 1, e005 (2012) DOI: 10.7875/leading.author.1.e005
Yasuhiro Araki & Yoshinori Ohsumi: Awakening the hibernation of autophagy research using yeast.
要 約
オートファジーは,タンパク質など細胞質成分のみならずオルガネラのような巨大な構造体を丸ごと分解する,真核生物に広く保存されたバルク分解系である.オートファジーの概念は最初に哺乳動物の系から提唱されたが,その分子実体が明らかになるのに40年もの年月を要した.このブレイクスルーは,もっともシンプルな真核生物のモデル系である出芽酵母によるものであった.ここでは,出芽酵母がどのようにオートファジー研究に飛躍をもたらしたかを,これまで筆者らが得たオートファジーの分子機構に関する知見を中心に解説する.
はじめに
オートファジーとは,細胞の自己成分を細胞内の消化器官であるリソソームあるいは液胞に輸送し分解する,酵母からヒトまで真核生物に広く保存された一連の過程をさす.当初,想定された栄養飢餓の際の生体物質のリサイクル系としての機能だけでなく,最近では,発生と分化,がんや神経変性疾患の抑制,老化,免疫応答,抗原提示,細胞死,病原体の排除など,オートファジーのかかわる生命現象は枚挙にいとまがない1).このように,オートファジーのテリトリーが急速に拡張したのはここ10年のことであることから,オートファジーを新しい学問領域だと思っている人は多いのではないだろうか.オートファジーの概念が提唱されたのはいまからほぼ50年前のことであり,これは,もうひとつの細胞内タンパク質分解系であるユビキチン-プロテアソーム系の発見より25年も前になる.しかしながら,オートファジーは誕生からほどなく表舞台から消えていった2).最近になり,ふたたび脚光をあびるようになったのは,出芽酵母Saccharomyces cerevisiaeを用いた研究を端緒とするものである.ここでは,出芽酵母がどのようにオートファジー研究の推進に寄与したのかを,オートファジー研究の歴史的な推移,また,オートファジーの分子機構に関する知見をまじえ紹介する.これまで,3種類のオートファジー,ミクロオートファジー,マクロオートファジー,シャペロン介在オートファジーが知られているが,ここではそのうち,主要な経路でありもっとも知見の蓄積しているマクロオートファジーをとりあげ,以降,これを単にオートファジーとよぶ.
1.オートファジーの誕生から眠りにつくまで
近年,“動的平衡”という言葉をよく耳にすることもあり,現代のわれわれは自分のからだを構成している物質は合成と分解との平衡状態にあることを知っている.生体構成物質,とくにタンパク質のターンオーバーの概念は,ちょうど70年前の1942年,生物に対しはじめて同位体標識を試みたSchoenheimerによりもたらされた3).しかし,タンパク質の合成に関する知見の膨大な蓄積に対し,もう一方の分解についての知見は皆無なままであった.
タンパク質分解の概念が具現化したのは,1950年代から1960年代,米国Rockefeller大学においてであった.当時,電子顕微鏡により細胞の微細構造が観察されるようになり,その微細構造体を細胞分画により分離して生化学的な方法で機能を明らかにするという細胞生物学がまさにここで花開きつつあった.こうしたなか,1953年から1955年にかけて,de Duveにより多様な加水分解酵素を含むオルガネラとしてリソソームが発見された4,5).de Duveは,この発見により1974年,Rockefeller大学の同僚であったPalade,Claudeとともにノーベル医学生理学賞を受賞している.加水分解酵素が細胞質に散在することは生物にとり危険きわまりないことは容易に想像され,これらをリソソームという膜コンパートメントに隔離することは非常に理にかなっていたが,同時に,分解されるべき基質をどのようにこのオルガネラに輸送しているのかが問題になった.1960年前後,ミトコンドリアや小胞体といったオルガネラやリボソームなど細胞質成分を含む小胞が電子顕微鏡により観察された.さらに,内容物がリソソームのもつ加水分解酵素により消化されつつある小胞の像が得られた6).de Duveは,細胞質成分が小胞としてリソソームと融合し分解される一連の過程をオートファジー,そして,その小胞をオートファゴソームとはじめて命名した.いまからほぼ50年前の1963年のことである7).その翌年には,栄養飢餓によりオートファジーの亢進されることが観察され,オートファジーは自己の細胞質成分をリソソームにおいて分解し,アミノ酸などの生体内物質をリサイクルする機構であると考えられた.しかしながら,この時点ではオートファジーは細胞におけるタンパク質分解マシナリーとしての地位をゆるぎないものとするまではいたらなかった.おのおののタンパク質が固有の半減期をもつという事実と,基質をバルクとしてリソソームにおいて非選択的に分解するオートファジーは概念的にあいいれなかった.さらに,反証する結果がde DuveのいたRockefeller大学のPooleらによりもたらされた8).クロロキンやイオノホアX537Aの処理によりリソソームのもつ加水分解酵素を阻害すると,ファゴサイトーシスにより細胞の外から取り込まれたタンパク質の分解は抑制されたものの,恒常的に起こっている生体でのタンパク質分解にはほとんど影響がみられなかったのである.
この発表がなされた1977年には,ユビキチン-プロテアソーム系の扉が開かれた.1964年,Rabinovitzにより,分化の途上でリソソームを消失する網状赤血球においても,タンパク質分解は促進されていることが判明していた9).網状赤血球で観察されたリソソームに依存しないタンパク質分解系,これこそが,ユビキチン-プロテアソーム系にほかならなかった.ユビキチン-プロテアソーム系の発見はリソソームおよびオートファジーの研究の副次的な産物であったことになる.1977年,Goldbergが網状赤血球抽出液を用いてATPに依存性のタンパク質分解無細胞系を確立し10),1978年,Herskoらは,さらに分画に着手した11).ユビキチン-プロテアソーム系の誕生である.1990年代に入るころにはユビキチン化反応の全貌が明らかにされ,さらに,ユビキチン化されたタンパク質を分解する本体であるプロテアソームが同定されるなど,ユビキチンはタンパク質分解シグナルとして機能し,ユビキチン化されたタンパク質はプロテアソームにより選択的に分解される,というスキームが確立した.さらに,ユビキチン-プロテアソーム系は,細胞周期やDNA損傷応答,転写,シグナル伝達など,生命現象の実に多様な局面にかかわっていることが明らかになり,細胞におけるタンパク質分解系としてユビキチン-プロテアソーム系が席巻した.ユビキチン-プロテアソーム系は無細胞系から研究がはじまり,ほどなくin vitroにおける完全再構成系が構築されるなど,生化学的な手法によりその分子機構にせまることができ,また,その過程で分子実体が同定されると,その遺伝子の同定,逆遺伝学による機能解析など,分子生物学的な手法によるアプローチがスムーズに遂行された.一方,オートファジーにかかわる遺伝子やタンパク質についてほとんど研究に進展がみられなかったのは,電子顕微鏡による観察がオートファゴソームを検出する唯一の手段であったことが大きな要因であった.オートファジーに特異的に関与する遺伝子がはじめてクローニングされたのは1996年であったが,その研究対象となったのは,それまでオートファジーの研究に使われてきた哺乳動物ではなく,誰にもかえりみられることのなかったもっともシンプルな真核生物のモデル系,出芽酵母であった12).
2.オートファジーの長き眠りからのめざめ
1992年,大隅良典らは,出芽酵母においてオートファジーをはじめて観察した13).液胞は真菌から高等植物まで存在するオルガネラである.細胞において最大のコンパートメントであり,出芽酵母では細胞の全体積の25%以上をしめる.液胞には多数の加水分解酵素が内在していることから,古くから細胞における分解コンパートメントであると考えられてきた.液胞に存在する加水分解酵素の活性は栄養状態により変動し,とくに栄養飢餓により誘導される.また,出芽酵母において液胞の加水分解酵素を欠損した株は富栄養状態においては顕著な表現型を示さないが,栄養飢餓により誘導される胞子形成が不全になる.このことから,液胞は栄養飢餓状態に対し重要な生理機能をもつことが示唆されていた.栄養飢餓培地に移したタンパク質分解酵素欠損株の形態変化を光学顕微鏡で観察することにより,液胞に球形の構造体が認められた.野生株では希有なこの構造体は,液胞に取り込まれたものの,タンパク質分解酵素の欠損のため分解されずに蓄積した小胞であった.電子顕微鏡でのさらなる観察により,これは細胞質成分をそのまま含む一重膜の構造体であることが示され,オートファジックボディーと名づけられた.また,細胞質成分をとりかこんださまざまな二重膜構造を細胞質において観察するにいたり,以下のオートファゴソーム形成の構図がうかびあがった(図1).栄養飢餓に応答して細胞質において隔離膜と名づけられた嚢状の膜構造が出現し,膜の伸張とともに細胞質成分をとりかこむ.そののち,最終形態であるオートファゴソームの外膜が液胞膜と融合することにより,内膜にとりかこまれた内容物は液胞の内部へと放出される.液胞がリソソームに比べはるかに大きいことを除けば,出芽酵母でみられたこの一連の膜動態は,まさにde Duveの提唱したオートファジーそのものであった.
この出芽酵母におけるオートファジーの発見が,オートファゴソームの形成をつかさどる分子実体の解明を一気に押し進めることになった.この推進の要因は2点に集約される.1つ目に,この時期の出芽酵母の研究は,従来の変異体の分離,遺伝解析といった古典的な遺伝学にくわえ,遺伝子クローニング,逆遺伝学といった分子生物学的な解析の技術基盤が確立していただけでなく,真核生物においてはじめてのゲノムプロジェクトとしてゲノム全塩基配列の解読が精力的になされていて,ゲノム情報の利用が可能になっていた.出芽酵母ゲノムプロジェクトの完了は1997年である.2つ目として,動物細胞におけるオートファジーは電子顕微鏡でしかとらえられなかったのに対し,出芽酵母のオートファジーは液胞酵素の不活性株を用いることで位相差顕微鏡によりオートファジックボディーの蓄積として容易にとらえることができた.
ひきつづいて1993年,大隅らは出芽酵母のオートファジー不全変異体を分離することに成功した14).まず,液胞酵素欠損株にみられるはずのオートファジックボディーが液胞に蓄積しない変異株を光学顕微鏡での観察により単離した.このとき,唯一,得られたのがapg1変異株であった.このAPG1遺伝子は現在ではATG1遺伝子にあたる(後述).このapg1変異株は栄養飢餓状態では急速に生存率の低下することがわかり,栄養飢餓状態での生存率の低下とオートファジックボディーの形成不全の2つを指標としたスクリーニングにより,apg1変異株にくわえて,さらに13の相補群に分類されるオートファジー不全変異体を分離した.そして,これらの変異体からオートファジーに必須の14個の遺伝子が同定された.これらは1つを除きすべてが新規の遺伝子であり,オートファジーがこれまでにない分子機構で駆動されていることが予想された.オートファジー不全変異体は,栄養飢餓がシグナルへと変換し伝達される段階,オートファゴソーム形成の段階,オートファゴソームが液胞と融合し内容物が分解される段階,のいずれかに異常を生じていると考えられた.同定されたすべての遺伝子を欠損した株では電子顕微鏡ではオートファゴソームがまったく観察されなかったことから,これらの遺伝子はオートファゴソーム形成の段階,もしくは,それ以前に関与しているものと考えられた.さらに2001年には,GFPとの融合タンパク質を用いることで,これら遺伝子のほぼすべての産物は栄養飢餓状態に応答して液胞の近傍の一点に局在することが明らかになった15).この構造体は蛍光顕微鏡でのみ観察され電子顕微鏡ではいまだとらえられていないなど,その実体の詳細は不明であるもののオートファゴソームの形成の場であると考えられ,PAS(pre-autophagosomal structure,プレオートオートファゴソーム構造体)とよばれる.
2004年には,おもにThummらにより分離されたaut変異株や16),Klionskyらにより分離されたcvt変異株17),さらに,そのほかの出芽酵母オートファジー関連遺伝子をあわせて,名称がATG(autophagy-related)遺伝子に統一された18).現在では30をこえるATG遺伝子が見い出されている.そのうち,最初に発見された14個にくわえ,合計18個(Atg1~Atg10,Atg12~Atg14,Atg16~Atg18,Atg29,Atg31)がオートファゴソームの形成に必須の遺伝子とされている.これまでの解析により,これら遺伝子の産物であるオートファゴソームの形成に必須な18個のタンパク質(以下,“Atgタンパク質”とよぶ)は,いくつかの機能単位を構成していることが明らかになっている19)(図2).以下,この機能単位ごとに,オートファゴソーム形成の分子機構を,筆者らのこれまで得た知見を中心に解説する.
3.オートファジーのめざめ以降の知見:Atgタンパク質の機能
1)Atg1キナーゼ複合体
Atgタンパク質について,その1次構造から得られる情報はほとんど皆無であった.例外のひとつがAtg1である.ATG1遺伝子は1997年にクローニングされ,その1次構造からAtg1はリン酸化酵素(キナーゼ)であることがわかった.この酵素の過剰発現により出芽酵母のatg13変異株のオートファジー活性が回復したことからAtg1とAtg13の機能的な関連性が示唆されたが,その機構は不明のままであった.ここに,“生物が細胞内外の栄養状態をどのように感知しているか”という生物学において大きな問題がかかわってきた.1991年,免疫抑制剤ラパマイシンの生体における標的タンパク質としてTorが同定され,ラパマイシンはTorの特異的な阻害剤として作用することが実証された.Torは脂質リン酸化酵素に相同性をもっていたがその基質は不明であった.ラパマイシン処理またはTorへの変異導入によりTorを不活性化すると,G1期の初期において細胞周期が停止するなど栄養飢餓のときと同じ挙動が観察され,Torは栄養状態に応じた細胞の増殖制御を担っていると考えられた.1998年,Torの不活性化にともない富栄養状態においてもオートファジーの誘導されることが明らかになり,富栄養状態ではTorがオートファジーを抑制していることがわかったが,Torによりリン酸化される基質は不明なままであった20).2000年,これらの疑問が解決された.富栄養状態では高度にリン酸化されているAtg13がラパマイシン処理により脱リン酸化型に変換され,Atg1と複合体を形成すること,それにともないAtg1のリン酸化酵素活性の亢進することが示されたのである21).Atg13がTorに依存してリン酸化されることでオートファジーが抑制され,栄養飢餓のときには脱リン酸化型Atg13とAtg1からなるAtg1キナーゼ複合体が形成される,そして,Atg1のもつリン酸化酵素活性の亢進がオートファジー誘導のもっとも初期段階である,というスキームができた(図3).2010年には,Torによるリン酸化をうけない変異型のAtg13をもつ出芽酵母ではオートファジーが恒常的に活性化されていたことから,オートファジーの抑制の際のTorのおもな基質はAtg13であることが確認された22).Atg1のリン酸化酵素活性はオートファジーに必須であるが,その基質はまだ明らかになっていない.近年,栄養飢餓状態において,Atg1キナーゼ複合体にはAtg13にくわえ,Atg17,Atg29,Atg31が含まれ,これらを欠損した株ではAtgタンパク質のPASへの局在が失われたことから,Atg1キナーゼ複合体はPASのもっとも基部になっていると考えられる23).
2)PI3キナーゼ複合体とそのエフェクタータンパク質
1998年にクローニングされたATG6遺伝子は,オートファゴソームの形成に必須な18個の遺伝子のなかで唯一,既知の遺伝子であり,Emrらにより,カルボキシペプチダーゼYが本来の液胞に局在せず細胞外に分泌されるvps(vacuolar protein sorting)変異体の責任遺伝子のひとつとして分離されVPS30遺伝子と名づけられていた.出芽酵母のatg6変異株はオートファジーと液胞酵素の輸送に異常を生じた.しかし,同時に遺伝子クローニングの報告されたAtg14は,栄養状態に関係なくつねにAtg6と複合体を形成するにもかかわらず,Atg14の過剰発現ではatg6変異株のオートファジー不全のみが抑圧され,かつ,atg14変異体はオートファジーにのみ異常を生じた24).これは,同じ複合体の構成タンパク質であるにもかかわらず,変異体の表現型は異なるという不可解な結果であった.2001年,Atg6と相互作用するタンパク質として新たにVps15,Vps34,Vps38が同定された.さらなる生化学的な解析から,Atg6は2つの複合体,すなわち,Vps15,Vps34,Atg6,Atg14からなる複合体Iと,Vps15,Vps34,Atg6,Vps38からなる複合体IIを形成しており,複合体Iがオートファジーに,複合体IIが液胞酵素の輸送に寄与していることが判明した25)(図4).この2つの複合体に共通する構成タンパク質であるAtg6の変異体が2つの異常を示すのは当然であった.Vps34は生体膜リン脂質の一種,ホスファチジルイノシトール3-リン酸を合成するPI3キナーゼであった.その活性はオートファジーに必須であり,産生されるホスファチジルイノシトール3-リン酸は下流のエフェクタータンパク質であるAtg2を,ホスファチジルイノシトール3-リン酸との結合能をもつAtg18との複合体としてリクルートした26).しかし,Atg18のPASへの局在はAtg2に依存していたこと,atg18欠損株においてAtg2を別のホスファチジルイノシトール3-リン酸結合ドメインとの融合タンパク質により人工的にPASへと局在させてもオートファジーに異常を示したことから,Atg18のホスファチジルイノシトール3-リン酸との結合能とは別の分子機能がオートファゴソームの形成に必須であると考えられた.Atg2は約1600アミノ酸残基からなる巨大なタンパク質だが,既知の機能ドメインがなくその機能の詳細は不明である.PASへの局在におけるほかのAtgタンパク質への依存性から考えてオートファゴソーム形成の最終段階に機能することが示唆され,Atg2の機能,すなわち,Atg2の未知の酵素活性やさらなる下流タンパク質を探ることが,隔離膜の脂質はどこからどのように供給されるのか,膜の伸張や曲がりはどのようになされるのか,といったオートファゴソーム形成の本質的な疑問の解決に直結すると考えられる.
3)膜タンパク質Atg9
Atg9はAtgタンパク質のなかで唯一,膜貫通領域をもつ膜タンパク質であり,長いあいだ,オートファゴソームの形成において脂質の供給源であると考えられてきた.ごく最近,細胞質を激しくうごめく約30分子のAtg9が膜に存在する30~60 nmの小胞の存在がとらえられた27).蛍光顕微鏡による定量解析から,栄養飢餓に応じPASには平均3個のAtg9小胞しかリクルートされず,それ以上のAtg9小胞の流入はみられないことから,Atg9は脂質の供給源として膜の伸張に関与するより,オートファゴソーム形成の核として初期段階に機能しているようである(図5).Atg9は最終的にオートファゴソームの外膜に局在する.
4)オートファジーに内包される2つのユビキチン様結合反応系
オートファジー研究においてもっとも驚きであったのは,Atgタンパク質にユビキチン様結合反応系が2つも含まれることであったといっても過言ではない.ユビキチン結合反応系とは,ユビキチン活性化酵素(E1),ユビキチン結合酵素(E2),ユビキチンリガーゼ(E3)の3種の酵素のはたらきにより,ATPに依存的に,ユビキチンのC末端のグリシン残基と標的タンパク質のリジン残基のεアミノ基とのイソペプチド結合が形成される系である(図6a).
1998年,Atg12結合反応系が明らかになった28).186アミノ酸残基からなるAtg12は,ウェスタンブロットによりAtg12自体のほかに,40 kDほど大きいバンドが認められた.Atg5もウェスタンブロットにより同様に2本のバンドが認められ,高分子量のバンドはAtg12のウェスタンブロットにより得られたバンドと一致した.最終的に,この高分子量のバンドは,Atg12のC末端のアミノ酸残基がAtg5の149番目のリジン残基と共有結合したものであることが判明した.Atg12のC末端のグリシン残基の欠失,あるいは,Atg5の149番目のリジン残基の変異により,この結合体の形成不全とオートファジー不全が示されたことから,Atg5を標的としたAtg12によるユビキチン様結合反応系がオートファジーに必須であると考えられた.ついで,E1,E2,E3の実体が問題となった.出芽酵母のatg7変異株およびatg10変異株ではAtg12-Atg5結合体は消失した.ユビキチン様結合反応系の特徴であるチオエステル結合がAtg12とAtg7のあいだ,および,Atg12とAtg10のあいだでみられたこと,Atg7がユビキチン活性化酵素Uba1と相同性をもっていたこと,Atg12-Atg7結合体はATG10遺伝子に依存しないがAtg12-Atg10結合体はATG7遺伝子に依存したことから,Atg7がE1,Atg10がE2であることが判明した(図6a).E3は未同定であるが,in vitroにおける再構成系においてAtg7とAtg10のみでAtg12-Atg5結合体が高効率で生成されたことから,E3はこの系には必要ないと考えられている.Atg12結合反応系には逆反応,すなわち,脱Atg12化酵素の存在しないことも,ほかのユビキチン様結合反応系とは異なる点であった.Atg12はC末端がグリシン残基である成熟型として遺伝子にコードされていた.また,Atg12-Atg5結合体はAtg16と相互作用し,Atg16との結合を介し二量体として存在した.この二量体の形成はPASへの局在に必須であった.
Atg8がもうひとつのユビキチン様結合反応系にかかわることは,2000年に判明した.Atg8のC末端側にタグを導入すると,これは生体においてATG4遺伝子に依存的に除去された.精製標品を用いた実験から,Atg4はシステインプロテアーゼであり,Atg4によりAtg8のC末端のアルギニン残基が除去されグリシン残基の露出されることが明らかになった29).出芽酵母のatg4変異株におけるオートファジー不全が,C末端のアルギニン残基を欠きグリシン残基となったAtg8の変異体により一部抑圧されたことから,Atg8のC末端の露出したグリシン残基が重要であること,また,Atg12との1次構造における相同性からAtg8はユビキチン様タンパク質であることが予想された.免疫電子顕微鏡法によりAtg8のオートファゴソームへの局在が観察されたが,これに一致するように,生化学的な解析においてもAtg8は膜画分に存在した.atg4変異株やC末端のグリシン残基を欠損したAtg8の変異体ではAtg8は膜と相互作用できなかったことから,Atg8の生体における標的物質は膜に強く結合するものであると考えられ,精力的に探索された.その結果,高濃度の尿素の存在のもとでのウェスタンブロットによりごくわずかな移動度の差を示す2本のバンドが認められたが,この時点では標的物質は不明であった.同じ年,移動度の大きなバンドはホスファチジルエタノールアミンの頭部のアミノ基とAtg8のグリシン残基とがアミド結合を形成したものであることが明らかになった30).これは,現在までに知られているユビキチン様タンパク質のうち,タンパク質ではなく脂質を標的とする唯一の例である.Atg8-ホスファチジルエタノールアミン結合体はatg7変異体およびatg3変異体において消失し,Atg8とAtg7のあいだ,および,Atg8とAtg3のあいだにチオエステル結合がみられたこと,Atg3は活性化システイン残基の近傍においてAtg10と相同であったことから,Atg7がE1,Atg3がE2であることが明らかになった(図6a).Atg8とAtg12はおのおの異なるE2をもつにもかかわらず,共通のE1としてAtg7を用いるという特異な反応機構をもっていた.精製したAtg12-Atg5結合体をAtg8の再構成反応系に添加するとAtg8のホスファチジルエタノールアミン化が亢進した.2007年には,Atg8のオートファゴソーム形成における機能の一端が明らかになった31).in vitroにおける再構成反応系において,Atg8のホスファチジルエタノールアミン化にともない,ホスファチジルエタノールアミンを含むリポソームが凝集体を形成した.Atg8-ホスファチジルエタノールアミン結合体はホモ多量体を形成していたことから,この多量体化により膜どうしをつなぎあわせる機能をもつものと考えられた.さらに,この凝集した小胞の膜が,脂質二重層のうち外側の一層のみが融合するヘミフュージョンを誘起していることがわかった(図6b).in vitroにおいてリポソームの凝集やヘミフュージョンの起こらないあるいは低下しているAtg8の変異体を分離したところ,これらの変異体をもつ出芽酵母ではそのin vitroにおける活性に相関して,オートファジックボディーができないか,できても小型化しオートファジーに異常を生じたことから,Atg8は細胞においてもオートファゴソームの形成の際の膜の伸張や最終段階における隔離膜の末端の融合に寄与していると考えられた.
おわりに
哺乳動物においてはじまったオートファジーの研究は,長い停滞期をへて,出芽酵母においてその分子実体が解明された.Atgタンパク質は生物種をこえて進化的に広く保存されており,そのホモログをノックアウトまたはノックダウンすることにより,哺乳動物におけるオートファジーが再検証された32).その結果,生体物質のリサイクルという基本的な役割のほかにも実に多様な生命現象への関与が明らかになり,現在,哺乳動物のオートファジー研究は興隆をきわめている.しかし,Atgタンパク質がどのように有機的に連携してオートファゴソームの形成にいたるのかというオートファジーにおけるもっとも重要な問題は,出芽酵母においてもほとんど解明されていない.
その大きな障壁のひとつとして,オートファゴソーム形成の素過程を解明するための中間体が観察されていないことがあげられる.出芽酵母のオートファゴソーム形成必須遺伝子の変異株ではすべて,電子顕微鏡による観察ではオートファゴソームの形成がみられず,オートファゴソーム形成の進行の途中で停止したような膜中間体を検出できていない.この現状を打破するには,in vitroにおけるオートファゴソーム形成の再構成系の構築がさけてとおれないであろう.必要となるタンパク質や脂質などが多岐にわたり非常な困難が予想される一方で,現在までの網羅的なスクリーニングにもかかわらずオートファゴソーム形成必須遺伝子は18から増えていないという事実から,典型的なAtgタンパク質はすべて単離されていると考えられる.既知のAtgタンパク質とその関連タンパク質,および,脂質を混合することで,あんがい簡単にオートファゴソーム様の脂質二重膜からなる小胞の出現が観察されるかもしれない.これには,オートファゴソームの脂質組成の解明が必須であろう.これまで膜の融合においては,SNAREを用いた融合,液胞の融合33),エンドソーム膜の融合など34),in vitro完全再構成系の成功例が散見される.オートファゴソームは二重膜であり,さらに,その形成過程は膜の伸張,変形,融合からなるため,その複雑さはこれらとは比較にならない.しかし,いったんin vitroにおける再構成系が構築されれば,そこから得られる恩恵は計りしれないものになるであろう.
文 献
- Mizushima, N. & Komatsu, M.: Autophagy: renovation of cells and tissues. Cell, 147, 728-741 (2011)[PubMed]
- Ciechanover, A.: Intracellular protein degradation: from a vague idea thru the lysosome and the ubiquitin-proteasome system and onto human diseases and drug targeting. Cell Death Differ., 12, 1178-1190 (2005)[PubMed]
- Schoenheimer, R.: The Dynamic State of body Constituents. Harvard University Press, Cambridge (1942)
- De Duve, C., Gianetto, R., Appelmans, F. et al.: Enzymic content of the mitochondria fraction. Nature, 172, 1143-1144 (1953)[PubMed]
- Gianetto, R. & De Duve, C.: Tissue fractionation studies. 4. Comparative study of the binding of acid phosphatase, β-glucuronidase and cathepsin by rat-liver particles. Biochem. J., 59, 433-438 (1955)[PubMed]
- Ashford, T. P. & Porter, K. R.: Cytoplasmic components in hepatic cell lysosomes. J. Cell Biol., 12, 198-202 (1962)[PubMed]
- Yang, Z. & Klionsky, D. J.: Eaten alive: a history of macroautophagy. Nat. Cell Biol., 12, 814-822 (2010)[PubMed]
- Poole, B., Ohkuma, S., & Warburton, M. J.: The accumulation of weakly basic substances in lysosomes and the inhibition of intracellular protein degradation. Acta Biol. Med. Ger., 36, 1777-1788 (1977)[PubMed]
- Rabinovitz, M. & Fisher, J. M.: Characteristics of the inhibition of hemoglobin synthesis in rabbit reticulocytes by threo-α-amino-β-chlorobutyric acid. Biochim. Biophys. Acta, 91, 313-322 (1964)[PubMed]
- Etlinger, J. D. & Goldberg, A. L.: A soluble ATP-dependent proteolytic system responsible for the degradation of abnormal proteins in reticulocytes. Proc. Natl. Acad. Sci. USA, 74, 54-58 (1977)[PubMed]
- Ciehanover, A., Hod, Y. & Hershko, A.: A heat-stable polypeptide component of an ATP-dependent proteolytic system from reticulocytes. Biochem. Biophys. Res. Commun., 81, 1100-1105 (1978)[PubMed]
- Kametaka, S., Matsuura, A., Wada, Y. et al.: Structural and functional analyses of APG5, a gene involved in autophagy in yeast. Gene, 178, 139-143 (1996)[PubMed]
- Takeshige, K., Baba, M., Tsuboi, S. et al.: Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction. J. Cell Biol., 119, 301-311 (1992)[PubMed]
- Tsukada, M. & Ohsumi, Y.: Isolation and characterization of autophagy-defective mutants of Saccharomyces cerevisiae. FEBS Lett., 333, 169-174 (1993)[PubMed]
- Suzuki, K., Kirisako, T., Kamada, Y. et al.: The pre-autophagosomal structure organized by concerted functions of APG genes is essential for autophagosome formation. EMBO J., 20, 5971-5981 (2001)[PubMed]
- Thumm, M., Egner, R., Koch, B. et al.: Isolation of autophagocytosis mutants of Saccharomyces cerevisiae. FEBS Lett., 349, 275-280 (1994)[PubMed]
- Scott, S. V., Hefner-Gravink, A., Morano, K. A. et al.: Cytoplasm-to-vacuole targeting and autophagy employ the same machinery to deliver proteins to the yeast vacuole. Proc. Natl .Acad. Sci. USA, 93, 12304-12308 (1996)[PubMed]
- Klionsky, D. J., Cregg, J. M., Dunn, W. A. Jr. et al.: A unified nomenclature for yeast autophagy-related genes. Dev. Cell, 5, 539-545 (2003)[PubMed]
- Nakatogawa, H., Suzuki, K., Kamada, Y. et al.: Dynamics and diversity in autophagy mechanisms: lessons from yeast. Nat. Rev. Mol. Cell Biol., 10, 458-467 (2009)[PubMed]
- Noda, T. & Ohsumi, Y.: Tor, a phosphatidylinositol kinase homologue, controls autophagy in yeast. J. Biol. Chem., 273, 3963-3966 (1998)[PubMed]
- Kamada, Y., Funakoshi, T., Shintani, T. et al.: Tor-mediated induction of autophagy via an Apg1 protein kinase complex. J. Cell Biol., 150, 1507-1513 (2000)[PubMed]
- Kamada, Y., Yoshino, K., Kondo, C. et al.: Tor directly controls the Atg1 kinase complex to regulate autophagy. Mol. Cell. Biol., 30, 1049-1058 (2010)[PubMed]
- Kawamata, T., Kamada, Y., Kabeya, Y. et al.: Organization of the pre- autophagosomal structure responsible for autophagosome formation. Mol. Biol. Cell, 19, 2039-2050 (2008)[PubMed]
- Kametaka, S., Okano, T., Ohsumi, M. et al.: Apg14p and Apg6/Vps30p form a protein complex essential for autophagy in the yeast, Saccharomyces cerevisiae. J. Biol. Chem., 273, 22284-22291 (1998)[PubMed]
- Kihara, A., Noda, T., Ishihara, N. et al.: Two distinct Vps34 phosphatidylinositol 3-kinase complexes function in autophagy and carboxypeptidase Y sorting in Saccharomyces cerevisiae. J. Cell Biol., 152, 519-530 (2001)[PubMed]
- Obara, K., Sekito, T., Niimi, K. et al.: The Atg18-Atg2 complex is recruited to autophagic membranes via phosphatidylinositol 3-phosphate and exerts an essential function. J. Biol. Chem., 283, 23972-23980 (2008)[PubMed]
- Yamamoto, H., Kakuta, S., Watanabe, T. M. et al.: Atg9 vesicles are an important membrane source during early steps of autophagosome formation. J. Cell Biol., 198, 219-233 (2012)[PubMed]
- Mizushima, N., Noda, T., Yoshimori, T. et al.: A protein conjugation system essential for autophagy. Nature, 395, 395-398 (1998)[PubMed]
- Kirisako, T., Ichimura, Y., Okada, H. et al.: The reversible modification regulates the membrane-binding state of Apg8/Aut7 essential for autophagy and the cytoplasm to vacuole targeting pathway. J. Cell Biol., 151, 263-276 (2000)[PubMed]
- Ichimura, Y., Kirisako, T., Takao, T. et al.: A ubiquitin-like system mediates protein lipidation. Nature, 408, 488-492 (2000)[PubMed]
- Nakatogawa, H., Ichimura, Y. & Ohsumi, Y.: Atg8, a ubiquitin-like protein required for autophagosome formation, mediates membrane tethering and hemifusion. Cell, 130, 165-178 (2007)[PubMed]
- Mizushima, N., Yoshimori, T. & Ohsumi, Y.: The role of Atg proteins in autophagosome formation. Annu. Rev. Cell Dev. Biol., 27, 107-132 (2011)[PubMed]
- Wickner, W. & Schekman, R.: Membrane fusion. Nat. Struct. Mol. Biol., 15, 658-664 (2008)[PubMed]
- Ohya, T., Miaczynska, M., Coskun, U. et al.: Reconstitution of Rab- and SNARE-dependent membrane fusion by synthetic endosomes. Nature, 459, 1091-1097 (2009)[PubMed]
参考図書
Special Issue on Autophagy. FEBS Lett., 584, 1279-1436 (2010)
著者プロフィール
略歴:2001年 東京大学大学院薬学系研究科博士課程 修了,同 助手,英国Paterson Institute for Cancer Research研究員,ドイツHeidelberg大学ZMBH研究員,東京工業大学先進研究機構 先進研究員を経て,2011年より東京工業大学フロンティア研究機構 特任助教.
研究テーマ:オートファジー,とくに,オートファゴソーム形成の分子機構.論理的かつ厳密な出芽酵母ならではの研究によりオートファジーを理解したい.
大隅 良典(Yoshinori Ohsumi)
東京工業大学フロンティア研究機構 特任教授.
研究室URL:http://www.ohsumilab.aro.iri.titech.ac.jp/
© 2012 荒木保弘・大隅良典 Licensed under CC 表示 2.1 日本