植物の生殖過程におけるエピジェネティックな情報のリプログラミング
2013/01/15
木下 哲
(奈良先端科学技術大学院大学バイオサイエンス研究科 植物生殖遺伝学研究チーム)
email:木下 哲
領域融合レビュー, 2, e001 (2013) DOI: 10.7875/leading.author.2.e001
Tetsu Kinoshita: Epigenetic reprograming of maternal and paternal genomes during plant sexual reproduction.

近年のエピジェネティクスの研究の進捗により,DNA塩基配列以外の情報が遺伝情報としてはたらいていることへの理解は急速に進んだ.しかしながら,その全容は依然として混沌としており,多くの研究者を魅惑してやまない.ここでは,多くの生命現象のなかでも,エピジェネティクスととくに関連の深い植物の生殖過程をとりあげる.植物の複雑な生殖過程において,どのような分子機構によりエピジェネティックな情報が制御されているのかを解説し,その生物学的な意義を考察する.
暖かな春の季節になると,多くの草花がわれわれの目を楽しませてくれる.一方で,植物にとって花を咲かせることは,つぎの世代へ種子を残すための重要な営みのひとつである.このような植物の生殖過程では,おしべとめしべの相互作用により,種のアイデンティティの維持や,集団における多様性の維持に関して,驚くべきダイナミックなせめぎあいが起こっている.エピジェネティックな情報を含めた遺伝情報をどのように“正しく”伝達するか,あるいは,ある“許容範囲内”での変化を伝えるのか,また,その結果,どのような個性や多様性が付与されるのか,はたまた,世代のくり返しのはてに植物の進化に対しどのような影響をあたえるのか,その詳細はほとんど明らかになっていない.研究の進捗により,DNAのメチル化,ヒストンの修飾,低分子RNAなど,DNAの塩基配列以外のエピジェネティックな情報そのものに対する理解は深まったが,その情報が生殖過程においてどのようにリプログラミング(書き換え)されるのか,現在は,新たな知見がつぎつぎと報告されるようになった段階である1,2).
一般的な植物のDNAメチル化制御に関してはほかに詳細な総説があるため3),ここでは簡単に述べるにとどめるが,植物には3つのカテゴリーのDNAメチル化部位が存在する.最近では,哺乳動物でもES細胞などごく一部の細胞においてCpG配列以外の配列でのDNAメチル化がみつかっているが,植物では多くの場合,CpG配列のほか,CpHpG配列およびCpHpH配列(H = C,T,A)のシトシン塩基の5位にメチル基が付加され5-メチルシトシンとなる.シロイヌナズナでは,CpG配列はおもにDNA複製にともない一方のDNA鎖のみがメチル化したヘミメチル化部位において維持型DNAメチル化酵素MET1によりメチル化される.CpHpG配列はヒストンH3の9番目のリジン残基のメチル化を担うヒストンメチル化酵素KYPのはたらきに依存して植物に特異的なクロモメチル化酵素CMT3によりメチル化される.CpHpH配列はRdDM(RNA-directed DNA methylation,RNA指向型DNAメチル化)の機構により低分子RNAを介してde novo型メチル化酵素DRMによりメチル化される3).
植物の生殖過程におけるエピジェネティックな情報,とりわけ,DNAのメチル化がどのように制御されるかを理解するにあたっては,被子植物の重複受精のしくみを理解することが必須である.したがって,まず,最後に参考図書としてあげた,“東山哲也: 植物の生殖細胞と受精戦略. 領域融合レビュー, 1, e007 (2012)”を熟読してほしい.また,RdDMの機構に関しても,ここでは誌面の都合からくわしくは解説しないので,もう一方の参考図書,“植物におけるRNAサイレンシング機構”を熟読してほしい.そのうえで,植物の生殖過程においては,雌雄の配偶体,受精後の胚発生および胚乳発生において,エピジェネティックな情報が制御されリプログラミングされることに理解を進めてほしい.以下,現在,明らかになりつつある,植物の生殖過程におけるエピジェネティックな制御を順に解説する.
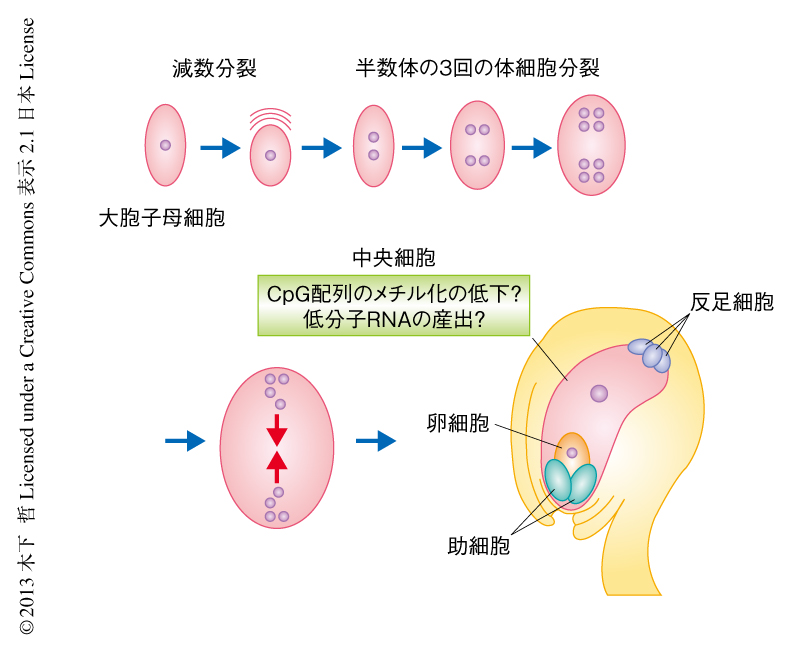
雌性配偶体は,めしべの奥深くに存在する胚珠組織において,大胞子母細胞の減数分裂ののち1つだけ残される大胞子が3回の半数体の体細胞分裂をへることで成熟する.多くの被子植物は,卵細胞,中央細胞,2つの助細胞,3つの反足細胞の8核7細胞からなり,胚嚢(embryo sac)ともよばれる(図1).このうち,花粉により運ばれてくる2つの精細胞との受精にあずかるのは卵細胞と中央細胞であり,それぞれ胚と胚乳を形成する.これらの細胞のうち,中央細胞においてもっとも顕著にDNAメチル化によるエピジェネティックな情報の変化がみられることが知られている4,5).
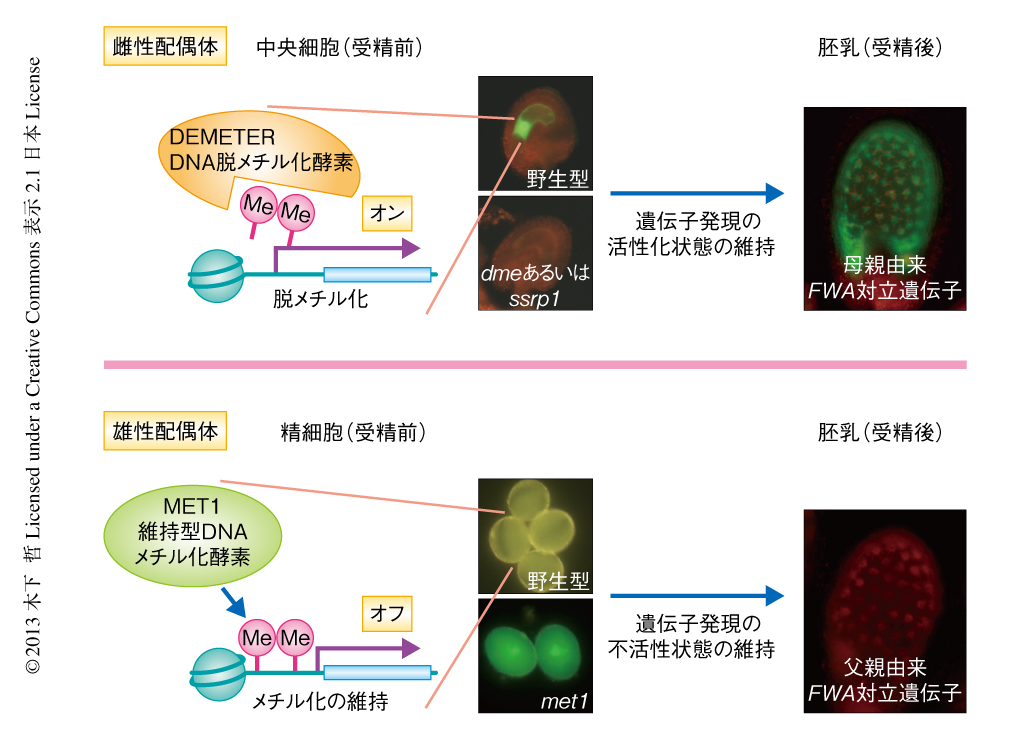
中央細胞におけるエピジェネティックな制御の研究の発端となったのは,インプリント遺伝子としてMEDEA(MEA)遺伝子の発見である6,7).インプリント遺伝子は,その対立遺伝子が父親から由来したか母親から由来したかに応じて遺伝子発現のオン・オフが決定される.このような現象はおもに哺乳動物と被子植物においてみつかっており,ゲノムインプリンティングとよばれている8).MEA遺伝子や,機能未知のHD-ZIP型の転写因子をコードするFWA遺伝子などのインプリント遺伝子は,DEMETER(DME)遺伝子にコードされるDNA脱メチル化酵素により,受精前の雌性配偶体の中央細胞においてDNA脱メチル化され,遺伝子発現が誘導される9,10)(図2).最近では,MEA遺伝子に関してはDNA脱メチル化のほかにもクロマチンのループ構造を介したエピジェネティックな制御のあることがモデルとして提唱されている11).さらには,マウスと同様に,次世代シークエンサーを用いたSNP(single nucleotide polymorphism,一塩基多型)の解析から,父親に由来する対立遺伝子あるいは母親に由来する対立遺伝子に特異的に発現するインプリント遺伝子の多数の候補がみつかってきている12).古くは,インプリント遺伝子はシロイヌナズナを用いた変異体の研究により発見されてきた.シロイヌナズナでは,ポリコーム複合体の構成タンパク質をコードするMEA遺伝子とFIS2(Fertilization-Independent-Seed)遺伝子にゲノムインプリンティングがみつかっている.また,遺伝子は異なっていたものの,単子葉植物および双子葉植物においてポリコーム複合体構成タンパク質をコードする遺伝子のどれかがゲノムインプリンティングをうけていたため,哺乳動物および被子植物におけるインプリント遺伝子は,ある進化的な選択圧の結果として生じたとするコンフリクト仮説13,14) を支持していた.しかしながら,次世代シーケンサーを用いた解析の結果は,イネ,シロイヌナズナ,トウモロコシのあいだで驚くほどの多様性があった12).インプリント遺伝子がどのように生じてきたかは,今後,検証する必要があるが,いずれにせよ,植物の生殖過程におけるエピジェネティックな制御は,こうしたインプリント遺伝子の解析をモデルとして理解されるようになってきたといっても過言ではないだろう.
DNA複製の過程では5-メチルシトシンは娘DNA鎖に取り込まれないため,DNAメチル化の維持機構がはたらかない場合にはDNA複製にともないDNAメチル化は減少していく.これを受動的DNA脱メチル化(passive DNA demethylation)とよぶ.一方,DNA複製に依存せずメチル基を積極的に取り除く機構も存在する.これを能動的なDNA脱メチル化(active DNA demethylation)とよぶ.
シロイヌナズナにおけるDME遺伝子の変異体の単離と,そののちの“能動的な”DNA脱メチル化酵素としてのDMEの研究は,植物の生殖過程におけるエピジェネティックなリプログラミングを知るうえでひとつの突破口となった15).また,哺乳動物におけるDNA脱メチル化の研究にも多大な影響を及ぼした16,17).DME遺伝子は比較的大きなタンパク質をコードするが,そのC末端側に塩基除去修復にかかわるDNAグリコシラーゼとよく似たドメイン構造をもつ18).シロイヌナズナにおいては4つのホモログ,DME遺伝子,DML2遺伝子,DML3遺伝子,ROS1遺伝子が存在する16).このうち,中央細胞において中心的な役割をはたしているのはDME遺伝子である19).単子葉植物であるイネにおいても,遺伝子ターゲティングの手法を用いてOsROS1a遺伝子を破壊すると,生殖過程にシビアな影響の生じることが示されている20).DMEをはじめ,これらのタンパク質にドメイン構造として存在するDNAグリコシラーゼは,多くの生物をつうじ塩基除去修復機構においてはたらく酵素として知られている.塩基除去修復機構では,まずG/Tなどミスマッチ塩基の認識とその切り出しが起こり,ヌクレオチド骨格の切断,修復DNAの合成とDNAリガーゼによるギャップの修復が起こり,DNA修復は完了する.シロイヌナズナにおけるROS1やDMEの生化学的な解析の結果,これらのタンパク質はグアニンと5-メチルシトシンとのペアをあたかもミスマッチペアのように認識し,5-メチルシトシン塩基の切り出しとヌクレオチド骨格の切断を行うことが示された.したがって,これらの酵素はDNA脱メチル化酵素であると理解されている.また,DNAリガーゼやDNAホスファターゼなど塩基除去修復機構にかかわる酵素が,このあとの段階においてDNA脱メチル化に必要であることが示されつつあり21,22),植物のDNA脱メチル化は塩基除去修復機構の分子機構と類似の経路により行われていることが理解されている(図3).
DNA脱メチル化の過程は,このような塩基除去修復機構による5-メチルシトシンの除去のほかにも,DNA脱メチル化配列の認識や決定,クロマチン構造やクロマチン機能の変化,あるいは,ヒストン修飾の変化をともなうことが予想される19).最近の研究により,これらの分子機構の一端がようやくみえはじめてきていた23,24).筆者らは,さきに述べた内在性のインプリント遺伝子であるFWA遺伝子の発現を可視化できるFWA-GFP遺伝子を利用して,ゲノムインプリンティングに異常のある変異体の選抜を行ってきた.FWA遺伝子は,その5’側の領域にSINEレトロトランスポゾンの挿入に由来すると考えられる反復配列が存在し,これがインプリンティングのシス制御領域であることが示されている25).この反復配列は,通常,高度にDNAメチル化をうけており遺伝子発現は抑制されているが,中央細胞においてDMEによりDNA脱メチル化をうけると遺伝子発現は誘導される9).変異体の選抜に用いたコンストラクトは,FWA遺伝子のゲノムインプリンティングの制御に必要なシス制御領域を含んでいる.
こうした系を用いて,HMGドメインをもつSSRP1(structure specific recognition protein 1)遺伝子がみつかった23)(新着論文レビュー でも掲載).SSRP1遺伝子はFACT(facilitate to chromatin transcription/transaction)ヒストンシャペロン複合体の構成タンパク質をコードしており26),酵母からヒトまで広く保存されている.SSRP1はSPT16とヘテロ二量体を形成し,転写の開始,転写伸張,DNA複製,DNA修復など,クロマチンの機能変換が必要な数多くの局面に登場するヒストンシャペロンであることが知られている26).ssrp1変異体は,FWA-GFP遺伝子を活性化できないことにくわえ,受精していないにもかかわらず鞘の伸張や中央細胞の核分裂がみられた.このような表現型は自発的な胚乳発生(autonomous endosperm development)とよばれ,ほかのインプリント遺伝子であるMEA遺伝子の変異体などにもみられる.そのため,FWA-GFP遺伝子の発現を指標に選抜した変異体ではあったが,SSRP1遺伝子はほかのインプリント遺伝子の発現にも影響することが考えられた.実際に,さまざまなインプリント遺伝子の発現を調べた結果,ssrp1変異体では多くのインプリント遺伝子の発現に影響のみられることがわかった.さらに,FWA遺伝子のDNAメチル化領域,すなわち,ゲノムインプリンティングのシス制御領域におけるDNA脱メチル化にも影響することがわかった23).
DNA脱メチル化研究の難問は,いつどこでどの遺伝子がどのようにDNA脱メチル化されるかという質問に生化学的にせまることがむずしいことにある.動物および植物をつうじて,ゲノムワイドなDNA脱メチル化の起こる細胞や組織は限られている.たとえば,哺乳動物では生殖系列や受精後の初期胚など限られた細胞において能動的なDNA脱メチル化が起こる.このことは植物においても同様であり,中央細胞は胚珠組織のなかのたった1個の細胞にすぎない.したがって,生化学的な解析によりこの質問にせまるためには,エピゲノム解析における解析感度の劇的な向上と,細胞の効率的な単離法の確立が必要である.一方で,栄養組織においてDNA脱メチル化にはたらくROS1に関しても,いつどこでどの遺伝子をDNA脱メチル化しているかという質問に答えるのはむずかしい.このことは,いつどこでどの遺伝子がという問いに答えられる,すなわち,受精前に,中央細胞において,FWA遺伝子が,DNA脱メチル化されることの明らかなFWA-GFP遺伝子を用いた系において遺伝学的な解析を行うことが現状では効果的であることを意味している.そこで筆者らは,維持型DNAメチル化酵素MET1の変異体met1とDNA脱メチル化酵素DMEの変異体dmeとの二重ヘテロ接合体を作製し,FWA-GFP遺伝子の活性化にあたえる影響を解析した.その結果,中央細胞においてdme変異体はFWA-GFP遺伝子を活性化できないという表現型を,met1変異体は完全に抑圧することが示された.したがって,dme変異によりDNA脱メチル化のできなくなった細胞でも,あらかじめmet1変異によりDNAメチル化のレベルを低下させてやれば遺伝子発現はひき起こされることが示された.一方,FACTヒストンシャペロンの構成タンパク質をコードするSSRP1遺伝子の場合には,met1変異体とssrp1変異体との二重ヘテロ接合体では,中央細胞におけるmet1変異によるssrp1変異の抑圧効果は非常に弱く,さきほどのmet1変異体とdme変異体との二重へテロ接合体の結果とは明らかに異なっていた.このことから,DNAメチル化のほか,クロマチン機能がFWA遺伝子の転写に阻害的にはたらいていると考えることができ,ほかの生物において明らかになっているSSRP1遺伝子の機能26) とよく一致した.また,中央細胞において核が分裂をはじめると,それにともないFWA-GFP遺伝子の発現は徐々に強くなっていった.このことも,DNAメチル化が失われた状態で分裂をくり返すと,発現を示さないクロマチン情報も活性をもつクロマチン情報へと置き換わっていくことと一致していた23).以上の知見から,モデルをまとめた(図4).
中央細胞での機能が顕著なDNA脱メチル化酵素DMEについて,シロイヌナズナのdme変異体の胚乳画分におけるゲノムワイドなDNAメチル化解析がなされた.古くは,トウモロコシの胚乳を材料にDNAメチル化感受性酵素を用いてほかの組織とのDNAメチル化を比較した解析から,胚乳においては母親に由来するゲノムのDNAメチル化は低下することが示されていたが27),その詳細は明らかではなかった.ショットガンバイサルファイトシークエンス解析から,野生型のシロイヌナズナの胚乳画分ではすべてのカテゴリーのDNAメチル化のレベルが胚画分より低いことが示された4).また,注目すべき点は,胚画分におけるCpHpH配列のメチル化のレベルは地上部の栄養組織より高いことであった.それらの多くはトランスポゾン様配列のため,中央細胞や胚乳におけるDNA脱メチル化の結果,トランスポゾンの転写をひき起こし24塩基対の低分子RNAが蓄積し,それが卵細胞あるいは胚へ移行してトランスポゾンの抑制を強化する,という仮説が提唱されている2,4).最新の研究では,DMEの標的としているゲノム領域の詳細がみえてきている5).DMEはATリッチな500 bp以下の小さなトランスポゾンを標的としている.また,これらはヘテロクロマチン領域ではなく,ヌクレオソームの少ないユークロマチン領域に局在していることが示された.
DNA脱メチル化酵素の変異体として,栄養組織における機能の顕著なROS1遺伝子,DML2遺伝子,DML3遺伝子に対する三重変異体を用いたゲノムワイドなDNAメチル化解析もなされている4,5,28).くわえて,ROS1遺伝子の変異体の解析からも,これらのコードするDNA脱メチル化酵素は栄養組織において過剰に亢進した有害な5-メチルシトシンを取り除き,遺伝子発現を保証するはたらきをしていると理解されている16).これらの結果を考えあわせると,植物のDNAメチル化の情報もダイナミックに制御されていることがわかる.
植物のエピジェネティックな変異は後代に安定して伝達される例が多い29).DNAメチル化の情報が変化したエピジェネティック変異体は,生殖過程においてリプログラミングが起こると仮定すると,失われたDNAメチル化の情報ももとに戻るため後代に伝達されないことが予想される.このため,植物の生殖過程におけるDNAメチル化の情報のリプログラミングは,哺乳動物とは大きく異なることが予想されていた.また,さきに述べた中央細胞におけるDNA脱メチル化も,胚乳は役目をおえると退化する組織なので次世代には直接は影響しない.くわえて,受精にあずかる精核を形成する生殖系列や卵細胞への生殖系列において,微量な組織あるいは細胞に対する解析のむずかしさゆえ明確なリプログラミングに関する情報は乏しかった.しかしながら,過去には,タバコの花粉を5-メチルシトシンの抗体により染色した結果の報告が存在する30).精核の前駆細胞である雄原細胞においてDNAメチル化の著しく低下した像が得られており,リプログラミングの可能性を示唆していたが,抗体を使った実験のため正確な定量性に乏しくあまり考慮されてはいない.
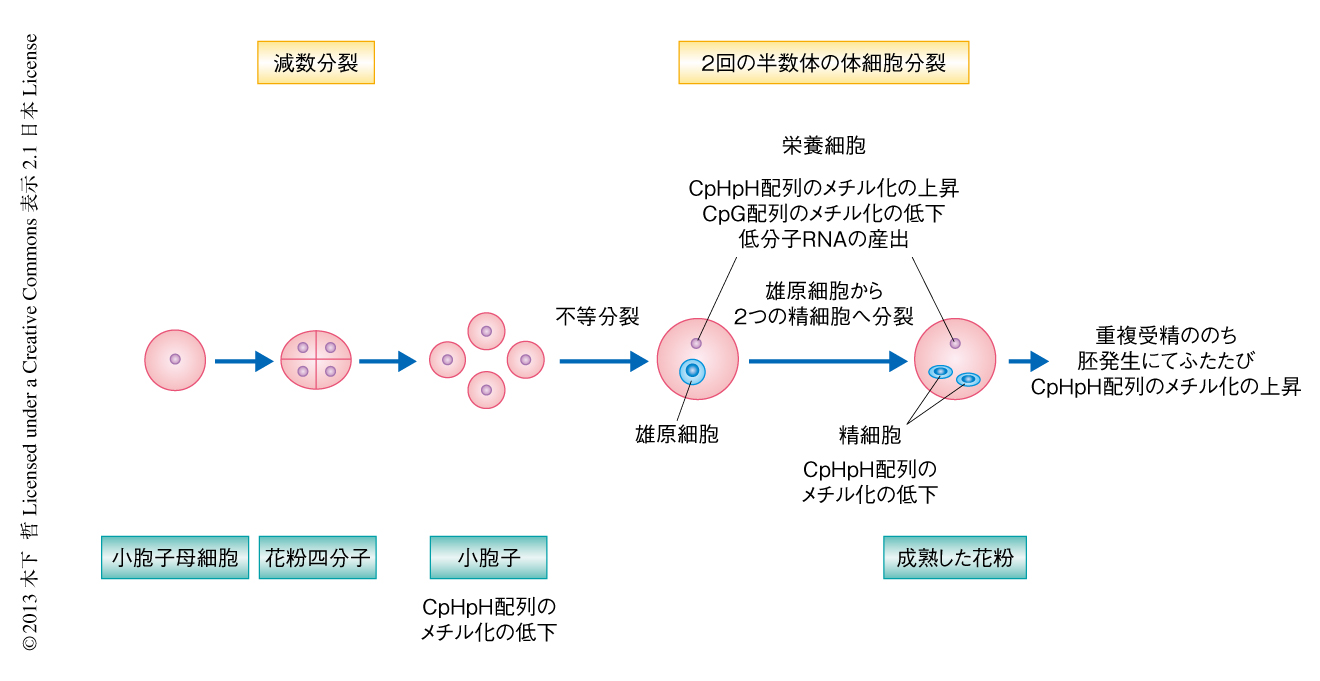
2009年に入ると,シロイヌナズナにおいてゲノムワイドなDNA低メチル化を誘導するクロマチンリモデリングタンパク質DDM1(decreased in DNA methylation)の変異体を用いて,いちど失われたDNAメチル化の情報が復帰する領域とそうでない領域とが存在することが明らかにされた31).野生型とDNAメチル化の低下したddm1変異体とを交配すると,ddm1変異に由来するゲノム領域にもふたたびDNAメチル化がみられるようになった.また,このことはRdDM経路の変異体では観察されないため,低分子RNAを介した機構であることが示された31).いつどこでという質問は残るものの,低分子RNAを介したDNAメチル化の補償作用のあることが明らかになった.また,雄性配偶体(花粉)において栄養細胞の核と精細胞の核とではそもそも核の大きさが異なることから,濃縮度の違いが考えられていた.シロイヌナズナでは,栄養細胞の核においてDDM1は蓄積しないこと,セントロメアに特異的なヒストンH3のバリアントは局在しないこと,花粉と精細胞の核に21塩基対の低分子RNAが蓄積すること,などがあいついで報告された32,33).これらのことから,栄養細胞ではゲノムワイドなDNA低メチル化の起こること,同時に観察される21塩基対の低分子RNAにより精細胞におけるトランスポゾンが抑制されること,がモデルとして提唱された32).
2012年に入り,このモデルを支持するばかりでなく,さらに画期的な発見を報告する論文が発表された34).花粉において栄養細胞の核と精細胞の核,さらには,両者の前駆細胞である小胞子細胞の核をセルソーターにより分収し,ゲノムワイドなDNAメチル化解析ならびに低分子RNAの解析が行われた.DNAメチル化解析では,ショットガンバイサルファイトシークエンスによるデータをもとに,DNAメチル化の程度に統計的に有意差のある領域(DMR:differentially methylated region)を決定している.その結果,CpG配列に関しては,これらの細胞のゲノムのあいだではDMRは明確に同定されるが,同定されたDMRは比較的少ないことがわかった.すなわち,哺乳動物の生殖系列ではCpG配列のメチル化がゲノムワイドに著しく低下することとは現象を異にしていた.また,CpHpG配列にはこれらの細胞において顕著なDMRは観察されていない.一方で,CpHpH配列に関しては小胞子細胞の段階ですでにそのDNAメチル化の情報が劇的に失われていることが明らかになった.このことは,de novo型DNAメチル化酵素DRM2の蓄積が小胞子細胞にみられないことからも支持された.一方,栄養細胞ではほかの細胞と比較してCpHpH配列のメチル化がふたたび高くなっており,このことはDRM2の局在パターンとも一致していたため,新規のDNAメチル化の起こっていることが予想された.すなわち,雄性配偶体(花粉)は,細胞のタイプごと,また,DNAメチル化のカテゴリーごとに異なる制御の起こっていることがわかる.
さて,つぎの質問は,雄性配偶体の形成過程においてインプリント遺伝子はどのような制御をうけるかである.栄養細胞のCpG配列は,DNA脱メチル化をうける程度は限定されるものの,精細胞とは明確な差がある.また,これらの多くはDNA脱メチル化酵素の標的配列とオーバーラップしていることが明らかになった34).これまでに,DNA脱メチル化酵素の花粉における機能は明確には報告されていなかったが,最近になり,dme変異体では花粉の発芽率が低下するという報告がなされた35).この表現型との因果関係は明らかになっていないが,栄養細胞と精細胞とでDNAメチル化を比較すると,インプリント遺伝子であるFWA遺伝子のシス制御領域,また,同じくインプリント遺伝子であるMEA遺伝子の5’側の領域と3’側の領域のCpG配列のメチル化が栄養細胞において低下していることが明らかになっている35).母親に由来する対立遺伝子に特異的に発現するFWA遺伝子やMEA遺伝子の活性化は,雌性配偶体の中央細胞に発現するDNA脱メチル化酵素をコードするDME遺伝子に依存している.一方で,DNA脱メチル化酵素が父親の側でもはたらいていることは,いっけん奇妙な現象である.しかしながらこれも,遺伝情報を伝達しない栄養細胞におけるDNA脱メチル化であることに注意する必要があるだろう.低分子RNAの蓄積を解析したデータでは,精細胞において母親に由来する対立遺伝子に特異的に発現するインプリント遺伝子のDMRにくわえて,父親に由来する対立遺伝子に特異的に発現するインプリント遺伝子のDMRに一致する,24塩基の低分子RNAの蓄積がみられる34).これらの低分子RNAの由来は栄養細胞であると考えられている.また,精細胞においてCpG配列あるいはCpHpH配列に対するDMRに一致する低分子RNAは,種子と比較して,21塩基の低分子RNAがより多く検出されている.これらの21塩基あるいは24塩基の低分子RNAは,栄養細胞の核において産出され精細胞の核へと移行して,それぞれトランスポゾンやインプリント遺伝子を制御していると考えられている.すなわち,雌性配偶体と同様に,遺伝情報を伝達する精細胞におけるエピゲノム情報をより強固に補強していると考えられている.
哺乳動物ではインプリント遺伝子のリプログラミングは生殖系列で起こる.生殖系列においては,前の世代のDNAメチル化の情報をいったん消去して,雌雄の生殖系列ごとにde novo型メチル化酵素によりDNAメチル化が書き込まれることによりリプログラミングは完了する.書き込まれたインプリント遺伝子のDNAメチル化の情報は,受精卵におけるゲノムワイドなDNA脱メチル化から保護され,体細胞へと受け継がれることにより片親性の発現が維持される2).一方で,シロイヌナズナの母親に由来する対立遺伝子に特異的なFWA遺伝子やMEA遺伝子に関して,父親に由来する対立遺伝子の不活性化はde novo型メチル化酵素をコードするDRM1遺伝子およびDRM2遺伝子には依存しないことが示されており9,36),DNAメチル化の書き込みのプロセスは必要ないと考えられている.つまり,動物および植物におけるゲノムインプリンティングでは,DNAメチル化はエピジェネティックな情報として共通して利用されているが,その制御機構は異なることが示されている.シロイヌナズナにおけるインプリント遺伝子のDNAメチル化の制御は,さきに述べたように,不活性化されている遺伝子を母親の側の中央細胞においてDNA脱メチル化することにより行われている.FWA遺伝子は,雄性配偶体におけるCpG配列のメチル化の維持が遺伝子の発現抑制に重要であることが示されているため37),精細胞のCpHpH配列におけるゲノムワイドなDNAメチル化の消失の影響はうけないと考えられ,結果と矛盾はない.一方で,最近のゲノムワイド解析により同定されたインプリント遺伝子であるSDC(Suppressor of drm1 drm2 cmt3)遺伝子は,プロモーター配列に存在する反復配列のシトシンがRdDM経路に依存してDNAメチル化されていることが知られている.おそらくは,この配列がSDC遺伝子のゲノムインプリンティングにおけるシス制御領域と考えられる.驚いたことに,SDC遺伝子のこの領域のDNAメチル化は,精細胞のCpHpH配列におけるゲノムワイドなDNAメチル化の消失から保護されていることが示された34).哺乳動物ではゲノムワイドなDNA脱メチル化から必要なメチル化情報を保護する機構が示されており,関連した機構が植物からもみつかる可能性がある.
受精後の胚発生のステージをおってさまざまな遺伝子のDNAメチル化について解析したデータから,胚発生ステージにおいてCpHpH配列のメチル化のレベルの上昇が観察されている38)(図5).また,このことはゲノムワイドなデータによっても裏づけられている4).生殖過程におけるエピジェネティックな制御が受精後のエピジェネティックな制御に影響する例として,ショウジョウバエの雑種致死現象(hybrid dysgenesis)があげられる.また,この現象はしばしば植物の生殖過程における低分子RNAの制御と比較して論じられている1).シロイヌナズナの胚発生の過程におけるCpHpH配列のメチル化はRdDMを介し低分子RNAにより制御されている38).また,胚乳発生に原因をもとめられる現象,たとえば,倍数体のあいだの交雑にも低分子RNAが関与しており,種子サイズの制御にかかわっていることが示唆されている39).種子の登熟の過程では母親に由来する低分子RNAが顕著に蓄積しており40),このこととの関連も興味深い.
これまで述べてきたように,次世代シークエンサーを用いたDNAメチル化解析などをとおして,植物の雄性配偶体の栄養細胞と精細胞という2種の細胞種における,3つのカテゴリーのDNAメチル化の制御が明らかになってきた.精細胞におけるCpHpH配列のメチル化の消失と受精後のリプログラミングの機構,生殖に対し付随的な機能をもつ栄養細胞におけるCpG配列のDNA脱メチル化の機構,そして,精細胞との低分子RNAのやりとりの結果もたらされるゲノムの安定性,これらの過程の生物学的な意義に関しても,今後ますます理解されることと予想される.これらの研究の突破口となったのは,いうまでもなく1細胞種レベルでの解析であった.つぎの課題として,さらに技術革新が必要と考えられるが,卵細胞へいたる細胞系譜におけるエピジェネティックな制御にせまる必要がある.おそらくそれは,そう遠くないうちに明らかになるであろう.
ここで解説した植物の生殖過程におけるエピジェネティックな制御機構は,シロイヌナズナのような均一なゲノム情報およびエピゲノム情報をもつモデル生物を中心に研究が進められてきた.一方で,今後は,われわれヒトを含め遺伝情報が均一ではない生物を対象に,集団および個体におけるエピゲノム情報に多様性をはらみながら,エピジェネティックな情報はどのように頑強性を維持しているのか,また,その多様性に軋轢が発生すると有性生殖にどのような破綻をきたすのか,それらが長い年月での進化の過程にどのように影響するのか,研究の対象も広がっていくものと思われる.とくに植物は,種間や倍数体間における雑種の形成が比較的容易であり,雑種強勢の機構など,それらの過程におけるエピジェネティックな制御機構の解明が今後の課題であると考える41).
略歴:1996年 総合研究大学院大学生命科学研究科博士後期課程 修了,1998年 米国California大学Berkeley校 博士研究員,2001年 国立遺伝学研究所 助手を経て,2007年より奈良先端科学技術大学院大学バイオサイエンス研究科 特任准教授(現 研究チーム長).
研究テーマ:植物の生殖過程におけるエピジェネティックな制御.ここでとりあげたように,エピゲノム情報がどのように維持され,植物の営みに影響をあたえているか,シロイヌナズナ,栽培イネと野生イネとの種間雑種などを用いて,多くの生物にあてはめられるような共通の原理機構の解明をめざす.
研究室URL:http://bsw3.naist.jp/courses/courses701.html
© 2013 木下 哲 Licensed under CC 表示 2.1 日本
(奈良先端科学技術大学院大学バイオサイエンス研究科 植物生殖遺伝学研究チーム)
email:木下 哲
領域融合レビュー, 2, e001 (2013) DOI: 10.7875/leading.author.2.e001
Tetsu Kinoshita: Epigenetic reprograming of maternal and paternal genomes during plant sexual reproduction.
要 約
近年のエピジェネティクスの研究の進捗により,DNA塩基配列以外の情報が遺伝情報としてはたらいていることへの理解は急速に進んだ.しかしながら,その全容は依然として混沌としており,多くの研究者を魅惑してやまない.ここでは,多くの生命現象のなかでも,エピジェネティクスととくに関連の深い植物の生殖過程をとりあげる.植物の複雑な生殖過程において,どのような分子機構によりエピジェネティックな情報が制御されているのかを解説し,その生物学的な意義を考察する.
はじめに
暖かな春の季節になると,多くの草花がわれわれの目を楽しませてくれる.一方で,植物にとって花を咲かせることは,つぎの世代へ種子を残すための重要な営みのひとつである.このような植物の生殖過程では,おしべとめしべの相互作用により,種のアイデンティティの維持や,集団における多様性の維持に関して,驚くべきダイナミックなせめぎあいが起こっている.エピジェネティックな情報を含めた遺伝情報をどのように“正しく”伝達するか,あるいは,ある“許容範囲内”での変化を伝えるのか,また,その結果,どのような個性や多様性が付与されるのか,はたまた,世代のくり返しのはてに植物の進化に対しどのような影響をあたえるのか,その詳細はほとんど明らかになっていない.研究の進捗により,DNAのメチル化,ヒストンの修飾,低分子RNAなど,DNAの塩基配列以外のエピジェネティックな情報そのものに対する理解は深まったが,その情報が生殖過程においてどのようにリプログラミング(書き換え)されるのか,現在は,新たな知見がつぎつぎと報告されるようになった段階である1,2).
一般的な植物のDNAメチル化制御に関してはほかに詳細な総説があるため3),ここでは簡単に述べるにとどめるが,植物には3つのカテゴリーのDNAメチル化部位が存在する.最近では,哺乳動物でもES細胞などごく一部の細胞においてCpG配列以外の配列でのDNAメチル化がみつかっているが,植物では多くの場合,CpG配列のほか,CpHpG配列およびCpHpH配列(H = C,T,A)のシトシン塩基の5位にメチル基が付加され5-メチルシトシンとなる.シロイヌナズナでは,CpG配列はおもにDNA複製にともない一方のDNA鎖のみがメチル化したヘミメチル化部位において維持型DNAメチル化酵素MET1によりメチル化される.CpHpG配列はヒストンH3の9番目のリジン残基のメチル化を担うヒストンメチル化酵素KYPのはたらきに依存して植物に特異的なクロモメチル化酵素CMT3によりメチル化される.CpHpH配列はRdDM(RNA-directed DNA methylation,RNA指向型DNAメチル化)の機構により低分子RNAを介してde novo型メチル化酵素DRMによりメチル化される3).
植物の生殖過程におけるエピジェネティックな情報,とりわけ,DNAのメチル化がどのように制御されるかを理解するにあたっては,被子植物の重複受精のしくみを理解することが必須である.したがって,まず,最後に参考図書としてあげた,“東山哲也: 植物の生殖細胞と受精戦略. 領域融合レビュー, 1, e007 (2012)”を熟読してほしい.また,RdDMの機構に関しても,ここでは誌面の都合からくわしくは解説しないので,もう一方の参考図書,“植物におけるRNAサイレンシング機構”を熟読してほしい.そのうえで,植物の生殖過程においては,雌雄の配偶体,受精後の胚発生および胚乳発生において,エピジェネティックな情報が制御されリプログラミングされることに理解を進めてほしい.以下,現在,明らかになりつつある,植物の生殖過程におけるエピジェネティックな制御を順に解説する.
1.雌性配偶体における母親に由来するゲノムのエピジェネティックな制御
雌性配偶体は,めしべの奥深くに存在する胚珠組織において,大胞子母細胞の減数分裂ののち1つだけ残される大胞子が3回の半数体の体細胞分裂をへることで成熟する.多くの被子植物は,卵細胞,中央細胞,2つの助細胞,3つの反足細胞の8核7細胞からなり,胚嚢(embryo sac)ともよばれる(図1).このうち,花粉により運ばれてくる2つの精細胞との受精にあずかるのは卵細胞と中央細胞であり,それぞれ胚と胚乳を形成する.これらの細胞のうち,中央細胞においてもっとも顕著にDNAメチル化によるエピジェネティックな情報の変化がみられることが知られている4,5).
中央細胞におけるエピジェネティックな制御の研究の発端となったのは,インプリント遺伝子としてMEDEA(MEA)遺伝子の発見である6,7).インプリント遺伝子は,その対立遺伝子が父親から由来したか母親から由来したかに応じて遺伝子発現のオン・オフが決定される.このような現象はおもに哺乳動物と被子植物においてみつかっており,ゲノムインプリンティングとよばれている8).MEA遺伝子や,機能未知のHD-ZIP型の転写因子をコードするFWA遺伝子などのインプリント遺伝子は,DEMETER(DME)遺伝子にコードされるDNA脱メチル化酵素により,受精前の雌性配偶体の中央細胞においてDNA脱メチル化され,遺伝子発現が誘導される9,10)(図2).最近では,MEA遺伝子に関してはDNA脱メチル化のほかにもクロマチンのループ構造を介したエピジェネティックな制御のあることがモデルとして提唱されている11).さらには,マウスと同様に,次世代シークエンサーを用いたSNP(single nucleotide polymorphism,一塩基多型)の解析から,父親に由来する対立遺伝子あるいは母親に由来する対立遺伝子に特異的に発現するインプリント遺伝子の多数の候補がみつかってきている12).古くは,インプリント遺伝子はシロイヌナズナを用いた変異体の研究により発見されてきた.シロイヌナズナでは,ポリコーム複合体の構成タンパク質をコードするMEA遺伝子とFIS2(Fertilization-Independent-Seed)遺伝子にゲノムインプリンティングがみつかっている.また,遺伝子は異なっていたものの,単子葉植物および双子葉植物においてポリコーム複合体構成タンパク質をコードする遺伝子のどれかがゲノムインプリンティングをうけていたため,哺乳動物および被子植物におけるインプリント遺伝子は,ある進化的な選択圧の結果として生じたとするコンフリクト仮説13,14) を支持していた.しかしながら,次世代シーケンサーを用いた解析の結果は,イネ,シロイヌナズナ,トウモロコシのあいだで驚くほどの多様性があった12).インプリント遺伝子がどのように生じてきたかは,今後,検証する必要があるが,いずれにせよ,植物の生殖過程におけるエピジェネティックな制御は,こうしたインプリント遺伝子の解析をモデルとして理解されるようになってきたといっても過言ではないだろう.
2.能動的なDNA脱メチル化の機構
DNA複製の過程では5-メチルシトシンは娘DNA鎖に取り込まれないため,DNAメチル化の維持機構がはたらかない場合にはDNA複製にともないDNAメチル化は減少していく.これを受動的DNA脱メチル化(passive DNA demethylation)とよぶ.一方,DNA複製に依存せずメチル基を積極的に取り除く機構も存在する.これを能動的なDNA脱メチル化(active DNA demethylation)とよぶ.
シロイヌナズナにおけるDME遺伝子の変異体の単離と,そののちの“能動的な”DNA脱メチル化酵素としてのDMEの研究は,植物の生殖過程におけるエピジェネティックなリプログラミングを知るうえでひとつの突破口となった15).また,哺乳動物におけるDNA脱メチル化の研究にも多大な影響を及ぼした16,17).DME遺伝子は比較的大きなタンパク質をコードするが,そのC末端側に塩基除去修復にかかわるDNAグリコシラーゼとよく似たドメイン構造をもつ18).シロイヌナズナにおいては4つのホモログ,DME遺伝子,DML2遺伝子,DML3遺伝子,ROS1遺伝子が存在する16).このうち,中央細胞において中心的な役割をはたしているのはDME遺伝子である19).単子葉植物であるイネにおいても,遺伝子ターゲティングの手法を用いてOsROS1a遺伝子を破壊すると,生殖過程にシビアな影響の生じることが示されている20).DMEをはじめ,これらのタンパク質にドメイン構造として存在するDNAグリコシラーゼは,多くの生物をつうじ塩基除去修復機構においてはたらく酵素として知られている.塩基除去修復機構では,まずG/Tなどミスマッチ塩基の認識とその切り出しが起こり,ヌクレオチド骨格の切断,修復DNAの合成とDNAリガーゼによるギャップの修復が起こり,DNA修復は完了する.シロイヌナズナにおけるROS1やDMEの生化学的な解析の結果,これらのタンパク質はグアニンと5-メチルシトシンとのペアをあたかもミスマッチペアのように認識し,5-メチルシトシン塩基の切り出しとヌクレオチド骨格の切断を行うことが示された.したがって,これらの酵素はDNA脱メチル化酵素であると理解されている.また,DNAリガーゼやDNAホスファターゼなど塩基除去修復機構にかかわる酵素が,このあとの段階においてDNA脱メチル化に必要であることが示されつつあり21,22),植物のDNA脱メチル化は塩基除去修復機構の分子機構と類似の経路により行われていることが理解されている(図3).
DNA脱メチル化の過程は,このような塩基除去修復機構による5-メチルシトシンの除去のほかにも,DNA脱メチル化配列の認識や決定,クロマチン構造やクロマチン機能の変化,あるいは,ヒストン修飾の変化をともなうことが予想される19).最近の研究により,これらの分子機構の一端がようやくみえはじめてきていた23,24).筆者らは,さきに述べた内在性のインプリント遺伝子であるFWA遺伝子の発現を可視化できるFWA-GFP遺伝子を利用して,ゲノムインプリンティングに異常のある変異体の選抜を行ってきた.FWA遺伝子は,その5’側の領域にSINEレトロトランスポゾンの挿入に由来すると考えられる反復配列が存在し,これがインプリンティングのシス制御領域であることが示されている25).この反復配列は,通常,高度にDNAメチル化をうけており遺伝子発現は抑制されているが,中央細胞においてDMEによりDNA脱メチル化をうけると遺伝子発現は誘導される9).変異体の選抜に用いたコンストラクトは,FWA遺伝子のゲノムインプリンティングの制御に必要なシス制御領域を含んでいる.
こうした系を用いて,HMGドメインをもつSSRP1(structure specific recognition protein 1)遺伝子がみつかった23)(新着論文レビュー でも掲載).SSRP1遺伝子はFACT(facilitate to chromatin transcription/transaction)ヒストンシャペロン複合体の構成タンパク質をコードしており26),酵母からヒトまで広く保存されている.SSRP1はSPT16とヘテロ二量体を形成し,転写の開始,転写伸張,DNA複製,DNA修復など,クロマチンの機能変換が必要な数多くの局面に登場するヒストンシャペロンであることが知られている26).ssrp1変異体は,FWA-GFP遺伝子を活性化できないことにくわえ,受精していないにもかかわらず鞘の伸張や中央細胞の核分裂がみられた.このような表現型は自発的な胚乳発生(autonomous endosperm development)とよばれ,ほかのインプリント遺伝子であるMEA遺伝子の変異体などにもみられる.そのため,FWA-GFP遺伝子の発現を指標に選抜した変異体ではあったが,SSRP1遺伝子はほかのインプリント遺伝子の発現にも影響することが考えられた.実際に,さまざまなインプリント遺伝子の発現を調べた結果,ssrp1変異体では多くのインプリント遺伝子の発現に影響のみられることがわかった.さらに,FWA遺伝子のDNAメチル化領域,すなわち,ゲノムインプリンティングのシス制御領域におけるDNA脱メチル化にも影響することがわかった23).
DNA脱メチル化研究の難問は,いつどこでどの遺伝子がどのようにDNA脱メチル化されるかという質問に生化学的にせまることがむずしいことにある.動物および植物をつうじて,ゲノムワイドなDNA脱メチル化の起こる細胞や組織は限られている.たとえば,哺乳動物では生殖系列や受精後の初期胚など限られた細胞において能動的なDNA脱メチル化が起こる.このことは植物においても同様であり,中央細胞は胚珠組織のなかのたった1個の細胞にすぎない.したがって,生化学的な解析によりこの質問にせまるためには,エピゲノム解析における解析感度の劇的な向上と,細胞の効率的な単離法の確立が必要である.一方で,栄養組織においてDNA脱メチル化にはたらくROS1に関しても,いつどこでどの遺伝子をDNA脱メチル化しているかという質問に答えるのはむずかしい.このことは,いつどこでどの遺伝子がという問いに答えられる,すなわち,受精前に,中央細胞において,FWA遺伝子が,DNA脱メチル化されることの明らかなFWA-GFP遺伝子を用いた系において遺伝学的な解析を行うことが現状では効果的であることを意味している.そこで筆者らは,維持型DNAメチル化酵素MET1の変異体met1とDNA脱メチル化酵素DMEの変異体dmeとの二重ヘテロ接合体を作製し,FWA-GFP遺伝子の活性化にあたえる影響を解析した.その結果,中央細胞においてdme変異体はFWA-GFP遺伝子を活性化できないという表現型を,met1変異体は完全に抑圧することが示された.したがって,dme変異によりDNA脱メチル化のできなくなった細胞でも,あらかじめmet1変異によりDNAメチル化のレベルを低下させてやれば遺伝子発現はひき起こされることが示された.一方,FACTヒストンシャペロンの構成タンパク質をコードするSSRP1遺伝子の場合には,met1変異体とssrp1変異体との二重ヘテロ接合体では,中央細胞におけるmet1変異によるssrp1変異の抑圧効果は非常に弱く,さきほどのmet1変異体とdme変異体との二重へテロ接合体の結果とは明らかに異なっていた.このことから,DNAメチル化のほか,クロマチン機能がFWA遺伝子の転写に阻害的にはたらいていると考えることができ,ほかの生物において明らかになっているSSRP1遺伝子の機能26) とよく一致した.また,中央細胞において核が分裂をはじめると,それにともないFWA-GFP遺伝子の発現は徐々に強くなっていった.このことも,DNAメチル化が失われた状態で分裂をくり返すと,発現を示さないクロマチン情報も活性をもつクロマチン情報へと置き換わっていくことと一致していた23).以上の知見から,モデルをまとめた(図4).
3.DNA脱メチル化酵素の変異体を利用したゲノムワイドなDNAメチル化解析
中央細胞での機能が顕著なDNA脱メチル化酵素DMEについて,シロイヌナズナのdme変異体の胚乳画分におけるゲノムワイドなDNAメチル化解析がなされた.古くは,トウモロコシの胚乳を材料にDNAメチル化感受性酵素を用いてほかの組織とのDNAメチル化を比較した解析から,胚乳においては母親に由来するゲノムのDNAメチル化は低下することが示されていたが27),その詳細は明らかではなかった.ショットガンバイサルファイトシークエンス解析から,野生型のシロイヌナズナの胚乳画分ではすべてのカテゴリーのDNAメチル化のレベルが胚画分より低いことが示された4).また,注目すべき点は,胚画分におけるCpHpH配列のメチル化のレベルは地上部の栄養組織より高いことであった.それらの多くはトランスポゾン様配列のため,中央細胞や胚乳におけるDNA脱メチル化の結果,トランスポゾンの転写をひき起こし24塩基対の低分子RNAが蓄積し,それが卵細胞あるいは胚へ移行してトランスポゾンの抑制を強化する,という仮説が提唱されている2,4).最新の研究では,DMEの標的としているゲノム領域の詳細がみえてきている5).DMEはATリッチな500 bp以下の小さなトランスポゾンを標的としている.また,これらはヘテロクロマチン領域ではなく,ヌクレオソームの少ないユークロマチン領域に局在していることが示された.
DNA脱メチル化酵素の変異体として,栄養組織における機能の顕著なROS1遺伝子,DML2遺伝子,DML3遺伝子に対する三重変異体を用いたゲノムワイドなDNAメチル化解析もなされている4,5,28).くわえて,ROS1遺伝子の変異体の解析からも,これらのコードするDNA脱メチル化酵素は栄養組織において過剰に亢進した有害な5-メチルシトシンを取り除き,遺伝子発現を保証するはたらきをしていると理解されている16).これらの結果を考えあわせると,植物のDNAメチル化の情報もダイナミックに制御されていることがわかる.
4.雄性配偶体におけるエピジェネティックな制御
植物のエピジェネティックな変異は後代に安定して伝達される例が多い29).DNAメチル化の情報が変化したエピジェネティック変異体は,生殖過程においてリプログラミングが起こると仮定すると,失われたDNAメチル化の情報ももとに戻るため後代に伝達されないことが予想される.このため,植物の生殖過程におけるDNAメチル化の情報のリプログラミングは,哺乳動物とは大きく異なることが予想されていた.また,さきに述べた中央細胞におけるDNA脱メチル化も,胚乳は役目をおえると退化する組織なので次世代には直接は影響しない.くわえて,受精にあずかる精核を形成する生殖系列や卵細胞への生殖系列において,微量な組織あるいは細胞に対する解析のむずかしさゆえ明確なリプログラミングに関する情報は乏しかった.しかしながら,過去には,タバコの花粉を5-メチルシトシンの抗体により染色した結果の報告が存在する30).精核の前駆細胞である雄原細胞においてDNAメチル化の著しく低下した像が得られており,リプログラミングの可能性を示唆していたが,抗体を使った実験のため正確な定量性に乏しくあまり考慮されてはいない.
2009年に入ると,シロイヌナズナにおいてゲノムワイドなDNA低メチル化を誘導するクロマチンリモデリングタンパク質DDM1(decreased in DNA methylation)の変異体を用いて,いちど失われたDNAメチル化の情報が復帰する領域とそうでない領域とが存在することが明らかにされた31).野生型とDNAメチル化の低下したddm1変異体とを交配すると,ddm1変異に由来するゲノム領域にもふたたびDNAメチル化がみられるようになった.また,このことはRdDM経路の変異体では観察されないため,低分子RNAを介した機構であることが示された31).いつどこでという質問は残るものの,低分子RNAを介したDNAメチル化の補償作用のあることが明らかになった.また,雄性配偶体(花粉)において栄養細胞の核と精細胞の核とではそもそも核の大きさが異なることから,濃縮度の違いが考えられていた.シロイヌナズナでは,栄養細胞の核においてDDM1は蓄積しないこと,セントロメアに特異的なヒストンH3のバリアントは局在しないこと,花粉と精細胞の核に21塩基対の低分子RNAが蓄積すること,などがあいついで報告された32,33).これらのことから,栄養細胞ではゲノムワイドなDNA低メチル化の起こること,同時に観察される21塩基対の低分子RNAにより精細胞におけるトランスポゾンが抑制されること,がモデルとして提唱された32).
2012年に入り,このモデルを支持するばかりでなく,さらに画期的な発見を報告する論文が発表された34).花粉において栄養細胞の核と精細胞の核,さらには,両者の前駆細胞である小胞子細胞の核をセルソーターにより分収し,ゲノムワイドなDNAメチル化解析ならびに低分子RNAの解析が行われた.DNAメチル化解析では,ショットガンバイサルファイトシークエンスによるデータをもとに,DNAメチル化の程度に統計的に有意差のある領域(DMR:differentially methylated region)を決定している.その結果,CpG配列に関しては,これらの細胞のゲノムのあいだではDMRは明確に同定されるが,同定されたDMRは比較的少ないことがわかった.すなわち,哺乳動物の生殖系列ではCpG配列のメチル化がゲノムワイドに著しく低下することとは現象を異にしていた.また,CpHpG配列にはこれらの細胞において顕著なDMRは観察されていない.一方で,CpHpH配列に関しては小胞子細胞の段階ですでにそのDNAメチル化の情報が劇的に失われていることが明らかになった.このことは,de novo型DNAメチル化酵素DRM2の蓄積が小胞子細胞にみられないことからも支持された.一方,栄養細胞ではほかの細胞と比較してCpHpH配列のメチル化がふたたび高くなっており,このことはDRM2の局在パターンとも一致していたため,新規のDNAメチル化の起こっていることが予想された.すなわち,雄性配偶体(花粉)は,細胞のタイプごと,また,DNAメチル化のカテゴリーごとに異なる制御の起こっていることがわかる.
5.雄性配偶体におけるインプリント遺伝子の制御
さて,つぎの質問は,雄性配偶体の形成過程においてインプリント遺伝子はどのような制御をうけるかである.栄養細胞のCpG配列は,DNA脱メチル化をうける程度は限定されるものの,精細胞とは明確な差がある.また,これらの多くはDNA脱メチル化酵素の標的配列とオーバーラップしていることが明らかになった34).これまでに,DNA脱メチル化酵素の花粉における機能は明確には報告されていなかったが,最近になり,dme変異体では花粉の発芽率が低下するという報告がなされた35).この表現型との因果関係は明らかになっていないが,栄養細胞と精細胞とでDNAメチル化を比較すると,インプリント遺伝子であるFWA遺伝子のシス制御領域,また,同じくインプリント遺伝子であるMEA遺伝子の5’側の領域と3’側の領域のCpG配列のメチル化が栄養細胞において低下していることが明らかになっている35).母親に由来する対立遺伝子に特異的に発現するFWA遺伝子やMEA遺伝子の活性化は,雌性配偶体の中央細胞に発現するDNA脱メチル化酵素をコードするDME遺伝子に依存している.一方で,DNA脱メチル化酵素が父親の側でもはたらいていることは,いっけん奇妙な現象である.しかしながらこれも,遺伝情報を伝達しない栄養細胞におけるDNA脱メチル化であることに注意する必要があるだろう.低分子RNAの蓄積を解析したデータでは,精細胞において母親に由来する対立遺伝子に特異的に発現するインプリント遺伝子のDMRにくわえて,父親に由来する対立遺伝子に特異的に発現するインプリント遺伝子のDMRに一致する,24塩基の低分子RNAの蓄積がみられる34).これらの低分子RNAの由来は栄養細胞であると考えられている.また,精細胞においてCpG配列あるいはCpHpH配列に対するDMRに一致する低分子RNAは,種子と比較して,21塩基の低分子RNAがより多く検出されている.これらの21塩基あるいは24塩基の低分子RNAは,栄養細胞の核において産出され精細胞の核へと移行して,それぞれトランスポゾンやインプリント遺伝子を制御していると考えられている.すなわち,雌性配偶体と同様に,遺伝情報を伝達する精細胞におけるエピゲノム情報をより強固に補強していると考えられている.
哺乳動物ではインプリント遺伝子のリプログラミングは生殖系列で起こる.生殖系列においては,前の世代のDNAメチル化の情報をいったん消去して,雌雄の生殖系列ごとにde novo型メチル化酵素によりDNAメチル化が書き込まれることによりリプログラミングは完了する.書き込まれたインプリント遺伝子のDNAメチル化の情報は,受精卵におけるゲノムワイドなDNA脱メチル化から保護され,体細胞へと受け継がれることにより片親性の発現が維持される2).一方で,シロイヌナズナの母親に由来する対立遺伝子に特異的なFWA遺伝子やMEA遺伝子に関して,父親に由来する対立遺伝子の不活性化はde novo型メチル化酵素をコードするDRM1遺伝子およびDRM2遺伝子には依存しないことが示されており9,36),DNAメチル化の書き込みのプロセスは必要ないと考えられている.つまり,動物および植物におけるゲノムインプリンティングでは,DNAメチル化はエピジェネティックな情報として共通して利用されているが,その制御機構は異なることが示されている.シロイヌナズナにおけるインプリント遺伝子のDNAメチル化の制御は,さきに述べたように,不活性化されている遺伝子を母親の側の中央細胞においてDNA脱メチル化することにより行われている.FWA遺伝子は,雄性配偶体におけるCpG配列のメチル化の維持が遺伝子の発現抑制に重要であることが示されているため37),精細胞のCpHpH配列におけるゲノムワイドなDNAメチル化の消失の影響はうけないと考えられ,結果と矛盾はない.一方で,最近のゲノムワイド解析により同定されたインプリント遺伝子であるSDC(Suppressor of drm1 drm2 cmt3)遺伝子は,プロモーター配列に存在する反復配列のシトシンがRdDM経路に依存してDNAメチル化されていることが知られている.おそらくは,この配列がSDC遺伝子のゲノムインプリンティングにおけるシス制御領域と考えられる.驚いたことに,SDC遺伝子のこの領域のDNAメチル化は,精細胞のCpHpH配列におけるゲノムワイドなDNAメチル化の消失から保護されていることが示された34).哺乳動物ではゲノムワイドなDNA脱メチル化から必要なメチル化情報を保護する機構が示されており,関連した機構が植物からもみつかる可能性がある.
6.受精後のエピジェネティックな制御
受精後の胚発生のステージをおってさまざまな遺伝子のDNAメチル化について解析したデータから,胚発生ステージにおいてCpHpH配列のメチル化のレベルの上昇が観察されている38)(図5).また,このことはゲノムワイドなデータによっても裏づけられている4).生殖過程におけるエピジェネティックな制御が受精後のエピジェネティックな制御に影響する例として,ショウジョウバエの雑種致死現象(hybrid dysgenesis)があげられる.また,この現象はしばしば植物の生殖過程における低分子RNAの制御と比較して論じられている1).シロイヌナズナの胚発生の過程におけるCpHpH配列のメチル化はRdDMを介し低分子RNAにより制御されている38).また,胚乳発生に原因をもとめられる現象,たとえば,倍数体のあいだの交雑にも低分子RNAが関与しており,種子サイズの制御にかかわっていることが示唆されている39).種子の登熟の過程では母親に由来する低分子RNAが顕著に蓄積しており40),このこととの関連も興味深い.
おわりに
これまで述べてきたように,次世代シークエンサーを用いたDNAメチル化解析などをとおして,植物の雄性配偶体の栄養細胞と精細胞という2種の細胞種における,3つのカテゴリーのDNAメチル化の制御が明らかになってきた.精細胞におけるCpHpH配列のメチル化の消失と受精後のリプログラミングの機構,生殖に対し付随的な機能をもつ栄養細胞におけるCpG配列のDNA脱メチル化の機構,そして,精細胞との低分子RNAのやりとりの結果もたらされるゲノムの安定性,これらの過程の生物学的な意義に関しても,今後ますます理解されることと予想される.これらの研究の突破口となったのは,いうまでもなく1細胞種レベルでの解析であった.つぎの課題として,さらに技術革新が必要と考えられるが,卵細胞へいたる細胞系譜におけるエピジェネティックな制御にせまる必要がある.おそらくそれは,そう遠くないうちに明らかになるであろう.
ここで解説した植物の生殖過程におけるエピジェネティックな制御機構は,シロイヌナズナのような均一なゲノム情報およびエピゲノム情報をもつモデル生物を中心に研究が進められてきた.一方で,今後は,われわれヒトを含め遺伝情報が均一ではない生物を対象に,集団および個体におけるエピゲノム情報に多様性をはらみながら,エピジェネティックな情報はどのように頑強性を維持しているのか,また,その多様性に軋轢が発生すると有性生殖にどのような破綻をきたすのか,それらが長い年月での進化の過程にどのように影響するのか,研究の対象も広がっていくものと思われる.とくに植物は,種間や倍数体間における雑種の形成が比較的容易であり,雑種強勢の機構など,それらの過程におけるエピジェネティックな制御機構の解明が今後の課題であると考える41).
文 献
- Bourc'his, D. & Voinnet, O.: A small-RNA perspective on gametogenesis, fertilization, and early zygotic development. Science, 330, 617-622 (2010)[PubMed]
- Feng, S., Jacobsen, S. E. & Reik, W.: Epigenetic reprogramming in plant and animal development. Science, 330, 622-627 (2010)[PubMed]
- Saze, H., Tsugane, K., Kanno, T. et al.: DNA methylation in plants: relationship to small RNAs and histone modifications, and functions in transposon inactivation. Plant Cell Physiol., 53, 766-784 (2012)[PubMed]
- Hsieh, T. F., Ibarra, C. A., Silva, P. et al.: Genome-wide demethylation of Arabidopsis endosperm. Science, 324, 1451-1454 (2009)[PubMed]
- Ibarra, C. A., Feng, X., Schoft, V. K. et al.: Active DNA demethylation in plant companion cells reinforces transposon methylation in gametes. Science, 337, 1360-1364 (2012)[PubMed]
- Kinoshita, T., Yadegari, R., Harada, J. J. et al.: Imprinting of the MEDEA polycomb gene in the Arabidopsis endosperm. Plant Cell, 11, 1945-1952 (1999)[PubMed]
- Grossniklaus, U., Vielle-Calzada, J. P., Hoeppner, M. A. et al.: Maternal control of embryogenesis by MEDEA, a polycomb group gene in Arabidopsis. Science, 280, 446-450 (1998)[PubMed]
- Kinoshita, T., Ikeda, Y. & Ishikawa, R.: Genomic imprinting: a balance between antagonistic roles of parental chromosomes. Semin. Cell Dev. Biol., 19, 574-579 (2008)[PubMed]
- Kinoshita, T., Miura, A., Choi, Y. et al.: One-way control of FWA imprinting in Arabidopsis endosperm by DNA methylation. Science, 303, 521-523 (2004)[PubMed]
- Gehring, M., Huh, J. H., Hsieh, T. F. et al.: DEMETER DNA glycosylase establishes MEDEA polycomb gene self-imprinting by allele-specific demethylation. Cell, 124, 495-506 (2006)[PubMed]
- Wohrmann, H. J., Gagliardini, V., Raissig, M. T. et al.: Identification of a DNA methylation-independent imprinting control region at the Arabidopsis MEDEA locus. Genes Dev., 26, 1837-1850 (2012)[PubMed]
- Ikeda, Y.: Plant imprinted genes identified by genome-wide approaches and their regulatory mechanisms. Plant Cell Physiol., 53, 809-816 (2012)[PubMed]
- Haig, D. & Westoby, M.: Genomic imprinting in endosperm: its effect on seed development in crosses between species, and between different ploidies of the same species, and its implications for the evolution of apomixis. Phil. Trans. R. Soc. Lond. B., 333, 1-14 (1991)
- Moore, T. & Haig, D.: Genomic imprinting in mammalian development: a parental tug-of-war. Trends Genet., 7, 45-49 (1991)[PubMed]
- Choi, Y., Gehring, M., Johnson, L. et al.: DEMETER, a DNA glycosylase domain protein, is required for endosperm gene imprinting and seed viability in Arabidopsis. Cell, 110, 33-42 (2002)[PubMed]
- Zhu, J. K.: Active DNA demethylation mediated by DNA glycosylases. Annu. Rev. Genet., 43, 143-166 (2009)[PubMed]
- Franchini, D. M., Schmitz, K. M. & Petersen-Mahrt, S. K.: 5-methylcytosine DNA demethylation: more than losing a methyl group. Annu. Rev. Genet., 46, 419-441 (2012)[PubMed]
- Mok, Y. G., Uzawa, R., Lee, J. et al.: Domain structure of the DEMETER 5-methylcytosine DNA glycosylase. Proc. Natl. Acad. Sci. USA, 107, 19225-19230 (2010)[PubMed]
- Ikeda, Y. & Kinoshita, T.: DNA demethylation: a lesson from the garden. Chromosoma, 118, 37-41 (2009)[PubMed]
- Ono, A., Yamaguchi, K., Fukada-Tanaka, S. et al.: A null mutation of ROS1a for DNA demethylation in rice is not transmittable to progeny. Plant J., 71, 564-574 (2012)[PubMed]
- Andreuzza, S., Li, J., Guitton, A. E. et al.: DNA LIGASE I exerts a maternal effect on seed development in Arabidopsis thaliana. Development, 137, 73-81 (2010)[PubMed]
- Martinez-Macias, M. I., Qian, W., Miki, D. et al.: A DNA 3' phosphatase functions in active DNA demethylation in Arabidopsis. Mol. Cell, 45, 357-370 (2012)[PubMed]
- Ikeda, Y., Kinoshita, Y., Susaki, D. et al.: HMG domain containing SSRP1 is required for DNA demethylation and genomic imprinting in Arabidopsis. Dev. Cell, 21, 589-596 (2011)[PubMed] [新着論文レビュー]
- Qian, W., Miki, D., Zhang, H. et al.: A histone acetyltransferase regulates active DNA demethylation in Arabidopsis. Science, 336, 1445-1448 (2012)[PubMed]
- Kinoshita, Y., Saze, H., Kinoshita, T. et al.: Control of FWA gene silencing in Arabidopsis thaliana by SINE-related direct repeats. Plant J., 49, 38-45 (2007)[PubMed]
- Formosa, T.: The role of FACT in making and breaking nucleosomes. Biochim. Biophys. Acta, 1819, 247-255 (2012)[PubMed]
- Lauria, M., Rupe, M., Guo, M. et al.: Extensive maternal DNA hypomethylation in the endosperm of Zea mays. Plant Cell, 16, 510-522 (2004)[PubMed]
- Penterman, J., Zilberman, D., Huh, J. H. et al.: DNA demethylation in the Arabidopsis genome. Proc. Natl. Acad. Sci. USA, 104, 6752-6757 (2007)[PubMed]
- Kakutani, T.: Epi-alleles in plants: inheritance of epigenetic information over generations. Plant Cell Physiol., 43, 1106-1111 (2002)[PubMed]
- Oakeley, E. J., Podesta, A. & Jost, J. P.: Developmental changes in DNA methylation of the two tobacco pollen nuclei during maturation. Proc. Natl. Acad. Sci. USA, 94, 11721-11725 (1997)[PubMed]
- Teixeira, F. K., Heredia, F., Sarazin, A. et al.: A role for RNAi in the selective correction of DNA methylation defects. Science, 323, 1600-1604 (2009)[PubMed]
- Slotkin, R. K., Vaughn, M., Borges, F. et al.: Epigenetic reprogramming and small RNA silencing of transposable elements in pollen. Cell, 136, 461-472 (2009)[PubMed]
- Schoft, V. K., Chumak, N., Mosiolek, M. et al.: Induction of RNA-directed DNA methylation upon decondensation of constitutive heterochromatin. EMBO Rep., 10, 1015-1021 (2009)[PubMed]
- Calarco, J. P., Borges, F., Donoghue, M. T. et al.: Reprogramming of DNA methylation in pollen guides epigenetic inheritance via small RNA. Cell, 151, 194-205 (2012)[PubMed]
- Schoft, V. K., Chumak, N., Choi, Y. et al.: Function of the DEMETER DNA glycosylase in the Arabidopsis thaliana male gametophyte. Proc. Natl. Acad. Sci. USA, 108, 8042-8047 (2011)[PubMed]
- Cao, X. & Jacobsen, S. E.: Role of the Arabidopsis DRM methyltransferases in de novo DNA methylation and gene silencing. Curr. Biol., 12, 1138-1144 (2002)[PubMed]
- Saze, H., Mittelsten Scheid, O. & Paszkowski, J.: Maintenance of CpG methylation is essential for epigenetic inheritance during plant gametogenesis. Nat. Genet., 34, 65-69 (2003)[PubMed]
- Jullien, P. E., Susaki, D., Yelagandula, R. et al.: DNA methylation dynamics during sexual reproduction in Arabidopsis thaliana. Curr Biol 22, 1825-1830 (2012)[PubMed]
- Chen, H. M., Chen, L. T., Patel, K. et al.: 22-Nucleotide RNAs trigger secondary siRNA biogenesis in plants. Proc. Natl. Acad. Sci. USA, 107, 15269-15274 (2010)[PubMed]
- Mosher, R. A., Melnyk, C. W., Kelly, K. A. et al.: Uniparental expression of PolIV-dependent siRNAs in developing endosperm of Arabidopsis. Nature, 460, 283-286 (2009)[PubMed]
- Ishikawa, R. & Kinoshita, T.: Epigenetic programming: the challenge to species hybridization. Mol. Plant, 2, 589-599 (2009)[PubMed]
参考図書
吉川 学, 菅野達夫, 土生芳樹: 植物におけるRNAサイレンシング機構:転写後型ジーンサイレンシング(PTGS)と転写型ジーンサイレンシング(TGS). 細胞工学, 30, 706-711 (2011)
東山哲也: 植物の生殖細胞と受精戦略. 領域融合レビュー, 1, e007 (2012)
著者プロフィール
略歴:1996年 総合研究大学院大学生命科学研究科博士後期課程 修了,1998年 米国California大学Berkeley校 博士研究員,2001年 国立遺伝学研究所 助手を経て,2007年より奈良先端科学技術大学院大学バイオサイエンス研究科 特任准教授(現 研究チーム長).
研究テーマ:植物の生殖過程におけるエピジェネティックな制御.ここでとりあげたように,エピゲノム情報がどのように維持され,植物の営みに影響をあたえているか,シロイヌナズナ,栽培イネと野生イネとの種間雑種などを用いて,多くの生物にあてはめられるような共通の原理機構の解明をめざす.
研究室URL:http://bsw3.naist.jp/courses/courses701.html
© 2013 木下 哲 Licensed under CC 表示 2.1 日本