RNA結合タンパク質と自然免疫
2013/11/26
竹内 理
(京都大学ウイルス研究所 感染防御研究分野)
email:竹内 理
領域融合レビュー, 2, e013 (2013) DOI: 10.7875/leading.author.2.e013
Osamu Takeuchi: Innate immunity and RNA binding proteins.

自然免疫は,病原体やストレスに対する内因性の物質をToll様受容体などの受容体により認識し,初期の炎症応答を起こす機構である.自然免疫によりマクロファージなどから産生される炎症性サイトカインは,炎症の惹起および制御に重要な役割をはたしている.Toll様受容体からのシグナルによるサイトカイン遺伝子をはじめとした炎症に関連する遺伝子の発現は,転写だけではなく,転写後のmRNAの分解および翻訳などによっても緻密に制御され,適切な炎症応答を保っている.mRNAの分解および翻訳の抑制にかかわることの知られるマイクロRNAにくわえ,mRNAの不安定化を促進するTristetraprolinやRNA分解酵素であるRegnase-1といったmRNAの3’側非翻訳領域に結合するRNA結合タンパク質について,炎症の制御における重要性が明らかになってきた.細胞内シグナル伝達系によりRegnase-1の発現は直接に制御されることから,転写だけでなくmRNAの安定性についても,細胞の外からのシグナルにより直接に制御されていると考えられる.ここでは,炎症の転写後制御の分子機構に焦点をあて解説する.
免疫系は大きく自然免疫と獲得免疫とに分類することができる.獲得免疫がT細胞やB細胞といった遺伝子の再構成を必要とする受容体により非自己を認識する機構をもち免疫記憶において重要であるのに対し,自然免疫はマクロファージや樹状細胞などにより担われ,ゲノムにコードされた受容体により病原体を認識し,初期における病原体の排除や炎症応答に重要な機構である1).また,自然免疫は樹状細胞を介し獲得免疫の活性化においても重要な役割をはたしている.炎症の惹起および制御には,インターロイキン1,インターロイキン6,TNF(tumor necrosis factor,腫瘍壊死因子)など炎症性サイトカインや,I型インターフェロンのかかわることが知られている.これらのサイトカインは病原体の感染により,活性化したマクロファージや樹状細胞などから産生される.
自然免疫は宿主に存在する受容体が病原微生物の構成成分(PAMP:pathogen-associated molecular pattern,病原微生物関連分子パターン)を検出することにより誘導される.このようなPAMPを認識するパターン認識受容体(pattern recognition receptor:PRR)として,Toll様受容体(Toll-like receptor:TLR),RIG-I様受容体(RIG-I-like receptor:RLR),NOD様受容体(NOD-like receptor:NLR),C型レクチン受容体(C-type lectin receptor:CLR)が知られている1).このうち,Toll様受容体およびC型レクチン受容体はI型膜タンパク質で細胞膜もしくはエンドソーム膜に局在し,微生物,あるいは,内在性の脂質,リポタンパク質,DNA,RNAといったさまざまなPAMPを認識する.これに対し,RIG-I様受容体は膜貫通領域をもたず細胞質内に局在する2本鎖RNA結合タンパク質であり,細胞内におけるRNAウイルスの感染の認識において重要である.NOD様受容体も細胞質に存在する受容体で,ペプチドグリカンやフラジェリンなど一部はToll様受容体と共通したPAMPの認識にかかわる.
インフラマソームを活性化する一部のNOD様受容体を除き,ほとんどのパターン認識受容体は細胞内シグナル伝達系を介して遺伝子発現を活性化する.たとえば,Toll様受容体はTLR3を除き,その細胞質内領域を介してアダプタータンパク質であるMyD88と結合し,さらに,IRAKを介して細胞内にシグナルを伝達し,TRAF6などのポリユビキチンシグナルタンパク質をへてIκBキナーゼ(IKK)を活性化する.IκBキナーゼはIKKα,IKKβ,NEMO/IKKγより構成され,NF-κB阻害タンパク質であるIκBをリン酸化し,ユビキチン-プロテオソーム系によるIκBの分解を誘導する.その結果,IκBから遊離したNF-κBは核へと移行し,炎症に関連する遺伝子のプロモーター領域に結合してその転写を活性化する.RIG-I様受容体はアダプタータンパク質としてIPS-1を介して細胞内シグナル伝達系を活性化するが,その下流ではIκBキナーゼを介してNF-κBの核への移行を促進するなど,共通のエフェクタータンパク質を活性化させている.また,NOD様受容体の一部やC型レクチン受容体からのシグナルもNF-κBを活性化させることが知られており,パターン認識受容体からのシグナルはその多くが下流に共通のモジュールをもつと考えられる.Toll様受容体の一部およびRIG-I様受容体からのシグナルは転写因子であるIRF3やIRF7(IRF:interferon-regulatory factor)を介してI型インターフェロンを産生させる.
このように,自然免疫においてパターン認識受容体から転写の活性化にいたる細胞内シグナル伝達系や,エピジェネティックな転写の制御機構に関しては,これまで詳細な解析がなされてきた.しかし,サイトカインの産生の制御は転写だけでは説明がつかず,マイクロRNAやさまざまなRNA結合タンパク質による転写後制御の重要性が明らかになってきている.ここでは,マイクロRNAに関する解説はほかにゆずり2),RNA結合タンパク質と遺伝子の発現制御との関連に焦点をあてる.
サイトカインや増殖因子をコードするmRNAには,その3’側非翻訳領域にAUを多く含むAUリッチ配列(典型的には,AUUUA)が高頻度に存在することが知られている.また,この配列の存在の頻度とmRNAの不安定性とが相関することも報告されている.たとえば,TNFの刺激に対し早期に誘導される遺伝子のmRNAの3’側非翻訳領域は多くのAUリッチ配列をもつことが報告されており,これらのmRNAはより速く分解されると考えられている3).これまで,AUリッチ配列に結合するタンパク質としては,Tristetraprolin(TTP)やそのファミリータンパク質であるZfp36L1,Zfp36L2,また,AUF1,KSRP,HuRなど,多くが知られている4).
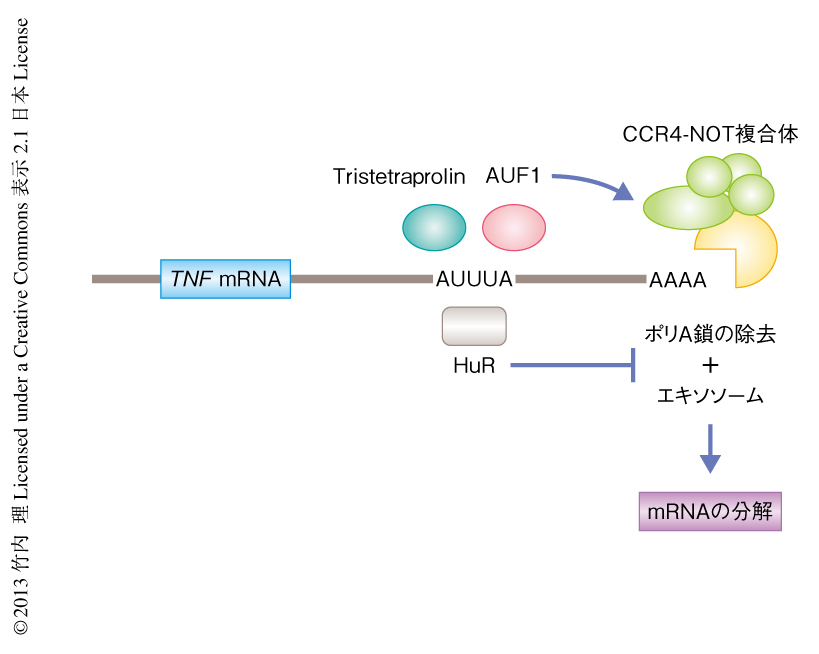
TristetraprolinはToll様受容体への刺激により早期に誘導されるタンパク質であり,2つのCCCH型ジンクフィンガー領域をもつ.この領域はRNA結合ドメインとして知られ,mRNAの3’側非翻訳領域に存在するAUリッチ配列との結合において重要である.TristetraprolinはTNFをコードするmRNAをAUリッチ配列を介し不安定化する(図1).これまでに,Tristetraprolinはインターフェロンγやインターロイキン10をはじめさまざまなサイトカインのmRNAの安定性の制御にかかわることが報告されている.加齢したTristetraprolinの欠損マウスはTNFの過剰な産生により関節炎を発症する5).TristetraprolinはTNFをコードするmRNAの3’側非翻訳領域に結合し,脱アデニル化酵素を含むCCR4-NOT複合体をリクルートすることにより,mRNAからポリA鎖が除去される.ポリA鎖の除去されたmRNAはつづいてキャップ構造が除去され,XRN1やエクソソームなどのRNA分解酵素により分解される.AUF1はTNF,インターロイキン1β,インターロイキン6などのサイトカインのmRNAの3’側非翻訳領域にAUリッチ配列を介して結合し,これらのmRNAを不安定化することが知られている4).AUF1を欠損するマウスはリポ多糖によるショックに対し野生型マウスよりも感受性であり,TNFやインターロイキン1βといった炎症性サイトカインを過剰に産生することが報告されている.KSRPはRNAスプライシングやマイクロRNAの産生などにかかわるタンパク質であるが,AUリッチ配列に結合しサイトカインをコードするmRNAを不安定化するほか,その翻訳を抑制することが報告されている6).これに対し,同様なAUリッチ配列結合タンパク質であるHuRは,AUリッチ配列をもつmRNAを細胞質において安定化することが知られている7).AUリッチ配列結合タンパク質は自然免疫だけでなく獲得免疫の制御にもかかわることが報告されている.その例として,Zfp36l1とZfp36l2は胸腺におけるT細胞の分化に必要であり,また,急性T細胞白血病の発症を抑制している.また,Zfp36L2は赤芽球系前駆細胞の自己複製にかかわることが知られている8).
このように,AUリッチ配列結合タンパク質は炎症性サイトカインの産生を正に負に制御し,炎症のバランスをとっていることが明らかになってきた.
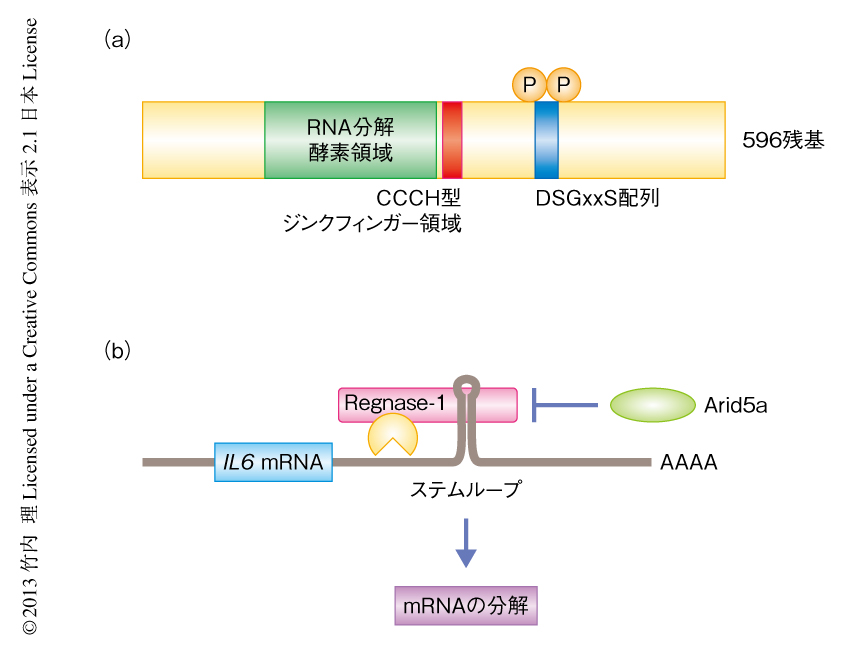
mRNAの安定性はAUリッチ配列のみで制御されているわけではない.Zc3h12a遺伝子はマクロファージにおいてToll様受容体からのシグナルにより発現が誘導され,この遺伝子のコードするタンパク質はRNase領域およびCCCH型ジンクフィンガー領域をもつ.また,このタンパク質はインターロイキン6をコードするmRNAと結合し,細胞に強制発現するとそのRNA分解酵素活性に依存してこのmRNAを分解した.また,のちに述べるように,このタンパク質は免疫系の制御においても重要であったことから,筆者らは,Regnase-1(regulatory RNase-1)と命名した(図2a).Regnase-1はインターロイキン6をコードするmRNAの3’側非翻訳領域に存在するステムループ領域を介してこのmRNAを不安定化した9)(図2b).また,Regnase-1を欠損したマクロファージにおいては,Toll様受容体への刺激に応答したインターロイキン6やインターロイキン12 p40などの産生が亢進していた.Regnase-1ノックアウトマウスはメンデルの法則にしたがって生まれるが成長の遅延を示し,生後3週目ほどで死亡しはじめ,12週までにはほぼ全例が死亡した.Regnase-1ノックアウトマウスは血清においてさまざまなクラスの免疫グロブリンが増加しており,また,抗核抗体や抗2本鎖DNA抗体など自己抗体を産生していた.Regnase-1の欠損のもとでは形質細胞の増加,また,エフェクターT細胞の増加が観察された.Regnase-1ノックアウトマウスの組織を観察すると,肺などの組織へのリンパ球,とくに,形質細胞の浸潤が著明であり,肺において免疫グロブリンG産生細胞や免疫グロブリンA産生細胞の著明な増加が認められた.
Regnase-1の欠損により炎症が惹起される分子機構を検討するため,Cre-loxP系を用いて細胞種に特異的なRegnase-1ノックアウトマウスを作製した.T細胞に特異的にRegnase-1を欠損させたところ,全身におけるRegnase-1ノックアウトマウスと同様の炎症性疾患を発症し,8週をこえてから自然に死亡しはじめた.また,このノックアウトマウスにもさまざまな臓器への炎症細胞の浸潤や著明な脾腫が認められ,エフェクターT細胞および形質細胞が増加していた.また,T細胞受容体への刺激によるCD4陽性T細胞からのサイトカインの産生も亢進しており,T細胞に発現するRegnase-1がT細胞の活性化を抑制する役割をはたしていることが明らかになった.CD4陽性T細胞におけるRegnase-1の標的mRNAについて検討したところ,サイトカインであるインターロイキン2や,細胞表面タンパク質であるICOSおよびOX40,転写因子のなかでもc-RelをコードするmRNAが分解されていることが明らかになり,Regnase-1は細胞種ごとに特徴的なmRNAを標的として免疫細胞の活性化を制御していると考えられた10).
以上の結果から,Regnase-1は免疫の活性化にかかわるタンパク質をコードするmRNAをRNA分解酵素として直接に分解すること,また,自然免疫および獲得免疫にはたらく細胞において,自己免疫性の炎症性疾患の発症を抑制するため重要なタンパク質であることが明らかになった.最近になり,Regnase-1と逆に,インターロイキン6をコードするmRNAを3’側非翻訳領域を介して安定化するタンパク質Arid5aの存在も報告されている11).
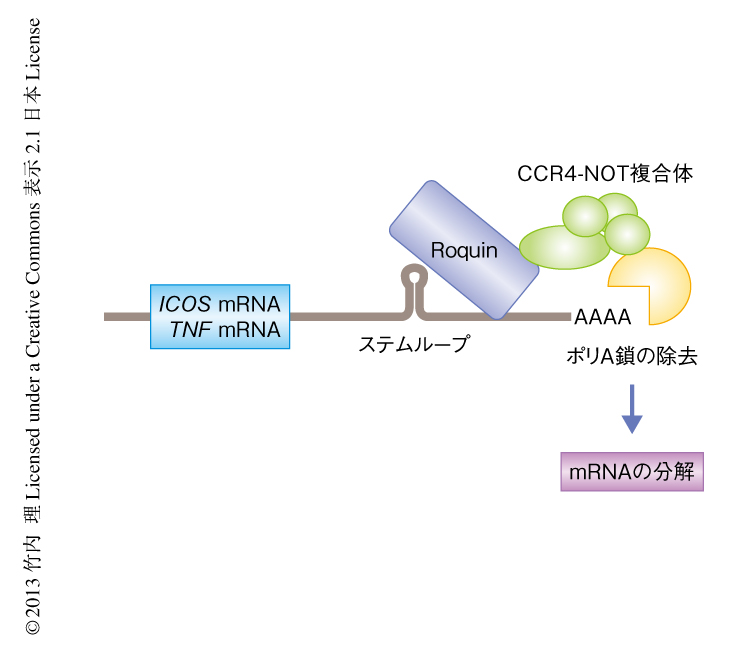
Roquinは,エチルニトロソ尿素の投与による変異により自己免疫疾患を発症するようになったマウス系統において,その原因遺伝子にコードされるタンパク質として見い出された12).この変異マウスは,自己抗体の産生,脾臓における胚中心や濾胞性ヘルパーT細胞の増加,T細胞におけるICOSの発現の増加を呈した.RoquinはRc3h1遺伝子にコードされ,ユビキチンリガーゼ活性をもつと考えられるRINGフィンガー領域にくわえ,RNAと結合するCCCH型ジンクフィンガー領域,および,ROQドメインをもつ.RoquinはICOSなどをコードするmRNAに直接もしくはマイクロRNAを介し間接的に結合し,その不安定化を誘導すると考えられている.Roquinは,標的RNAに脱アデニル化酵素を含むCCR4-NOT複合体をリクルートすることにより,その分解を促進する(図3).また,RoquinはTNFをコードするmRNAの3’側非翻訳領域に存在するステムループ構造に結合するとの報告もあり,Roquinに変異をもつマウスではリポ多糖の投与に対するTNFの産生が亢進している13).したがって,Regnase-1と同様に,Roquinは獲得免疫および自然免疫をともに制御するRNA結合タンパク質であると考えられる.
さきに述べたように,転写は細胞の外からの刺激により細胞内シグナル伝達系を介して緻密に制御されていることがよく知られている.くわえて,いくつかの研究により,mRNAの安定性それ自体も細胞の外からの刺激により制御をうけていることが明らかになりつつある.
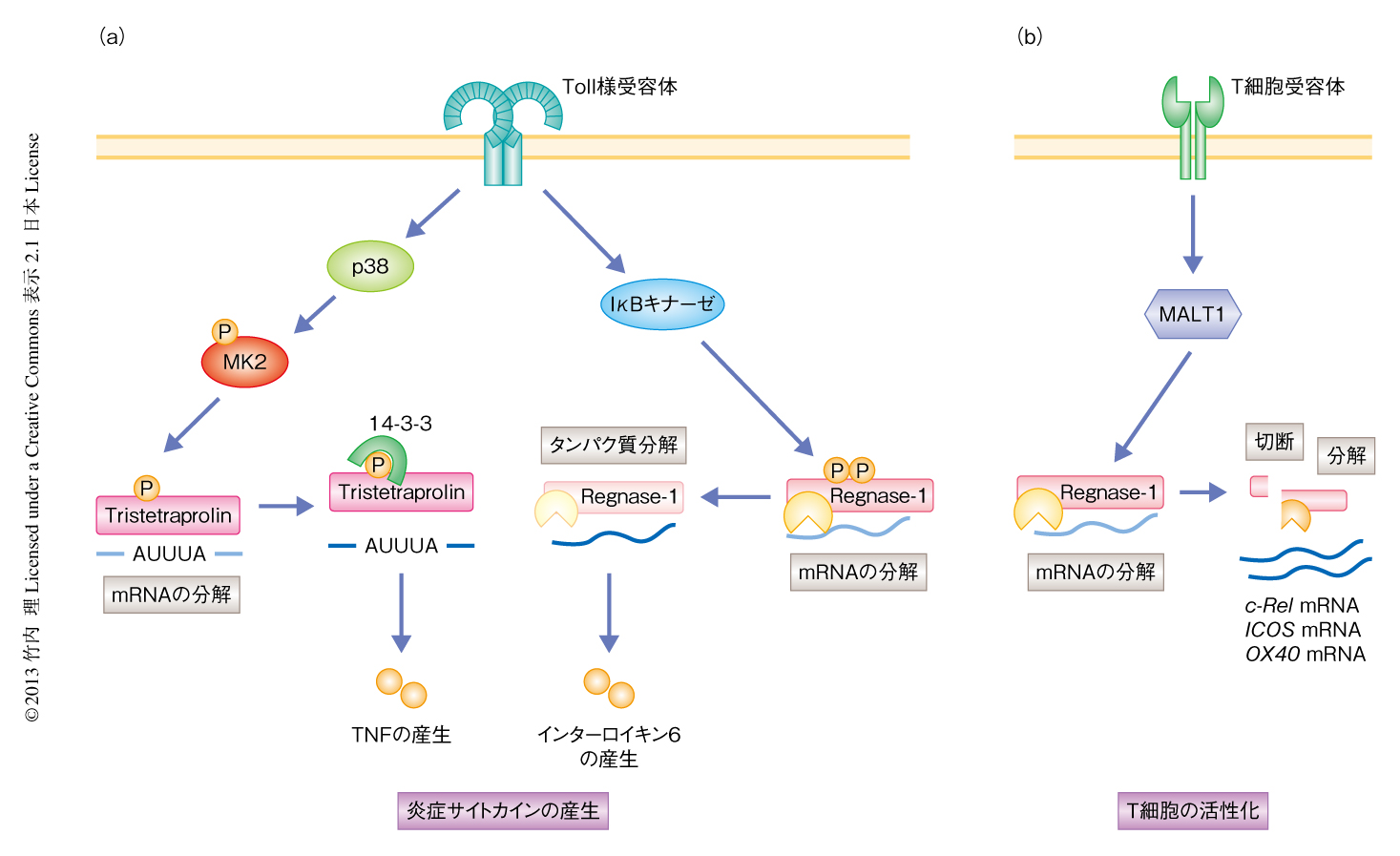
たとえば,自然免疫により活性化されるMAPキナーゼファミリーのひとつであるp38 MAPキナーゼは,AUリッチ配列をもつRNAを安定化することが知られている.この分子機構のひとつとして,p38 MAPキナーゼがMK2を活性化し,このMK2がTristetraprolinをリン酸化することがあげられる.リン酸化されたTristetraprolinは14-3-3とよばれるタンパク質と結合することにより,TNFなどをコードするmRNAを不安定化する活性が低下すると考えられている(図4a).
Regnase-1もタンパク質の修飾を介して免疫の活性化によるダイナミックな制御をうけている.Regnase-1は定常状態においても免疫細胞に発現しており,これは,マクロファージにおいてインターロイキン6をコードするmRNAの発現を完全に抑制するために重要である.このことは,Regnase-1を欠損した細胞においては定常状態でも野生型の細胞と比較してインターロイキン6のmRNAが高く発現していることからも明らかである.Toll様受容体への刺激あるいはインターロイキン1βによるMyD88を介した刺激に対して,Regnase-1がIκBキナーゼ複合体により直接にリン酸化され,ユビキチン-プロテオソーム系により早期に分解されることが明らかになった14).Regnase-1にはIκBキナーゼによりリン酸化をうけるAsp-Ser-Gly-X-X-Serというアミノ酸配列が保存されており(図2a),IκBキナーゼを欠損する細胞ではToll様受容体への刺激に対するRegnase-1の分解はみられなくなるほか,リン酸化をうける2つのセリン残基に変異を導入した場合にはRegnase-1の分解は起こらなくなり,また,インターロイキン6をコードするmRNAの産生をより強く抑制した.このように,Toll様受容体-IκBキナーゼ経路は,NF-κBを介した転写の活性化(アクセル)のみならず,Regnase-1を分解することで転写に対するブレーキを解除することにより,インターロイキン6などの炎症性サイトカインの迅速な産生を実現していると考えられる(図4a).また,Regnase-1はマクロファージにおいて分解されたのち,数時間で再発現してくる.この再発現にはRegnase-1の転写の活性化がかかわっているが,それにくわえ,Regnase-1をコードするmRNAの安定性も増加していることが明らかになった.Regnase-1は,Regnase-1それ自体をコードするmRNAも標的とする.Regnase-1タンパク質が分解されると,それによりRegnase-1をコードするmRNAが安定化され,このことが時間差をおいたRegnase-1の再発現に寄与しているものと考えられた.
また,Regnase-1はT細胞においても動的な制御をうけていることが明らかになってきた.T細胞受容体の活性化からのシグナルによってもRegnase-1の分解が認められるが,その機構はプロテオソームの阻害剤により抑制されず,マクロファージにおけるToll様受容体への刺激に対する分解の機構とは異なるものと考えられた.T細胞におけるRegnase-1の分解は,BCL10やMALT1/paracaspaseを欠損するT細胞では認められなかったことから,BCL10とMALT1/paracaspaseとの複合体が重要であると思われた.MALT1はカスパーゼのようにアルギニン残基のつぎで切断するタンパク質分解酵素活性をもち,T細胞においてNF-κBの活性化に重要なタンパク質である.しかしながら,MALT1のタンパク質分解酵素活性はNF-κBの活性化には必須ではないと考えられており,その基質に関しては,脱ユビキチン化酵素であるA20のほかにはわかっていなかった.筆者らは,T細胞をMALT1のタンパク質分解酵素活性に対する阻害剤であるzVRPR-fmkにより処理すると,T細胞受容体への刺激に対するRegnase-1の分解が起こらなくなることを見い出した.また,MALT1はRegnase-1を111番目のアルギニン残基のつぎで切断する活性をもつことを見い出した.Regnase-1の111番目のアルギニン残基をアラニン残基に置換した変異体はT細胞受容体への刺激に対しても分解をうけなかったことから,MALT1によるRegnase-1の切断がその分解およびタンパク質の不安定性において重要であることが明らかになった.また,T細胞受容体への刺激に対しc-Rel,ICOS,OX40をコードするmRNAといったRegnase-1の標的mRNAは安定化するが,T細胞をzVRPR-fmkにより処理することによりこれらのmRNAは不安定化したことから,MALT1によるRegnase-1の分解はT細胞におけるエフェクタータンパク質の発現に影響することが示唆された10)(図4b).
このように,免疫細胞を活性化する細胞の外からのシグナルは,これまでのセントラルドグマとして転写を介すのみではなく,mRNAの安定性をも直接に制御することにより,免疫系の制御に重要な役割をはたしていることが明らかになってきた.
サイトカインの発現は翻訳によっても制御をうけている.たとえば,GAIT複合体は,リボソームの構成タンパク質であるL13a,グルタミル-プロリルtRNA合成酵素,NS1関連タンパク質1,グリセルアルデヒド-3-リン酸デヒドロゲナーゼより構成され.インターフェロンγをコードするmRNAの3’側非翻訳領域に存在するGAIT配列に結合し,その翻訳を抑制することが知られている15).
また,I型インターフェロンの産生も翻訳において制御されていることが知られている.翻訳抑制因子である4E-BP1および4E-BP2は転写因子であるIRF7をコードするmRNAの翻訳を抑制しており,これらの翻訳抑制因子を欠損する細胞ではウイルスの感染に対するI型インターフェロンの産生が亢進しており,また,ウイルスの感染に対する感受性が低下していた16).また,インターフェロンにより発現の誘導されるOASL1はIRF7をコードするmRNAの5’側非翻訳領域に結合し,その翻訳を負に制御することも報告されている17).これに対し,翻訳開始因子であるeIF4Eは,209番目のセリン残基がリン酸化されることによりIκBαの翻訳を開始する.eIF4Eのリン酸化変異体を発現するマウスではIκBαの翻訳が低下することによりNF-κBがより活性化し,インターフェロンβの産生の亢進が起こる18).このように,mRNAの安定化だけではなく,翻訳もまた自然免疫により巧妙に制御されている.
ここでは,自然免疫の転写後の制御機構をRNA結合タンパク質の観点から解説した.Regnase-1を欠損するマウスの例からみても,転写後制御は生体における恒常性の維持に重要であることは明らかである.さらに,これまで,炎症応答の制御における細胞内シグナル伝達系は転写因子の活性化によるものが中心であると考えられてきたが,mRNAの安定性や翻訳の制御も,細胞内シグナル伝達系の下流において直接に制御をうけていることが,少しずつではあるが明らかになってきた.しかしながら,この分野はいまだ発展途上であるといわざるをえない.これまで,ヒトではRNA結合領域をもつタンパク質が400以上も報告されているが,その多くは機能が十分には明らかになっていない19).また,タンパク質をコードしないRNAとしては,マイクロRNAのほかにもlincRNA(large intervening non-coding RNA)なども存在し,特異な細胞内局在をとるものが存在することから注目をあつめている.lincRNAにはToll様受容体のうちTLR4への刺激に対し発現の誘導されるものが多く存在し20),なかでも,lincRNA-Cox2は免疫に関連する遺伝子の発現を転写の段階において制御していることが報告されている21).今後の研究により,RNA結合タンパク質を介したmRNA制御の全体像が明らかになり,これまでの転写を中心とした理解から転写後制御を含めた包括的なの理解へと,炎症の分子機構の考え方も転換していくことが期待される.また,Regnase-1はRNA分解酵素活性をもつだけでなく,さらにダイナミックな発現の変化を起こすことから,この活性や発現を制御することにより炎症性疾患の治療法につながることも期待される.
略歴:2001年 大阪大学大学院医学系研究科博士課程 修了,2002年 米国Dana-Farber Cancer Institute研究員,2004年 大阪大学微生物病研究所 助手,2007年 同 准教授を経て,2012年より京都大学ウイルス研究所 教授.
研究テーマ:自然免疫における炎症の制御機構を,とくに転写後制御の観点から明らかにしたい.
研究室URL:http://www.virus.kyoto-u.ac.jp/Lab/Takeuchi_HP/index.html
© 2013 竹内 理 Licensed under CC 表示 2.1 日本
(京都大学ウイルス研究所 感染防御研究分野)
email:竹内 理
領域融合レビュー, 2, e013 (2013) DOI: 10.7875/leading.author.2.e013
Osamu Takeuchi: Innate immunity and RNA binding proteins.
要 約
自然免疫は,病原体やストレスに対する内因性の物質をToll様受容体などの受容体により認識し,初期の炎症応答を起こす機構である.自然免疫によりマクロファージなどから産生される炎症性サイトカインは,炎症の惹起および制御に重要な役割をはたしている.Toll様受容体からのシグナルによるサイトカイン遺伝子をはじめとした炎症に関連する遺伝子の発現は,転写だけではなく,転写後のmRNAの分解および翻訳などによっても緻密に制御され,適切な炎症応答を保っている.mRNAの分解および翻訳の抑制にかかわることの知られるマイクロRNAにくわえ,mRNAの不安定化を促進するTristetraprolinやRNA分解酵素であるRegnase-1といったmRNAの3’側非翻訳領域に結合するRNA結合タンパク質について,炎症の制御における重要性が明らかになってきた.細胞内シグナル伝達系によりRegnase-1の発現は直接に制御されることから,転写だけでなくmRNAの安定性についても,細胞の外からのシグナルにより直接に制御されていると考えられる.ここでは,炎症の転写後制御の分子機構に焦点をあて解説する.
はじめに
免疫系は大きく自然免疫と獲得免疫とに分類することができる.獲得免疫がT細胞やB細胞といった遺伝子の再構成を必要とする受容体により非自己を認識する機構をもち免疫記憶において重要であるのに対し,自然免疫はマクロファージや樹状細胞などにより担われ,ゲノムにコードされた受容体により病原体を認識し,初期における病原体の排除や炎症応答に重要な機構である1).また,自然免疫は樹状細胞を介し獲得免疫の活性化においても重要な役割をはたしている.炎症の惹起および制御には,インターロイキン1,インターロイキン6,TNF(tumor necrosis factor,腫瘍壊死因子)など炎症性サイトカインや,I型インターフェロンのかかわることが知られている.これらのサイトカインは病原体の感染により,活性化したマクロファージや樹状細胞などから産生される.
自然免疫は宿主に存在する受容体が病原微生物の構成成分(PAMP:pathogen-associated molecular pattern,病原微生物関連分子パターン)を検出することにより誘導される.このようなPAMPを認識するパターン認識受容体(pattern recognition receptor:PRR)として,Toll様受容体(Toll-like receptor:TLR),RIG-I様受容体(RIG-I-like receptor:RLR),NOD様受容体(NOD-like receptor:NLR),C型レクチン受容体(C-type lectin receptor:CLR)が知られている1).このうち,Toll様受容体およびC型レクチン受容体はI型膜タンパク質で細胞膜もしくはエンドソーム膜に局在し,微生物,あるいは,内在性の脂質,リポタンパク質,DNA,RNAといったさまざまなPAMPを認識する.これに対し,RIG-I様受容体は膜貫通領域をもたず細胞質内に局在する2本鎖RNA結合タンパク質であり,細胞内におけるRNAウイルスの感染の認識において重要である.NOD様受容体も細胞質に存在する受容体で,ペプチドグリカンやフラジェリンなど一部はToll様受容体と共通したPAMPの認識にかかわる.
インフラマソームを活性化する一部のNOD様受容体を除き,ほとんどのパターン認識受容体は細胞内シグナル伝達系を介して遺伝子発現を活性化する.たとえば,Toll様受容体はTLR3を除き,その細胞質内領域を介してアダプタータンパク質であるMyD88と結合し,さらに,IRAKを介して細胞内にシグナルを伝達し,TRAF6などのポリユビキチンシグナルタンパク質をへてIκBキナーゼ(IKK)を活性化する.IκBキナーゼはIKKα,IKKβ,NEMO/IKKγより構成され,NF-κB阻害タンパク質であるIκBをリン酸化し,ユビキチン-プロテオソーム系によるIκBの分解を誘導する.その結果,IκBから遊離したNF-κBは核へと移行し,炎症に関連する遺伝子のプロモーター領域に結合してその転写を活性化する.RIG-I様受容体はアダプタータンパク質としてIPS-1を介して細胞内シグナル伝達系を活性化するが,その下流ではIκBキナーゼを介してNF-κBの核への移行を促進するなど,共通のエフェクタータンパク質を活性化させている.また,NOD様受容体の一部やC型レクチン受容体からのシグナルもNF-κBを活性化させることが知られており,パターン認識受容体からのシグナルはその多くが下流に共通のモジュールをもつと考えられる.Toll様受容体の一部およびRIG-I様受容体からのシグナルは転写因子であるIRF3やIRF7(IRF:interferon-regulatory factor)を介してI型インターフェロンを産生させる.
このように,自然免疫においてパターン認識受容体から転写の活性化にいたる細胞内シグナル伝達系や,エピジェネティックな転写の制御機構に関しては,これまで詳細な解析がなされてきた.しかし,サイトカインの産生の制御は転写だけでは説明がつかず,マイクロRNAやさまざまなRNA結合タンパク質による転写後制御の重要性が明らかになってきている.ここでは,マイクロRNAに関する解説はほかにゆずり2),RNA結合タンパク質と遺伝子の発現制御との関連に焦点をあてる.
1.AUリッチ配列結合タンパク質による制御機構
サイトカインや増殖因子をコードするmRNAには,その3’側非翻訳領域にAUを多く含むAUリッチ配列(典型的には,AUUUA)が高頻度に存在することが知られている.また,この配列の存在の頻度とmRNAの不安定性とが相関することも報告されている.たとえば,TNFの刺激に対し早期に誘導される遺伝子のmRNAの3’側非翻訳領域は多くのAUリッチ配列をもつことが報告されており,これらのmRNAはより速く分解されると考えられている3).これまで,AUリッチ配列に結合するタンパク質としては,Tristetraprolin(TTP)やそのファミリータンパク質であるZfp36L1,Zfp36L2,また,AUF1,KSRP,HuRなど,多くが知られている4).
TristetraprolinはToll様受容体への刺激により早期に誘導されるタンパク質であり,2つのCCCH型ジンクフィンガー領域をもつ.この領域はRNA結合ドメインとして知られ,mRNAの3’側非翻訳領域に存在するAUリッチ配列との結合において重要である.TristetraprolinはTNFをコードするmRNAをAUリッチ配列を介し不安定化する(図1).これまでに,Tristetraprolinはインターフェロンγやインターロイキン10をはじめさまざまなサイトカインのmRNAの安定性の制御にかかわることが報告されている.加齢したTristetraprolinの欠損マウスはTNFの過剰な産生により関節炎を発症する5).TristetraprolinはTNFをコードするmRNAの3’側非翻訳領域に結合し,脱アデニル化酵素を含むCCR4-NOT複合体をリクルートすることにより,mRNAからポリA鎖が除去される.ポリA鎖の除去されたmRNAはつづいてキャップ構造が除去され,XRN1やエクソソームなどのRNA分解酵素により分解される.AUF1はTNF,インターロイキン1β,インターロイキン6などのサイトカインのmRNAの3’側非翻訳領域にAUリッチ配列を介して結合し,これらのmRNAを不安定化することが知られている4).AUF1を欠損するマウスはリポ多糖によるショックに対し野生型マウスよりも感受性であり,TNFやインターロイキン1βといった炎症性サイトカインを過剰に産生することが報告されている.KSRPはRNAスプライシングやマイクロRNAの産生などにかかわるタンパク質であるが,AUリッチ配列に結合しサイトカインをコードするmRNAを不安定化するほか,その翻訳を抑制することが報告されている6).これに対し,同様なAUリッチ配列結合タンパク質であるHuRは,AUリッチ配列をもつmRNAを細胞質において安定化することが知られている7).AUリッチ配列結合タンパク質は自然免疫だけでなく獲得免疫の制御にもかかわることが報告されている.その例として,Zfp36l1とZfp36l2は胸腺におけるT細胞の分化に必要であり,また,急性T細胞白血病の発症を抑制している.また,Zfp36L2は赤芽球系前駆細胞の自己複製にかかわることが知られている8).
このように,AUリッチ配列結合タンパク質は炎症性サイトカインの産生を正に負に制御し,炎症のバランスをとっていることが明らかになってきた.
2.Regnase-1の免疫応答における役割
mRNAの安定性はAUリッチ配列のみで制御されているわけではない.Zc3h12a遺伝子はマクロファージにおいてToll様受容体からのシグナルにより発現が誘導され,この遺伝子のコードするタンパク質はRNase領域およびCCCH型ジンクフィンガー領域をもつ.また,このタンパク質はインターロイキン6をコードするmRNAと結合し,細胞に強制発現するとそのRNA分解酵素活性に依存してこのmRNAを分解した.また,のちに述べるように,このタンパク質は免疫系の制御においても重要であったことから,筆者らは,Regnase-1(regulatory RNase-1)と命名した(図2a).Regnase-1はインターロイキン6をコードするmRNAの3’側非翻訳領域に存在するステムループ領域を介してこのmRNAを不安定化した9)(図2b).また,Regnase-1を欠損したマクロファージにおいては,Toll様受容体への刺激に応答したインターロイキン6やインターロイキン12 p40などの産生が亢進していた.Regnase-1ノックアウトマウスはメンデルの法則にしたがって生まれるが成長の遅延を示し,生後3週目ほどで死亡しはじめ,12週までにはほぼ全例が死亡した.Regnase-1ノックアウトマウスは血清においてさまざまなクラスの免疫グロブリンが増加しており,また,抗核抗体や抗2本鎖DNA抗体など自己抗体を産生していた.Regnase-1の欠損のもとでは形質細胞の増加,また,エフェクターT細胞の増加が観察された.Regnase-1ノックアウトマウスの組織を観察すると,肺などの組織へのリンパ球,とくに,形質細胞の浸潤が著明であり,肺において免疫グロブリンG産生細胞や免疫グロブリンA産生細胞の著明な増加が認められた.
Regnase-1の欠損により炎症が惹起される分子機構を検討するため,Cre-loxP系を用いて細胞種に特異的なRegnase-1ノックアウトマウスを作製した.T細胞に特異的にRegnase-1を欠損させたところ,全身におけるRegnase-1ノックアウトマウスと同様の炎症性疾患を発症し,8週をこえてから自然に死亡しはじめた.また,このノックアウトマウスにもさまざまな臓器への炎症細胞の浸潤や著明な脾腫が認められ,エフェクターT細胞および形質細胞が増加していた.また,T細胞受容体への刺激によるCD4陽性T細胞からのサイトカインの産生も亢進しており,T細胞に発現するRegnase-1がT細胞の活性化を抑制する役割をはたしていることが明らかになった.CD4陽性T細胞におけるRegnase-1の標的mRNAについて検討したところ,サイトカインであるインターロイキン2や,細胞表面タンパク質であるICOSおよびOX40,転写因子のなかでもc-RelをコードするmRNAが分解されていることが明らかになり,Regnase-1は細胞種ごとに特徴的なmRNAを標的として免疫細胞の活性化を制御していると考えられた10).
以上の結果から,Regnase-1は免疫の活性化にかかわるタンパク質をコードするmRNAをRNA分解酵素として直接に分解すること,また,自然免疫および獲得免疫にはたらく細胞において,自己免疫性の炎症性疾患の発症を抑制するため重要なタンパク質であることが明らかになった.最近になり,Regnase-1と逆に,インターロイキン6をコードするmRNAを3’側非翻訳領域を介して安定化するタンパク質Arid5aの存在も報告されている11).
3.RoquinによるT細胞および自然免疫の制御
Roquinは,エチルニトロソ尿素の投与による変異により自己免疫疾患を発症するようになったマウス系統において,その原因遺伝子にコードされるタンパク質として見い出された12).この変異マウスは,自己抗体の産生,脾臓における胚中心や濾胞性ヘルパーT細胞の増加,T細胞におけるICOSの発現の増加を呈した.RoquinはRc3h1遺伝子にコードされ,ユビキチンリガーゼ活性をもつと考えられるRINGフィンガー領域にくわえ,RNAと結合するCCCH型ジンクフィンガー領域,および,ROQドメインをもつ.RoquinはICOSなどをコードするmRNAに直接もしくはマイクロRNAを介し間接的に結合し,その不安定化を誘導すると考えられている.Roquinは,標的RNAに脱アデニル化酵素を含むCCR4-NOT複合体をリクルートすることにより,その分解を促進する(図3).また,RoquinはTNFをコードするmRNAの3’側非翻訳領域に存在するステムループ構造に結合するとの報告もあり,Roquinに変異をもつマウスではリポ多糖の投与に対するTNFの産生が亢進している13).したがって,Regnase-1と同様に,Roquinは獲得免疫および自然免疫をともに制御するRNA結合タンパク質であると考えられる.
4.免疫細胞におけるシグナル伝達によるmRNAの安定性の制御
さきに述べたように,転写は細胞の外からの刺激により細胞内シグナル伝達系を介して緻密に制御されていることがよく知られている.くわえて,いくつかの研究により,mRNAの安定性それ自体も細胞の外からの刺激により制御をうけていることが明らかになりつつある.
たとえば,自然免疫により活性化されるMAPキナーゼファミリーのひとつであるp38 MAPキナーゼは,AUリッチ配列をもつRNAを安定化することが知られている.この分子機構のひとつとして,p38 MAPキナーゼがMK2を活性化し,このMK2がTristetraprolinをリン酸化することがあげられる.リン酸化されたTristetraprolinは14-3-3とよばれるタンパク質と結合することにより,TNFなどをコードするmRNAを不安定化する活性が低下すると考えられている(図4a).
Regnase-1もタンパク質の修飾を介して免疫の活性化によるダイナミックな制御をうけている.Regnase-1は定常状態においても免疫細胞に発現しており,これは,マクロファージにおいてインターロイキン6をコードするmRNAの発現を完全に抑制するために重要である.このことは,Regnase-1を欠損した細胞においては定常状態でも野生型の細胞と比較してインターロイキン6のmRNAが高く発現していることからも明らかである.Toll様受容体への刺激あるいはインターロイキン1βによるMyD88を介した刺激に対して,Regnase-1がIκBキナーゼ複合体により直接にリン酸化され,ユビキチン-プロテオソーム系により早期に分解されることが明らかになった14).Regnase-1にはIκBキナーゼによりリン酸化をうけるAsp-Ser-Gly-X-X-Serというアミノ酸配列が保存されており(図2a),IκBキナーゼを欠損する細胞ではToll様受容体への刺激に対するRegnase-1の分解はみられなくなるほか,リン酸化をうける2つのセリン残基に変異を導入した場合にはRegnase-1の分解は起こらなくなり,また,インターロイキン6をコードするmRNAの産生をより強く抑制した.このように,Toll様受容体-IκBキナーゼ経路は,NF-κBを介した転写の活性化(アクセル)のみならず,Regnase-1を分解することで転写に対するブレーキを解除することにより,インターロイキン6などの炎症性サイトカインの迅速な産生を実現していると考えられる(図4a).また,Regnase-1はマクロファージにおいて分解されたのち,数時間で再発現してくる.この再発現にはRegnase-1の転写の活性化がかかわっているが,それにくわえ,Regnase-1をコードするmRNAの安定性も増加していることが明らかになった.Regnase-1は,Regnase-1それ自体をコードするmRNAも標的とする.Regnase-1タンパク質が分解されると,それによりRegnase-1をコードするmRNAが安定化され,このことが時間差をおいたRegnase-1の再発現に寄与しているものと考えられた.
また,Regnase-1はT細胞においても動的な制御をうけていることが明らかになってきた.T細胞受容体の活性化からのシグナルによってもRegnase-1の分解が認められるが,その機構はプロテオソームの阻害剤により抑制されず,マクロファージにおけるToll様受容体への刺激に対する分解の機構とは異なるものと考えられた.T細胞におけるRegnase-1の分解は,BCL10やMALT1/paracaspaseを欠損するT細胞では認められなかったことから,BCL10とMALT1/paracaspaseとの複合体が重要であると思われた.MALT1はカスパーゼのようにアルギニン残基のつぎで切断するタンパク質分解酵素活性をもち,T細胞においてNF-κBの活性化に重要なタンパク質である.しかしながら,MALT1のタンパク質分解酵素活性はNF-κBの活性化には必須ではないと考えられており,その基質に関しては,脱ユビキチン化酵素であるA20のほかにはわかっていなかった.筆者らは,T細胞をMALT1のタンパク質分解酵素活性に対する阻害剤であるzVRPR-fmkにより処理すると,T細胞受容体への刺激に対するRegnase-1の分解が起こらなくなることを見い出した.また,MALT1はRegnase-1を111番目のアルギニン残基のつぎで切断する活性をもつことを見い出した.Regnase-1の111番目のアルギニン残基をアラニン残基に置換した変異体はT細胞受容体への刺激に対しても分解をうけなかったことから,MALT1によるRegnase-1の切断がその分解およびタンパク質の不安定性において重要であることが明らかになった.また,T細胞受容体への刺激に対しc-Rel,ICOS,OX40をコードするmRNAといったRegnase-1の標的mRNAは安定化するが,T細胞をzVRPR-fmkにより処理することによりこれらのmRNAは不安定化したことから,MALT1によるRegnase-1の分解はT細胞におけるエフェクタータンパク質の発現に影響することが示唆された10)(図4b).
このように,免疫細胞を活性化する細胞の外からのシグナルは,これまでのセントラルドグマとして転写を介すのみではなく,mRNAの安定性をも直接に制御することにより,免疫系の制御に重要な役割をはたしていることが明らかになってきた.
5.サイトカインの産生の翻訳による制御
サイトカインの発現は翻訳によっても制御をうけている.たとえば,GAIT複合体は,リボソームの構成タンパク質であるL13a,グルタミル-プロリルtRNA合成酵素,NS1関連タンパク質1,グリセルアルデヒド-3-リン酸デヒドロゲナーゼより構成され.インターフェロンγをコードするmRNAの3’側非翻訳領域に存在するGAIT配列に結合し,その翻訳を抑制することが知られている15).
また,I型インターフェロンの産生も翻訳において制御されていることが知られている.翻訳抑制因子である4E-BP1および4E-BP2は転写因子であるIRF7をコードするmRNAの翻訳を抑制しており,これらの翻訳抑制因子を欠損する細胞ではウイルスの感染に対するI型インターフェロンの産生が亢進しており,また,ウイルスの感染に対する感受性が低下していた16).また,インターフェロンにより発現の誘導されるOASL1はIRF7をコードするmRNAの5’側非翻訳領域に結合し,その翻訳を負に制御することも報告されている17).これに対し,翻訳開始因子であるeIF4Eは,209番目のセリン残基がリン酸化されることによりIκBαの翻訳を開始する.eIF4Eのリン酸化変異体を発現するマウスではIκBαの翻訳が低下することによりNF-κBがより活性化し,インターフェロンβの産生の亢進が起こる18).このように,mRNAの安定化だけではなく,翻訳もまた自然免疫により巧妙に制御されている.
おわりに
ここでは,自然免疫の転写後の制御機構をRNA結合タンパク質の観点から解説した.Regnase-1を欠損するマウスの例からみても,転写後制御は生体における恒常性の維持に重要であることは明らかである.さらに,これまで,炎症応答の制御における細胞内シグナル伝達系は転写因子の活性化によるものが中心であると考えられてきたが,mRNAの安定性や翻訳の制御も,細胞内シグナル伝達系の下流において直接に制御をうけていることが,少しずつではあるが明らかになってきた.しかしながら,この分野はいまだ発展途上であるといわざるをえない.これまで,ヒトではRNA結合領域をもつタンパク質が400以上も報告されているが,その多くは機能が十分には明らかになっていない19).また,タンパク質をコードしないRNAとしては,マイクロRNAのほかにもlincRNA(large intervening non-coding RNA)なども存在し,特異な細胞内局在をとるものが存在することから注目をあつめている.lincRNAにはToll様受容体のうちTLR4への刺激に対し発現の誘導されるものが多く存在し20),なかでも,lincRNA-Cox2は免疫に関連する遺伝子の発現を転写の段階において制御していることが報告されている21).今後の研究により,RNA結合タンパク質を介したmRNA制御の全体像が明らかになり,これまでの転写を中心とした理解から転写後制御を含めた包括的なの理解へと,炎症の分子機構の考え方も転換していくことが期待される.また,Regnase-1はRNA分解酵素活性をもつだけでなく,さらにダイナミックな発現の変化を起こすことから,この活性や発現を制御することにより炎症性疾患の治療法につながることも期待される.
文 献
- Takeuchi, O. & Akira, S.: Pattern recognition receptors and inflammation. Cell, 140, 805-820 (2010)[PubMed]
- O'Neill, L. A., Sheedy, F. J. & McCoy, C. E.: MicroRNAs: the fine-tuners of Toll-like receptor signalling. Nat. Rev. Immunol., 11, 163-175 (2011)[PubMed]
- Hao, S. & Baltimore, D.: The stability of mRNA influences the temporal order of the induction of genes encoding inflammatory molecules. Nat. Immunol., 10, 281-288 (2009)[PubMed]
- Anderson, P.: Post-transcriptional regulons coordinate the initiation and resolution of inflammation. Nat. Rev. Immunol., 10, 24-35 (2010)[PubMed]
- Carballo, E., Lai, W. S. & Blackshear, P. J.: Feedback inhibition of macrophage tumor necrosis factor-alpha production by tristetraprolin. Science, 281, 1001-1005 (1998)[PubMed]
- Ruggiero, T., Trabucchi, M., De Santa, F. et al.: LPS induces KH-type splicing regulatory protein-dependent processing of microRNA-155 precursors in macrophages. FASEB J., 23, 2898-2908 (2009)[PubMed]
- Brennan, C. M. & Steitz, J. A.: HuR and mRNA stability. Cell. Mol. Life Sci., 58, 266-277 (2001)[PubMed]
- Zhang, L., Prak, L., Rayon-Estrada, V. et al.: ZFP36L2 is required for self-renewal of early burst-forming unit erythroid progenitors. Nature, 499, 92-96 (2013)[PubMed]
- Matsushita, K., Takeuchi, O., Standley, D. M. et al.: Zc3h12a is an RNase essential for controlling immune responses by regulating mRNA decay. Nature, 458, 1185-1190 (2009)[PubMed]
- Uehata, T., Iwasaki, H., Vandenbon, A. et al.: Malt1-induced cleavage of regnase-1 in CD4+ helper T cells regulates immune activation. Cell, 153, 1036-1049 (2013)[PubMed] [新着論文レビュー]
- Masuda, K., Ripley, B., Nishimura, R. et al.: Arid5a controls IL-6 mRNA stability, which contributes to elevation of IL-6 level in vivo. Proc. Natl. Acad. Sci. USA, 110, 9409-9414 (2013)[PubMed]
- Vinuesa, C. G., Cook, M. C., Angelucci, C. et al.: A RING-type ubiquitin ligase family member required to repress follicular helper T cells and autoimmunity. Nature, 435, 452-458 (2005)[PubMed]
- Leppek, K., Schott, J., Reitter, S. et al.: Roquin promotes constitutive mRNA decay via a conserved class of stem-loop recognition motifs. Cell, 153, 869-881 (2013)[PubMed]
- Iwasaki, H., Takeuchi, O., Teraguchi, S. et al.: The IκB kinase complex regulates the stability of cytokine-encoding mRNA induced by TLR-IL-1R by controlling degradation of regnase-1. Nat. Immunol., 12, 1167-1175 (2011)[PubMed] [新着論文レビュー]
- Mukhopadhyay, R., Jia, J., Arif, A. et al.: The GAIT system: a gatekeeper of inflammatory gene expression. Trends Biochem. Sci., 34, 324-331 (2009)[PubMed]
- Colina, R., Costa-Mattioli, M., Dowling, R. J. et al.: Translational control of the innate immune response through IRF-7. Nature, 452, 323-328 (2008)[PubMed]
- Lee, M. S., Kim, B., Oh, G. T. et al.: OASL1 inhibits translation of the type I interferon-regulating transcription factor IRF7. Nat. Immunol., 14, 346-355 (2013)[PubMed]
- Yanagiya, A., Suyama, E., Adachi, H. et al.: Translational homeostasis via the mRNA cap-binding protein, eIF4E. Mol. Cell, 46, 847-858 (2012)[PubMed] [新着論文レビュー]
- Ray, D., Kazan, H., Cook, K. B. et al.: A compendium of RNA-binding motifs for decoding gene regulation. Nature, 499, 172-177 (2013)[PubMed]
- Guttman, M., Amit, I., Garber, M. et al.: Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature, 458, 223-227 (2009)[PubMed]
- Carpenter, S., Aiello, D., Atianand, M. K. et al.: A long noncoding RNA mediates both activation and repression of immune response genes. Science, 341, 789-792 (2013)[PubMed]
著者プロフィール
略歴:2001年 大阪大学大学院医学系研究科博士課程 修了,2002年 米国Dana-Farber Cancer Institute研究員,2004年 大阪大学微生物病研究所 助手,2007年 同 准教授を経て,2012年より京都大学ウイルス研究所 教授.
研究テーマ:自然免疫における炎症の制御機構を,とくに転写後制御の観点から明らかにしたい.
研究室URL:http://www.virus.kyoto-u.ac.jp/Lab/Takeuchi_HP/index.html
© 2013 竹内 理 Licensed under CC 表示 2.1 日本