シグナル伝達系における制御の規則性と細胞応答の予測
2014/01/06
岡田 眞里子
(理化学研究所統合生命医科学研究センター 統合細胞システム研究チーム)
email:岡田眞里子
領域融合レビュー, 3, e001 (2014) DOI: 10.7875/leading.author.3.e001
Mariko Okada: Prediction of cellular responses based on regulatory rules of signal transduction pathways.

細胞内のシグナル伝達系は,1980年代より,がんをはじめとした疾病との関連性により隆盛をきわめた分野である.近年では,2003年のヒトゲノム解読完了と前後して注目をあびはじめたシステム生物学において,実験と理論との融合がもっとも進んでいる分野でもある.このレビューでは,細胞内のシグナル伝達系のシステム生物学的な研究に対し期待されている社会的および学術的な背景をはじめ,シグナル伝達系のダイナミクスとその背後にある制御のデザイン,頻繁にみられる制御の規則性,細胞の制御への応用などにふれる.対象としては,システム生物学の立場からみてさまざまな研究の進んでいるERK経路およびNF-κB経路について中心に紹介する.最後に,今後,このようなシステム生物学が何をめざして進んでいくのか,その将来を展望する.
細胞内シグナル伝達系は,環境からの情報を転写因子に伝達し遺伝子の発現を制御することにより細胞の運命決定を担う分子機構で,タンパク質のリン酸化,相互作用,分解など生化学的な反応の集合体としてなりたつ.ヒトの全遺伝子の約20%がシグナル伝達系にかかわるともいわれ,シグナル伝達系はそれ自体が大きなネットワークである.しかし,このネットワークの入出力の関係はさまざまな制御のために非線形性であり,おのおののシグナル活性を足し算することにより細胞の応答を予測することはほとんどできない.このことはシグナル伝達系の異常と関連の深い,がん,糖尿病,炎症など,さまざまな疾病の解明および治療において大きな問題である.このような背景から,1990年代の後半から,シグナル伝達系の分子ダイナミクスと細胞応答とに規則性を見い出そうとする“システム生物学”がさかんになった.このレビューでは,約15年間にわたるシグナル伝達系のシステム生物学における研究の動向とその将来について紹介する.
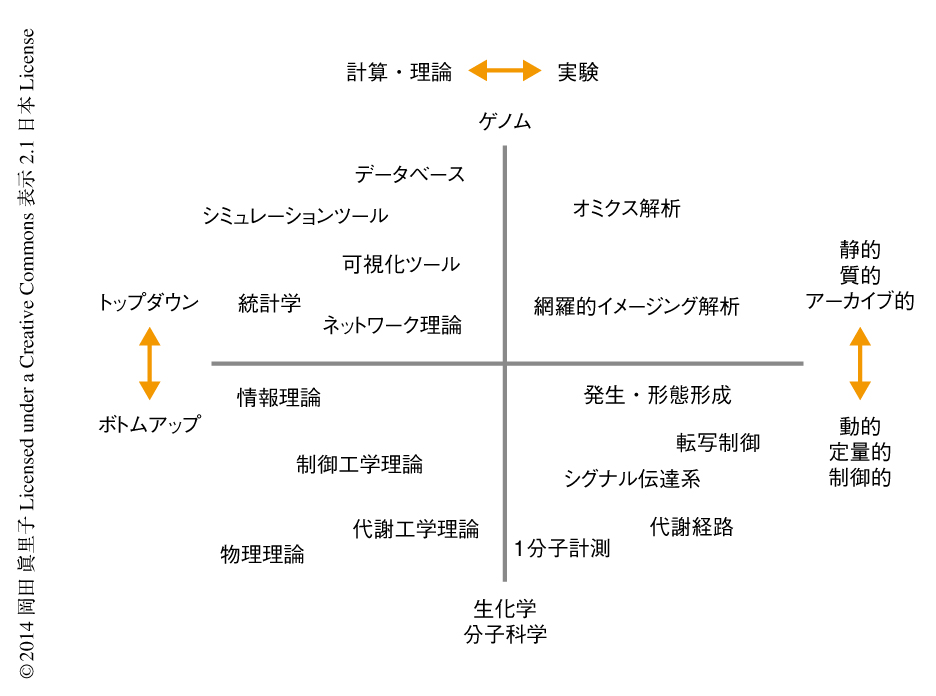
1990年代の後半から2000年代の初頭にかけて,主要なシグナル伝達系であるERK経路およびNF-κB経路のデジタルな応答および振動とその機序に関する研究が発表された1,2).これらの論文により,シグナル伝達系はそれまで考えられていた以上にダイナミクスに富んだ系であり,そこにはいまだ明らかにされていない分子制御のかくされていることが示された.さらにこのころ,ヒトゲノムが解読された.それを機に,ゲノム情報をもとにしたハイスループットな解析技術やそれをサポートするツールが充実し,これらがオミクス解析を中心としたトップダウン型の研究,および,ターゲットをしぼってモデルを構築して解析するボトムアップ型の研究の2つのタイプのシステム生物学の研究を加速した.トップダウン型の研究においては,生物における大規模かつ静的な分子間相互作用のネットワークが社会的なネットワークと類似した構造をもつことが明らかにされ,そこに頻出するネットワークモチーフを同定することにより生物のネットワークの普遍性が指摘された3,4).また,ボトムアップ型の研究においては,制御工学,代謝工学,生化学の影響をうけた動的なモデルが構築され,シグナルの動態を決定する分子制御が予測された5,6).このようにして,さまざまな実験データを理論的に整理するため,あるいは,生物をシステムとして理解するため,システム生物学が広まっていった(図1).
細胞内シグナル伝達系のシステム生物学的な研究の目的のひとつは,複雑なシグナルのネットワークから細胞の応答を“予測”することである.この背景には,1990年代後半から市販されるようになった分子標的薬,たとえば,EGF(ErbB1)受容体の標的薬剤であるゲフィチニブ(肺がんを対象とする),Bcr-Ablの標的薬剤であるイマチニブ(慢性骨髄性白血病を対象とする),ErbB2の標的薬剤であるトラスツズマブ(乳がんを対象とする),などの登場がある7).ゲフィチニブおよびトラスツズマブは膜受容体であるErbBファミリーを標的とするが,ErbBファミリーは活性化にともないホモ二量体あるいはヘテロ二量体を形成することから,たとえば,ErbB1を第1の標的としても細胞において共発現するほかのErbBファミリーを間接的に阻害してしまう.しかし,どれだけ実験の精度をあげても,細胞においてErbB1とほかのErbBファミリーに対する薬剤の効果の違いを定量的に切り分けることは困難である.また,ゲフィチニブあるいはトラスツズマブの投与によりErbBファミリーを長期間にわたり阻害すると,ほかの受容体からの経路を経由してErbBファミリーの下流の経路が活性化するといった薬剤耐性の問題も生じていた8).こういったことから,薬剤の主効果および副効果の影響を定量的に評価し,そこから生じるかもしれない副作用を薬剤開発の早い段階から予測することが期待されるようになった.これらの薬剤を開発した製薬企業では早くから数理モデルを用いたシグナル伝達系の理論的な研究を行っており,これらと連携した研究グループ,また,そこから派生したベンチャー企業では,シグナル伝達系を対象とした大規模なモデルの構築や9),それを利用した制御分子や薬剤効果の予測,また,オミクスデータから統計的な手法を用いてネットワーク予測を行い10),細胞運命の決定因子を同定している.
細胞内シグナル伝達系のシステム生物学的な研究のもうひとつの目的は,シグナル伝達系の特異性およびシグナル伝達の分子機構の解明である.というのも,膜受容体とリガンドとの特異的な結合を除くと,シグナル伝達系にかかわるタンパク質の反応特異性は比較的低く,シグナル伝達系の特異性および認識をつかさどる実体はまだよくわかっていないからである.たとえば,酵素においては鍵と鍵穴のような酵素と基質との立体構造の認識に関する理論があり,酵素の特異性を表わすパラメーターなどから生成物の量をある程度は予測することができる.しかし,シグナル伝達のネットワークの特異性は,そこに含まれる酵素,たとえば,プロテインキナーゼの特異性として十分には説明できない.なぜなら,プロテインキナーゼは基質のひとつであるATPに対する結合特異性は高いが,タンパク質の基質に対する特異性は一般的に低いからである11).このことは,SH2ドメインとチロシンリン酸化タンパク質とのあいだのような分子間相互作用についても同じである12).このような分子認識の特異性の低さはシグナル伝達のネットワークに冗長性(redundancy)および頑強性(robustness)をあたえ,遺伝子に変異や欠損などの異常が起こってもネットワーク全体の機能を維持できるという点で生物にとり利点である.しかし一方で,シグナル伝達の存在の意味とは,このような冗長性のなかから特定の情報を選り分けて核において特定の転写因子を活性化することにあるはずである.その機構を明らかにすることができれば,そのことを細胞応答の予測に利用し,さらには,副作用のない細胞の制御が可能になるはずである.
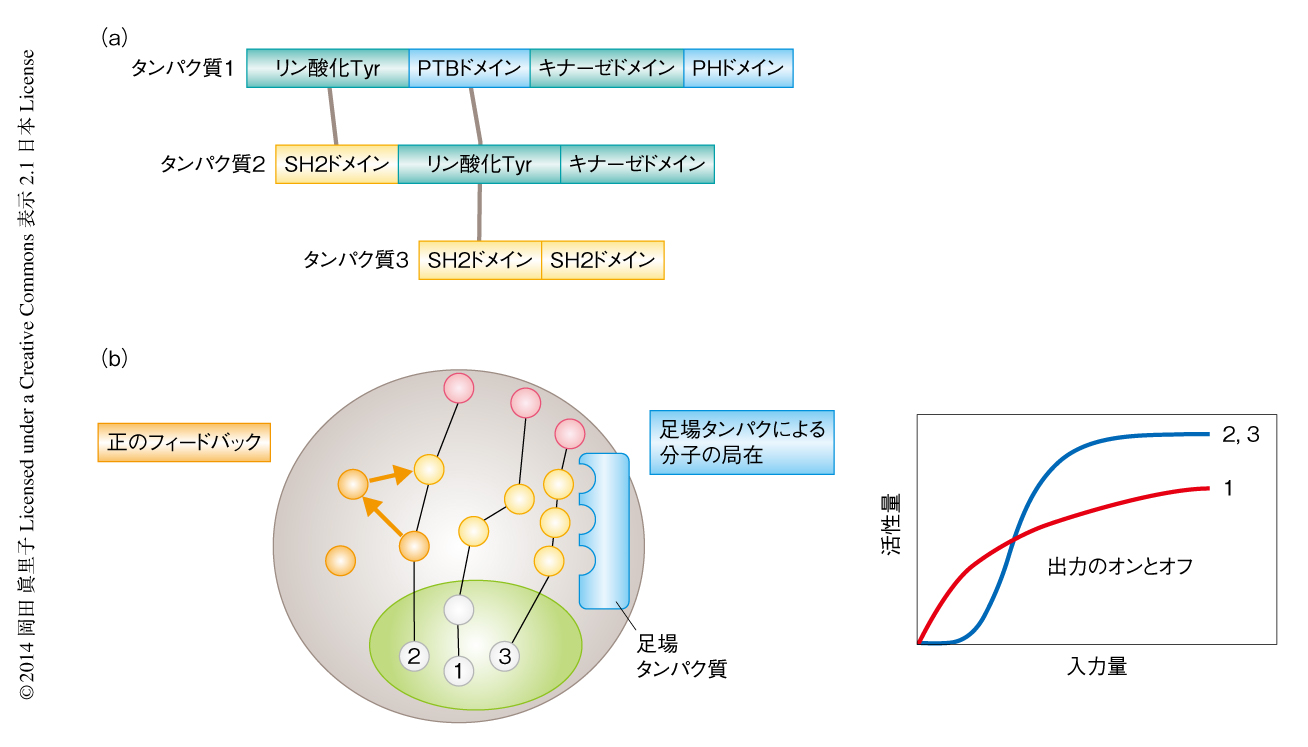
この冗長性および特異性という特徴をあわせもつシグナル伝達系の基盤のひとつは,シグナルタンパク質のマルチドメイン性13)(モジュール性),および,タンパク質のもつ活性部位を直接に制御するのではなく,そこから離れた制御部位を修飾し,タンパク質の立体構造を変化させることにより活性を遠隔制御するというアロステリック制御11) である.マルチドメイン性はひとつのシグナルタンパク質に複数のタンパク質との相互作用を可能にし,たとえば,アダプタータンパク質であるGab2,Grb2,ShcはSH2ドメインあるいはPTBドメインを介して互いに膜受容体と結合し,どのアダプタータンパク質を経由してもERK経路を活性化できるような冗長なネットワークを形成する(図2a).また同様に,これらのシグナルタンパク質にはリン酸化などアロステリックな制御がくわわり,正のフィードバックあるいは負のフィードバックの作用点としてはたらいたり,シグナルタンパク質の局在性を変化させたりする.このなかで,正のアロステリック制御はシグナルタンパク質のあいだの協同性を高め,非線形な出力,すなわち,特異的な活性をひき出す(図2b).
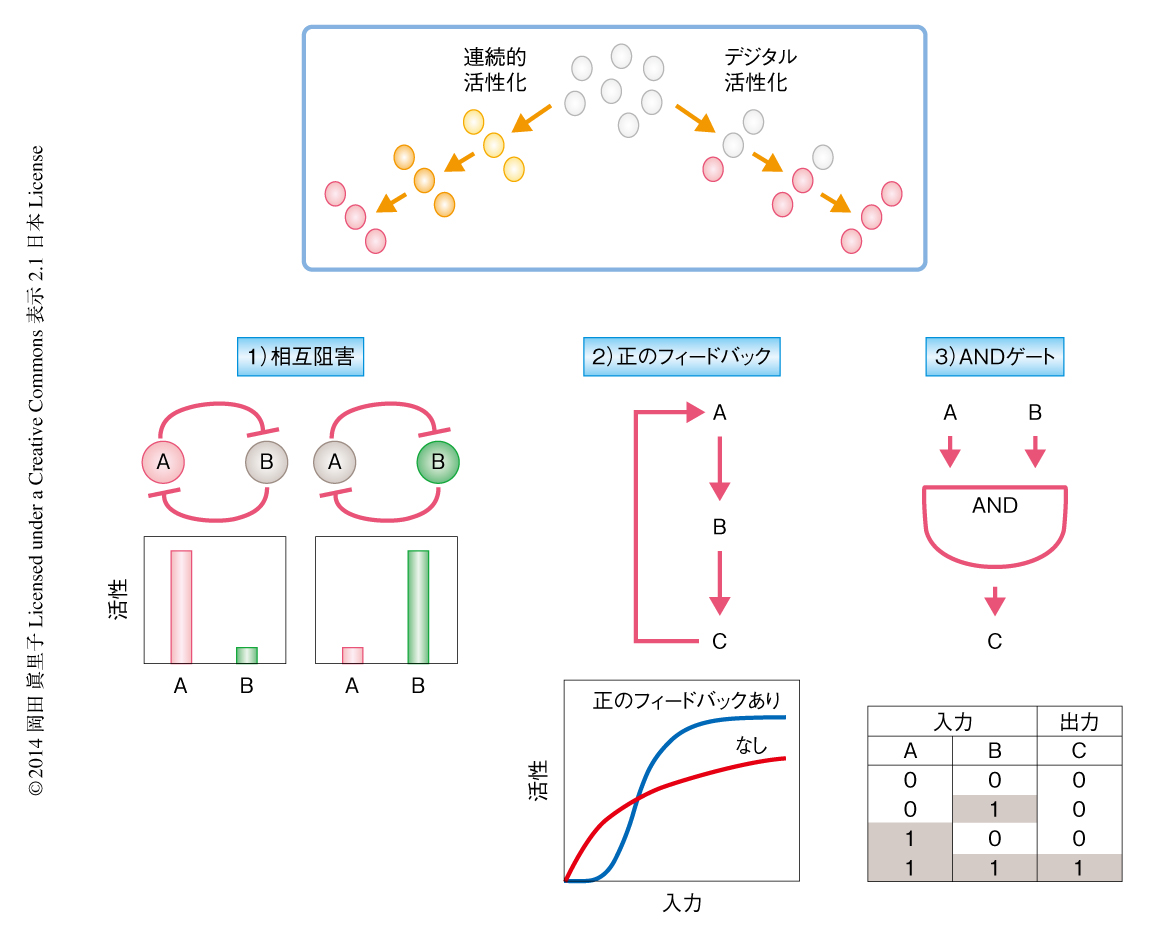
シグナル伝達系の役目のひとつはアナログなシグナルのデジタルへの変換である.これは,少しずつ異なる情報量を1か0の情報量に変える機構である.たとえば,リガンドと受容体との反応強度は,シグナル全体の強度と転写による出力に大きく影響する.これらの入力の違いは,成長因子や抗体など異なるリガンドということもあるし,同じリガンドであっても濃度が異なる場合もある.このような違いには,リガンドの親和性あるいは受容体の発現量が大きく影響する.しかし,この親和性あるいは発現量の違いとは,あくまでも数値の大小の違い(アナログ性の違い)である.ところが,時間にして数分から数十分のシグナル伝達系の反応をへると,このアナログなシグナル量の違いが,0か1のデジタルな転写活性の違いとして現われる場合がある.このような転写による出力のデジタルへの変換は細胞内のシグナル伝達系のもつ機能のひとつであり,細胞を質的に変えることのできる可能性をもつ.これまでの理論的な研究により,正のフィードバック制御1,14),相互阻害15),のちに述べるANDゲート16),のようなネットワークモチーフが細胞のデジタルな活性化を生じることが示されている(図3).さらに,このような明らかなネットワークモチーフをもたない場合でも,たとえば,ERKの分配的な二重リン酸化反応の場合には,2つのリン酸化を脱リン酸化する酵素の量が少ないような特別なパタメーターの条件において系は実質的な相互阻害としてはたらき,デジタルな出力が生じるようになる17).ネットワークと動態との関係については,つぎに述べるERK経路がもっともよく研究されている.
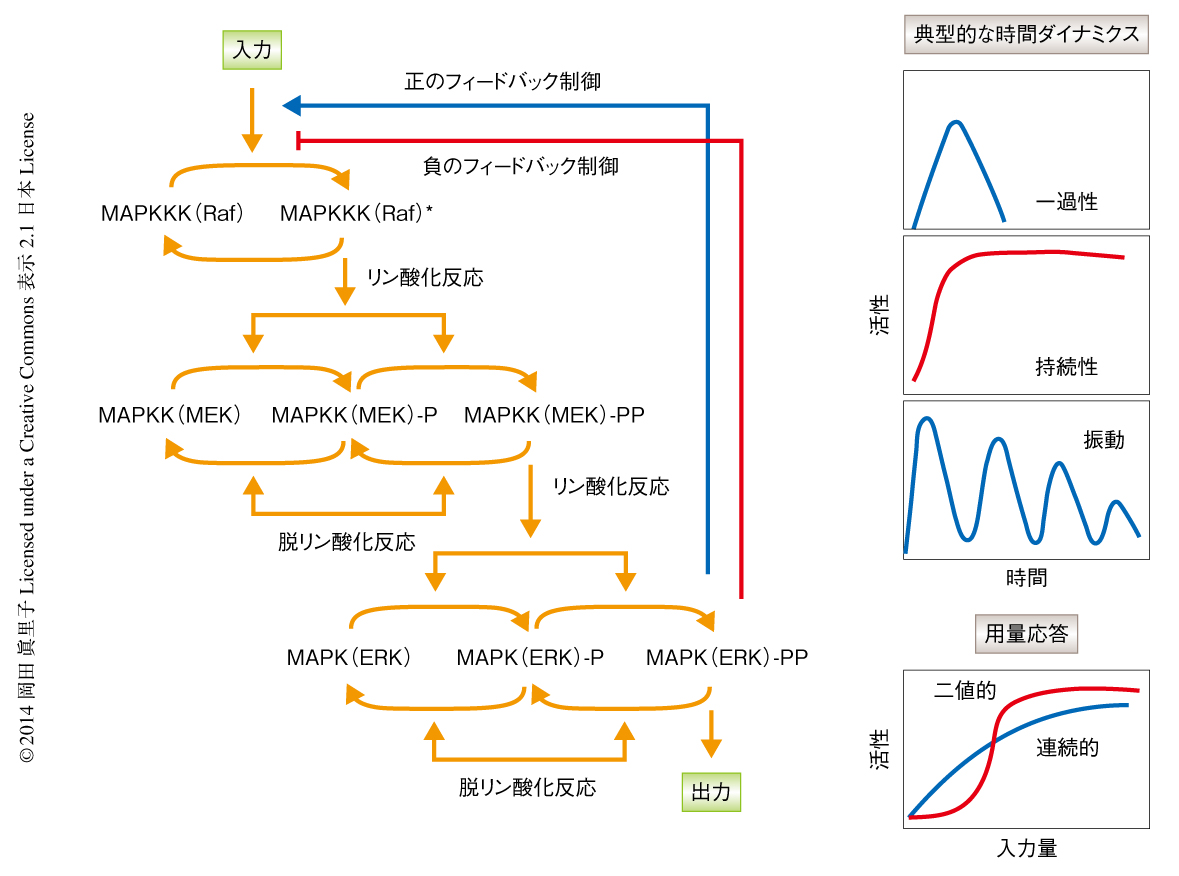
ERKは,EGF受容体,PDGF受容体,インスリン受容体,IGF受容体,FGF受容体,Trk受容体など,さまざまな膜受容体からのシグナルを集約するセリン/スレオニンキナーゼであり,細胞の増殖,分化,成長など,さまざまな細胞の制御にかかわる18).ERKはRas-Raf-MEK-ERK経路をへて活性され,活性化されると細胞質から核へと移行し,ELKあるいはCREBなどの転写因子を活性化し遺伝子の発現を誘導する.このERK経路では,さきに述べた,正のフィードバック制御14),相互阻害17),ANDゲート16) の,デジタル応答を生じるすべてのネットワークモチーフが報告されている.さらに,その活性の動態としても,一過性の活性あるいは持続性の活性のほか,活性の振動も報告されている19,20)(図4).このようなさまざまな反応の特性は理論から想定されるRaf-MEK-ERK経路における二重リン酸化の反応様式(distributiveあるいはprocessive)の違いにより大きな影響をうける.ERK経路におけるデジタルな出力としては,ERKそのもののデジタルな活性,および,ERKによる転写因子c-Fosのデジタルへの変換が報告されている.この2つのケースではどちらもERKの持続的な活性が必要で,ERKの持続性はERKからRafへの正のフィードバック14),受容体の活性化の協同性21),RasあるいはRap1の活性キネティクスにおける相加的な効果22) などに由来することが報告されており,細胞あるいはリガンドごとに決定されていると考えられる.
ERKの持続的な活性は細胞の制御に大きな影響をあたえる.もっともよく研究されているラットの副腎髄質に由来するPC12細胞14,22) やヒトの乳がんに由来するMCF-7細胞23) においては,一過的なERKの活性では細胞増殖,持続的なERKの活性では細胞分化,という異なる表現型を示すことがわかっている.異なる表現型を誘導できる理由のひとつは,細胞の制御の初期の段階において,持続的なERKの活性のみが,ERK自体により遺伝子発現の誘導された転写因子を活性化できることに由来すると考えられる.c-Fosあるいはc-Mycなどの転写因子はERKに依存的な早期発現遺伝子としてコードされ,かつ,DEFドメインというERKセンサー配列をもつ24).DEFドメインをもつタンパク質はERKにより安定化されるが,一方で,そのmRNAが発現したときERKの活性がすでに抑制されている場合にはそのタンパク質はすみやかに分解される.よって,ERKの活性が持続的な場合にのみ,この遺伝子はタンパク質として発現する.膜受容体の活性化によるc-fos mRNAの発現には約30分,c-Fosタンパク質の発現には約60~90分かかるため,これらの遺伝子がタンパク質として発現するためには90分以上の持続性をもつERKの活性が必要となる.このネットワークでは同じERKの活性でも一過性の活性はただのノイズと考えられ,転写にはシグナルとして伝達されない.よって,このc-Fosの発現の段階で0か1かのデジタルな出力が生じる25).
c-Fosの発現のためには,核に存在するERKによるc-fos mRNAの発現と,細胞質における持続的なERKの活性によるc-Fosの安定化の2つの条件が必要である.この分子制御ネットワークは,さきに述べたANDゲートというネットワークモチーフで表わされる16).ANDゲートは,2つの入力がそろったときのみ1のシグナルを出力し,どちらか一方の入力,あるいは,どちらの入力もない場合には0のシグナルを出力する論理回路で,シグナルの選り分けを行っている.おもしろいことに,この経路では細胞質におけるERKの活性が持続的であっても,核におけるERKの活性は脱リン酸化酵素により早々に不活性化され一過性となる.このことにより,c-fos mRNAの発現は一過性となる.細胞質と核におけるERKの活性の持続性の違いがc-Fosのデジタルな出力を定まった時間内に生じ,転写因子c-Fosによる遺伝子発現を厳密に制御していることが示唆される.
ERK経路と同様に,NF-κB経路もまたデジタルな活性を生じる機能をもつことがわかってきた.NF-κBは炎症やがんなどにおけるシグナル伝達系においてシグナルを集約する転写因子であり,免疫細胞の分化においても重要な役割をはたしている.NF-κB経路には,TNFαなどの炎症性サイトカインあるいは抗原受容体により活性化されるp50,RelA,c-Relに依存的な古典的な経路(canonical pathway)と,BAFFあるいはCD40Lにより活性化されるp52およびRelBに依存的な非古典的な経路(non-canonical pathway)の2つがある26,27).このうち,古典的な経路はそのダイナミクスが比較的よく研究されている.TNFαなどの刺激により,NF-κBの阻害タンパク質であるIκBがシグナルに依存的に分解されることによりNF-κBが活性化され,NF-κBは細胞質から核へと移行し遺伝子の発現を誘導する.しかし,その早期発現遺伝子にコードされるタンパク質にはNF-κBの阻害タンパク質であるIκBおよびA20が含まれているので,活性化されたNF-κBはこれらのタンパク質によりふたたび阻害され,細胞に刺激をあたえてから約60分のちには不活性な状態にもどる.このような正と負の反応制御によりNF-κBは細胞質と核とを行き来する振動を誘発すること,IκBのアイソフォームのあいだでその振動に対する効果の異なることが見い出されている2).このとき,ウェスタンブロット法などを用いて調べた細胞の集団全体におけるNF-κBの活性は,細胞にあたえた刺激の濃度の上昇にともないだんだんと上昇するアナログなものにみえる.しかしそののち,1細胞イメージング法による解析により,刺激の量が増加すると振動する細胞の個数が増加するという0か1かのデジタルな活性であることが明らかになった28,29).この現象の背景には,NF-κBの活性化の有無を決定する閾値を決定する機構の存在が考えられてきた.しかし,これを証明する適切なレポーターアッセイ系がないなどの理由により,それをつかさどるシグナル伝達の制御機構はいままで明らかにされていなかった.
最近,筆者らは,このNF-κBの活性化の閾値を決定する機構の存在とその制御の実体を明らかにするため,B細胞受容体のシグナル伝達系におけるCARMA1-TAK1-IKK経路に着目し,これらのタンパク質の活性の動態とNF-κBの活性との関係をシステム生物学的に解析した.B細胞受容体の活性化により,プロテインキナーゼCによりCARMA1のSer668がリン酸化されるとCARMA1は足場タンパク質として機能し,TAK1などと複合体を形成してそれを活性化させ,下流のIKKの活性化を促進する.このとき,IKKが活性化されるとCARMA1のSer578のリン酸化を介した正のフィードバックがはたらき,IKKの活性をより増強させることが報告されていた30).一般的に,正のフィードバック制御はERKの場合のようにシグナルの活性を持続させるが,ときとして,デジタルな応答を生じることもできる.トリB細胞DT40株を用いた定量的な実験の結果,この正のフィードバックはTAK1の活性化の持続時間を伸ばし,さらに,IKKおよびNF-κBのオンオフ活性を導くことが明らかになった.さらに,この数理解析では,IKKが活性化したときのIKKオリゴマーの協同的な活性化が重要であるという仮説が得られた.最近の研究ではこの仮説を裏づけるように,タンパク質の立体構造解析の結果からIKKのあいだの複合体の形成がその活性化の制御に重要であることが報告されており31),IKKのあいだの協同的な活性化の機構がNF-κBのシグナルのデジタルな出力に大きく関与すると考えられる.このことは,IKKが単にNF-κBの上流にあるというだけでなく,系のなかで重要な役割をはたしているというこれまでの知見とも一致している.
ここまでおもに,正のフィードバック制御によりひき起こされる分子ダイナミクスの顕著な変化について解説してきたが,正のフィードバック制御の報告はむしろ多くはない.一方で,負のフィードバック制御はシグナル伝達系には必ずといっていいほどよくみられる.これらには,Gab,Shc,Crkなどアダプタータンパク質の負のリン酸化制御によるもの,また,DUSP(ERKを標的),A20(IKKを標的),IκB(NF-κBを標的),Socs3(STAT3を標的),Mig6(EGFRを標的)などの転写産物を介したシグナルの減衰などが多く報告されている32).負のフィードバック制御はシグナルの停止にくわえ,非線形な出力をより線形にしたり,系の全体を安定に保ったり,ノイズを除去したりする役割がある.反応を停止することから,負のフィードバック制御は単純な一過性のピークを示すことが多い.そのなかで,多くの研究者の興味をひいているのはシグナルの振動である.
細胞周期や概日リズムの振動は,さきのNF-κB経路の例において解説した正のフィードバックにくわえ,負のフィードバック制御により形成されている33).ERKシグナルやNF-κBシグナルについても振動が報告されている20,28,29,34).しかし,細胞周期や概日リズムのように振動の意味が明確な系とは異なり,このシグナルの振動が細胞の機能として必要なのかどうかという点についてはいまだ明確な答えは得られていない.とくに,ERKシグナルの振動は細胞密度の違いやパラメーターの違いなどある特定の条件のもとでしかみられないのに対し20,34),NF-κBシグナルの振動はリガンドが異なっても細胞が異なっても再現性よく観察される.このように再現性の高い挙動の場合,それを生じる機構および機能があるはずである.現在,NF-κBシグナルの振動の意義としては,その振動の回数が遺伝子発現の強度を決める29),あるいは,振動が収束するときのエンドポイントの活性が重要である,という仮説がある.実際に,NF-κBの研究者の多くは,NF-κBシグナルの振動にある種のロマンを求めているようなところがある.
これらの振動の意義に関しては今後の研究により明らかにされてくるであろうが,いっけん何の意味があるのかもわからない振動の生理的な意義を解くことにより得られる利点は,振動を生じる制御のしくみを新たな細胞の制御の方法として用いることができることである.一般的に,このような特徴的な分子ダイナミクスはそれを生じるネットワークモチーフが存在してこそ生じる.米国を中心に急速に進められている合成生物学は,シグナル伝達系のネットワークモチーフを組み合わせ,それを人工的に構成し細胞を操作しようとするものである.そのさきがけとなる研究では,キメラのシグナルタンパク質を利用した酵母の細胞転換などに成功している35,36).
ERK経路あるいはNF-κB経路のネットワークの構造は分子ダイナミクスに大きな影響をあたえるが,細胞における分子の局在もまた,ダイナミクスの形成に重要な役割をはたす.細胞における局在は分子の濃度を局所的に高め,分子のあいだの反応の効率をあげたり,シグナルのオンオフ応答をひき出したりする効果がある.たとえば,PHドメインをもつアダプタータンパク質はPI3Kの活性により細胞膜に生じるPIP3に結合して局在し,その代表例であるGabやIRSはPI3K,RasGAP,SHP2などのタンパク質をひきつけることによりこれらのあいだの反応の効率をあげる.GabおよびIRSなどのアダプタータンパク質はシグナルの活性化の役割をもつとともに,シグナルが上昇した場合には負のフィードバックを介してシグナルを停止する役割も担う.これらのシグナルのオンオフのしくみは数理モデルにより明確に説明されている37).
また,NF-κB経路におけるCARMA1 38) やERK経路におけるKSR 35) は,それぞれTAK-IKK経路やRaf-MEK-ERK経路などのキナーゼ反応の足場タンパク質としてはたらき,これらのタンパク質を集合させてカスケード反応の特異性および効率をあげたり,シグナルのスイッチ応答をひき起こしたりする39).しかし,これらの反応効率の上昇やスイッチ応答は足場タンパク質とそれと結合するキナーゼとの比率に大きく依存し,この比率が至適でない場合には逆にキナーゼ反応の効率を下げることもある40).また,さきに述べたNF-κB経路の例にあるように,細胞質と核という異なるコンパートメントのあいだの移行もまた,反応の特異性の上昇に貢献する.このような細胞における局在制御の興味深い例として,最近,報告された細胞周期に関連するタンパク質の細胞質と核のあいだの移行と正のフィードバックとの連動がある41).ここでは,なぜ細胞分裂は不可逆反応なのかということに注目し,そのことがシステム生物学的に証明された.すなわち,間期から分裂期にかかる細胞周期においてCdk1-Cyclin B1複合体は細胞質から核へと移行するが,このとき,Cyclin B1のリン酸化を介して細胞質のCdk1-Cyclin B1複合体をさらに核へと取り込む正のフィードバック制御がはたらくことがわかった.この正のフィードバック制御の高い協同性により,ヒステリシス(くわえる力を最初の状態のときと同じにしても,状態は完全にはもどらないこと)という反応の不可逆性が生じ細胞分裂に突入するのである.このように,細胞の動態あるいはネットワークだけではなく,細胞における局在もまた,反応の特異性の発揮に大きく貢献することが徐々にわかってきている.
これまで述べてきたように,ボトムアップ型のモデリングのアプローチは,さまざまな制御の機構を明らかにすることにより,細胞の制御の機構には一定の規則性のあることを示してきた.しかし,これらのモデリングの研究には多くの事前知識が必要である.一方で,近年のオミクス技術の発展により,トランスクリプトーム,プロテオーム,メタボローム,エピジェネティクスなどの解析が比較的容易に行えるようになり,これらのトップダウン型の研究とボトムアップ型の研究とを融合し,仮説のない状態から分子の機能を特定したり,細胞の形質を決定する分子マーカーを見い出したりしようという研究が増加している.もともと,トップダウン型のオミクス研究は米国においてさかんで,ボトムアップ型のモデリング研究は欧州においてさかんという傾向があったが,最近では,欧州においてもオミクス研究が広く行われるようになり,これらの方法論の統合は標準になりつつある.現在,このような研究はさまざまながんを対象としているが,最近では,免疫学などのほかの分野においてもオミクス研究とモデリング研究とをあわせた研究が増加してきている.制御性T細胞におけるトランスクリプトームのデータから転写因子Foxp3のネットワークのデジタルな制御をモデリングにより同定したのは,そういった例のひとつである42).
今後,こういったオミクス研究とモデリング研究との統合解析のいくさきは,ゲノムワイド関連解析(genome-wide association study:GWAS)などゲノムコホート研究43) との融合であろう(図5).たとえば,ある疾病の発症の有無,あるいは,発症するときの時期は,各人で大きく異なる.発症の有無は各人にとっては0か1かのデジタルな応答と同義である.また,発症する時期は遺伝子だけでなく環境の影響により大きく異なるが,このことはこれらのパラメーターに対する個人ごとの違いとしてとらえることができるだろう.乳がんをはじめとしたヒトの大規模ゲノム解析の結果から,がんの遺伝子の異常は,PIK3CA,PIK3R1,PTEN,AKT1,MAP3K1,KRAS,BRAF,EGFRなどのEGF受容体あるいはErbBのかかわる膜受容体からのシグナル伝達系に極端にかたよっていることが報告されている44).しかし,おのおのの患者のレベルにおいてどのような遺伝子異常の組合せがあるのか,また,その異常度と予後との関係などについてはいまだ明らかにされていない.このような入力と出力との関係の予測はネットワークのモデリングにより可能になるかもしれない.たとえば,遺伝子の増幅はタンパク質の初期の濃度の上昇,タンパク質の変異は分子間相互作用や酵素-基質間の結合親和性の変化などのパラメーターとして表わすことができる.さらに,これにほかの遺伝子の相互作用やフィードバック制御などのネットワーク構造がくわわると,個体レベルでの発症は非線形になるはずである.このような系では,1遺伝子のわずかな発現量の違いやタンパク質のあいだの親和性の違いが,個体のあいだの決定的な違いを生みだしてもおかしくはない.また一方で,個人ごとに生活習慣などを変えて遺伝子の活性化のパラメーターを制御し発症を未然に防ぐという,個別化予防医学のようなことも可能になるのではないかと想像している.
このレビューでは,ヒトゲノムの研究と連動して発展したシグナル伝達系のシステム生物学的な研究について,ここ15年間の動向を駆け足で説明した.筆者が実験を中心に研究を進めていることから実験系の成果をおもにとりあげたが,ここでは説明しなかった理論的な研究も多くある45-48).ここで紹介したネットワーク制御の理論は,シグナル伝達系だけでなく転写因子あるいは細胞間の制御にも応用できる汎用性の高い考え方である.2012年,Mycoplasma菌の全細胞モデルが発表され49),大きな話題になった.まだ,1細菌のモデルではある.しかし,このことに触発された多くのグループは,このさきにヒトの全ゲノムモデルをみすえているのはまちがいないだろう.欧米のシステム生物学の研究の現場ではすでに臨床研究者との連携が急速に進んでおり,いまの時点ではありえないと思うかもしれないが,ヒトゲノムにもとづいた疾患の発症予測モデルが発表される日もそう遠くないような気がしている.ただ,こういった大規模モデルを構築する際に,ただ漫然と大きなモデルを構築するのではなく,なんらかの理論にもとづいたモデルを構築したほうが高い予測精度をもつことが,いままでのシグナル伝達のモデリングの研究の結果からも示唆されている.いずれにしても,システム生物学の強みは“予測”である.多くの実験研究者がモデルやシミュレーションをひとつの実験ツールと考えられるよう,今後,わが国においてもさまざまな研究の連携の進むことが望まれる.
略歴:2003年 東京農工大学大学院連合農学研究科にて博士号取得,理化学研究所ゲノム科学総合研究センター 研究員,上級研究員,チームリーダーを経て,2009年より同 免疫アレルギー科学総合研究センター(現 統合生命医科学研究センター)チームリーダー.
研究テーマ:シグナル転写ネットワークのシステム生物学.
抱負:いまは,細胞に含まれる異なる階層をつなげること.将来は,また創薬に貢献したい.
© 2014 岡田 眞里子 Licensed under CC 表示 2.1 日本
(理化学研究所統合生命医科学研究センター 統合細胞システム研究チーム)
email:岡田眞里子
領域融合レビュー, 3, e001 (2014) DOI: 10.7875/leading.author.3.e001
Mariko Okada: Prediction of cellular responses based on regulatory rules of signal transduction pathways.
要 約
細胞内のシグナル伝達系は,1980年代より,がんをはじめとした疾病との関連性により隆盛をきわめた分野である.近年では,2003年のヒトゲノム解読完了と前後して注目をあびはじめたシステム生物学において,実験と理論との融合がもっとも進んでいる分野でもある.このレビューでは,細胞内のシグナル伝達系のシステム生物学的な研究に対し期待されている社会的および学術的な背景をはじめ,シグナル伝達系のダイナミクスとその背後にある制御のデザイン,頻繁にみられる制御の規則性,細胞の制御への応用などにふれる.対象としては,システム生物学の立場からみてさまざまな研究の進んでいるERK経路およびNF-κB経路について中心に紹介する.最後に,今後,このようなシステム生物学が何をめざして進んでいくのか,その将来を展望する.
はじめに
細胞内シグナル伝達系は,環境からの情報を転写因子に伝達し遺伝子の発現を制御することにより細胞の運命決定を担う分子機構で,タンパク質のリン酸化,相互作用,分解など生化学的な反応の集合体としてなりたつ.ヒトの全遺伝子の約20%がシグナル伝達系にかかわるともいわれ,シグナル伝達系はそれ自体が大きなネットワークである.しかし,このネットワークの入出力の関係はさまざまな制御のために非線形性であり,おのおののシグナル活性を足し算することにより細胞の応答を予測することはほとんどできない.このことはシグナル伝達系の異常と関連の深い,がん,糖尿病,炎症など,さまざまな疾病の解明および治療において大きな問題である.このような背景から,1990年代の後半から,シグナル伝達系の分子ダイナミクスと細胞応答とに規則性を見い出そうとする“システム生物学”がさかんになった.このレビューでは,約15年間にわたるシグナル伝達系のシステム生物学における研究の動向とその将来について紹介する.
1.シグナル伝達系のシステム生物学的な研究の背景
1990年代の後半から2000年代の初頭にかけて,主要なシグナル伝達系であるERK経路およびNF-κB経路のデジタルな応答および振動とその機序に関する研究が発表された1,2).これらの論文により,シグナル伝達系はそれまで考えられていた以上にダイナミクスに富んだ系であり,そこにはいまだ明らかにされていない分子制御のかくされていることが示された.さらにこのころ,ヒトゲノムが解読された.それを機に,ゲノム情報をもとにしたハイスループットな解析技術やそれをサポートするツールが充実し,これらがオミクス解析を中心としたトップダウン型の研究,および,ターゲットをしぼってモデルを構築して解析するボトムアップ型の研究の2つのタイプのシステム生物学の研究を加速した.トップダウン型の研究においては,生物における大規模かつ静的な分子間相互作用のネットワークが社会的なネットワークと類似した構造をもつことが明らかにされ,そこに頻出するネットワークモチーフを同定することにより生物のネットワークの普遍性が指摘された3,4).また,ボトムアップ型の研究においては,制御工学,代謝工学,生化学の影響をうけた動的なモデルが構築され,シグナルの動態を決定する分子制御が予測された5,6).このようにして,さまざまな実験データを理論的に整理するため,あるいは,生物をシステムとして理解するため,システム生物学が広まっていった(図1).
細胞内シグナル伝達系のシステム生物学的な研究の目的のひとつは,複雑なシグナルのネットワークから細胞の応答を“予測”することである.この背景には,1990年代後半から市販されるようになった分子標的薬,たとえば,EGF(ErbB1)受容体の標的薬剤であるゲフィチニブ(肺がんを対象とする),Bcr-Ablの標的薬剤であるイマチニブ(慢性骨髄性白血病を対象とする),ErbB2の標的薬剤であるトラスツズマブ(乳がんを対象とする),などの登場がある7).ゲフィチニブおよびトラスツズマブは膜受容体であるErbBファミリーを標的とするが,ErbBファミリーは活性化にともないホモ二量体あるいはヘテロ二量体を形成することから,たとえば,ErbB1を第1の標的としても細胞において共発現するほかのErbBファミリーを間接的に阻害してしまう.しかし,どれだけ実験の精度をあげても,細胞においてErbB1とほかのErbBファミリーに対する薬剤の効果の違いを定量的に切り分けることは困難である.また,ゲフィチニブあるいはトラスツズマブの投与によりErbBファミリーを長期間にわたり阻害すると,ほかの受容体からの経路を経由してErbBファミリーの下流の経路が活性化するといった薬剤耐性の問題も生じていた8).こういったことから,薬剤の主効果および副効果の影響を定量的に評価し,そこから生じるかもしれない副作用を薬剤開発の早い段階から予測することが期待されるようになった.これらの薬剤を開発した製薬企業では早くから数理モデルを用いたシグナル伝達系の理論的な研究を行っており,これらと連携した研究グループ,また,そこから派生したベンチャー企業では,シグナル伝達系を対象とした大規模なモデルの構築や9),それを利用した制御分子や薬剤効果の予測,また,オミクスデータから統計的な手法を用いてネットワーク予測を行い10),細胞運命の決定因子を同定している.
2.シグナルの特異性と認識
細胞内シグナル伝達系のシステム生物学的な研究のもうひとつの目的は,シグナル伝達系の特異性およびシグナル伝達の分子機構の解明である.というのも,膜受容体とリガンドとの特異的な結合を除くと,シグナル伝達系にかかわるタンパク質の反応特異性は比較的低く,シグナル伝達系の特異性および認識をつかさどる実体はまだよくわかっていないからである.たとえば,酵素においては鍵と鍵穴のような酵素と基質との立体構造の認識に関する理論があり,酵素の特異性を表わすパラメーターなどから生成物の量をある程度は予測することができる.しかし,シグナル伝達のネットワークの特異性は,そこに含まれる酵素,たとえば,プロテインキナーゼの特異性として十分には説明できない.なぜなら,プロテインキナーゼは基質のひとつであるATPに対する結合特異性は高いが,タンパク質の基質に対する特異性は一般的に低いからである11).このことは,SH2ドメインとチロシンリン酸化タンパク質とのあいだのような分子間相互作用についても同じである12).このような分子認識の特異性の低さはシグナル伝達のネットワークに冗長性(redundancy)および頑強性(robustness)をあたえ,遺伝子に変異や欠損などの異常が起こってもネットワーク全体の機能を維持できるという点で生物にとり利点である.しかし一方で,シグナル伝達の存在の意味とは,このような冗長性のなかから特定の情報を選り分けて核において特定の転写因子を活性化することにあるはずである.その機構を明らかにすることができれば,そのことを細胞応答の予測に利用し,さらには,副作用のない細胞の制御が可能になるはずである.
この冗長性および特異性という特徴をあわせもつシグナル伝達系の基盤のひとつは,シグナルタンパク質のマルチドメイン性13)(モジュール性),および,タンパク質のもつ活性部位を直接に制御するのではなく,そこから離れた制御部位を修飾し,タンパク質の立体構造を変化させることにより活性を遠隔制御するというアロステリック制御11) である.マルチドメイン性はひとつのシグナルタンパク質に複数のタンパク質との相互作用を可能にし,たとえば,アダプタータンパク質であるGab2,Grb2,ShcはSH2ドメインあるいはPTBドメインを介して互いに膜受容体と結合し,どのアダプタータンパク質を経由してもERK経路を活性化できるような冗長なネットワークを形成する(図2a).また同様に,これらのシグナルタンパク質にはリン酸化などアロステリックな制御がくわわり,正のフィードバックあるいは負のフィードバックの作用点としてはたらいたり,シグナルタンパク質の局在性を変化させたりする.このなかで,正のアロステリック制御はシグナルタンパク質のあいだの協同性を高め,非線形な出力,すなわち,特異的な活性をひき出す(図2b).
3.細胞のデジタルな応答
シグナル伝達系の役目のひとつはアナログなシグナルのデジタルへの変換である.これは,少しずつ異なる情報量を1か0の情報量に変える機構である.たとえば,リガンドと受容体との反応強度は,シグナル全体の強度と転写による出力に大きく影響する.これらの入力の違いは,成長因子や抗体など異なるリガンドということもあるし,同じリガンドであっても濃度が異なる場合もある.このような違いには,リガンドの親和性あるいは受容体の発現量が大きく影響する.しかし,この親和性あるいは発現量の違いとは,あくまでも数値の大小の違い(アナログ性の違い)である.ところが,時間にして数分から数十分のシグナル伝達系の反応をへると,このアナログなシグナル量の違いが,0か1のデジタルな転写活性の違いとして現われる場合がある.このような転写による出力のデジタルへの変換は細胞内のシグナル伝達系のもつ機能のひとつであり,細胞を質的に変えることのできる可能性をもつ.これまでの理論的な研究により,正のフィードバック制御1,14),相互阻害15),のちに述べるANDゲート16),のようなネットワークモチーフが細胞のデジタルな活性化を生じることが示されている(図3).さらに,このような明らかなネットワークモチーフをもたない場合でも,たとえば,ERKの分配的な二重リン酸化反応の場合には,2つのリン酸化を脱リン酸化する酵素の量が少ないような特別なパタメーターの条件において系は実質的な相互阻害としてはたらき,デジタルな出力が生じるようになる17).ネットワークと動態との関係については,つぎに述べるERK経路がもっともよく研究されている.
4.ERK経路の動態
ERKは,EGF受容体,PDGF受容体,インスリン受容体,IGF受容体,FGF受容体,Trk受容体など,さまざまな膜受容体からのシグナルを集約するセリン/スレオニンキナーゼであり,細胞の増殖,分化,成長など,さまざまな細胞の制御にかかわる18).ERKはRas-Raf-MEK-ERK経路をへて活性され,活性化されると細胞質から核へと移行し,ELKあるいはCREBなどの転写因子を活性化し遺伝子の発現を誘導する.このERK経路では,さきに述べた,正のフィードバック制御14),相互阻害17),ANDゲート16) の,デジタル応答を生じるすべてのネットワークモチーフが報告されている.さらに,その活性の動態としても,一過性の活性あるいは持続性の活性のほか,活性の振動も報告されている19,20)(図4).このようなさまざまな反応の特性は理論から想定されるRaf-MEK-ERK経路における二重リン酸化の反応様式(distributiveあるいはprocessive)の違いにより大きな影響をうける.ERK経路におけるデジタルな出力としては,ERKそのもののデジタルな活性,および,ERKによる転写因子c-Fosのデジタルへの変換が報告されている.この2つのケースではどちらもERKの持続的な活性が必要で,ERKの持続性はERKからRafへの正のフィードバック14),受容体の活性化の協同性21),RasあるいはRap1の活性キネティクスにおける相加的な効果22) などに由来することが報告されており,細胞あるいはリガンドごとに決定されていると考えられる.
ERKの持続的な活性は細胞の制御に大きな影響をあたえる.もっともよく研究されているラットの副腎髄質に由来するPC12細胞14,22) やヒトの乳がんに由来するMCF-7細胞23) においては,一過的なERKの活性では細胞増殖,持続的なERKの活性では細胞分化,という異なる表現型を示すことがわかっている.異なる表現型を誘導できる理由のひとつは,細胞の制御の初期の段階において,持続的なERKの活性のみが,ERK自体により遺伝子発現の誘導された転写因子を活性化できることに由来すると考えられる.c-Fosあるいはc-Mycなどの転写因子はERKに依存的な早期発現遺伝子としてコードされ,かつ,DEFドメインというERKセンサー配列をもつ24).DEFドメインをもつタンパク質はERKにより安定化されるが,一方で,そのmRNAが発現したときERKの活性がすでに抑制されている場合にはそのタンパク質はすみやかに分解される.よって,ERKの活性が持続的な場合にのみ,この遺伝子はタンパク質として発現する.膜受容体の活性化によるc-fos mRNAの発現には約30分,c-Fosタンパク質の発現には約60~90分かかるため,これらの遺伝子がタンパク質として発現するためには90分以上の持続性をもつERKの活性が必要となる.このネットワークでは同じERKの活性でも一過性の活性はただのノイズと考えられ,転写にはシグナルとして伝達されない.よって,このc-Fosの発現の段階で0か1かのデジタルな出力が生じる25).
c-Fosの発現のためには,核に存在するERKによるc-fos mRNAの発現と,細胞質における持続的なERKの活性によるc-Fosの安定化の2つの条件が必要である.この分子制御ネットワークは,さきに述べたANDゲートというネットワークモチーフで表わされる16).ANDゲートは,2つの入力がそろったときのみ1のシグナルを出力し,どちらか一方の入力,あるいは,どちらの入力もない場合には0のシグナルを出力する論理回路で,シグナルの選り分けを行っている.おもしろいことに,この経路では細胞質におけるERKの活性が持続的であっても,核におけるERKの活性は脱リン酸化酵素により早々に不活性化され一過性となる.このことにより,c-fos mRNAの発現は一過性となる.細胞質と核におけるERKの活性の持続性の違いがc-Fosのデジタルな出力を定まった時間内に生じ,転写因子c-Fosによる遺伝子発現を厳密に制御していることが示唆される.
5.NF-κB経路の動態
ERK経路と同様に,NF-κB経路もまたデジタルな活性を生じる機能をもつことがわかってきた.NF-κBは炎症やがんなどにおけるシグナル伝達系においてシグナルを集約する転写因子であり,免疫細胞の分化においても重要な役割をはたしている.NF-κB経路には,TNFαなどの炎症性サイトカインあるいは抗原受容体により活性化されるp50,RelA,c-Relに依存的な古典的な経路(canonical pathway)と,BAFFあるいはCD40Lにより活性化されるp52およびRelBに依存的な非古典的な経路(non-canonical pathway)の2つがある26,27).このうち,古典的な経路はそのダイナミクスが比較的よく研究されている.TNFαなどの刺激により,NF-κBの阻害タンパク質であるIκBがシグナルに依存的に分解されることによりNF-κBが活性化され,NF-κBは細胞質から核へと移行し遺伝子の発現を誘導する.しかし,その早期発現遺伝子にコードされるタンパク質にはNF-κBの阻害タンパク質であるIκBおよびA20が含まれているので,活性化されたNF-κBはこれらのタンパク質によりふたたび阻害され,細胞に刺激をあたえてから約60分のちには不活性な状態にもどる.このような正と負の反応制御によりNF-κBは細胞質と核とを行き来する振動を誘発すること,IκBのアイソフォームのあいだでその振動に対する効果の異なることが見い出されている2).このとき,ウェスタンブロット法などを用いて調べた細胞の集団全体におけるNF-κBの活性は,細胞にあたえた刺激の濃度の上昇にともないだんだんと上昇するアナログなものにみえる.しかしそののち,1細胞イメージング法による解析により,刺激の量が増加すると振動する細胞の個数が増加するという0か1かのデジタルな活性であることが明らかになった28,29).この現象の背景には,NF-κBの活性化の有無を決定する閾値を決定する機構の存在が考えられてきた.しかし,これを証明する適切なレポーターアッセイ系がないなどの理由により,それをつかさどるシグナル伝達の制御機構はいままで明らかにされていなかった.
最近,筆者らは,このNF-κBの活性化の閾値を決定する機構の存在とその制御の実体を明らかにするため,B細胞受容体のシグナル伝達系におけるCARMA1-TAK1-IKK経路に着目し,これらのタンパク質の活性の動態とNF-κBの活性との関係をシステム生物学的に解析した.B細胞受容体の活性化により,プロテインキナーゼCによりCARMA1のSer668がリン酸化されるとCARMA1は足場タンパク質として機能し,TAK1などと複合体を形成してそれを活性化させ,下流のIKKの活性化を促進する.このとき,IKKが活性化されるとCARMA1のSer578のリン酸化を介した正のフィードバックがはたらき,IKKの活性をより増強させることが報告されていた30).一般的に,正のフィードバック制御はERKの場合のようにシグナルの活性を持続させるが,ときとして,デジタルな応答を生じることもできる.トリB細胞DT40株を用いた定量的な実験の結果,この正のフィードバックはTAK1の活性化の持続時間を伸ばし,さらに,IKKおよびNF-κBのオンオフ活性を導くことが明らかになった.さらに,この数理解析では,IKKが活性化したときのIKKオリゴマーの協同的な活性化が重要であるという仮説が得られた.最近の研究ではこの仮説を裏づけるように,タンパク質の立体構造解析の結果からIKKのあいだの複合体の形成がその活性化の制御に重要であることが報告されており31),IKKのあいだの協同的な活性化の機構がNF-κBのシグナルのデジタルな出力に大きく関与すると考えられる.このことは,IKKが単にNF-κBの上流にあるというだけでなく,系のなかで重要な役割をはたしているというこれまでの知見とも一致している.
6.シグナル伝達系における負のフィードバック制御
ここまでおもに,正のフィードバック制御によりひき起こされる分子ダイナミクスの顕著な変化について解説してきたが,正のフィードバック制御の報告はむしろ多くはない.一方で,負のフィードバック制御はシグナル伝達系には必ずといっていいほどよくみられる.これらには,Gab,Shc,Crkなどアダプタータンパク質の負のリン酸化制御によるもの,また,DUSP(ERKを標的),A20(IKKを標的),IκB(NF-κBを標的),Socs3(STAT3を標的),Mig6(EGFRを標的)などの転写産物を介したシグナルの減衰などが多く報告されている32).負のフィードバック制御はシグナルの停止にくわえ,非線形な出力をより線形にしたり,系の全体を安定に保ったり,ノイズを除去したりする役割がある.反応を停止することから,負のフィードバック制御は単純な一過性のピークを示すことが多い.そのなかで,多くの研究者の興味をひいているのはシグナルの振動である.
細胞周期や概日リズムの振動は,さきのNF-κB経路の例において解説した正のフィードバックにくわえ,負のフィードバック制御により形成されている33).ERKシグナルやNF-κBシグナルについても振動が報告されている20,28,29,34).しかし,細胞周期や概日リズムのように振動の意味が明確な系とは異なり,このシグナルの振動が細胞の機能として必要なのかどうかという点についてはいまだ明確な答えは得られていない.とくに,ERKシグナルの振動は細胞密度の違いやパラメーターの違いなどある特定の条件のもとでしかみられないのに対し20,34),NF-κBシグナルの振動はリガンドが異なっても細胞が異なっても再現性よく観察される.このように再現性の高い挙動の場合,それを生じる機構および機能があるはずである.現在,NF-κBシグナルの振動の意義としては,その振動の回数が遺伝子発現の強度を決める29),あるいは,振動が収束するときのエンドポイントの活性が重要である,という仮説がある.実際に,NF-κBの研究者の多くは,NF-κBシグナルの振動にある種のロマンを求めているようなところがある.
これらの振動の意義に関しては今後の研究により明らかにされてくるであろうが,いっけん何の意味があるのかもわからない振動の生理的な意義を解くことにより得られる利点は,振動を生じる制御のしくみを新たな細胞の制御の方法として用いることができることである.一般的に,このような特徴的な分子ダイナミクスはそれを生じるネットワークモチーフが存在してこそ生じる.米国を中心に急速に進められている合成生物学は,シグナル伝達系のネットワークモチーフを組み合わせ,それを人工的に構成し細胞を操作しようとするものである.そのさきがけとなる研究では,キメラのシグナルタンパク質を利用した酵母の細胞転換などに成功している35,36).
7.細胞における局在と分子ダイナミクス
ERK経路あるいはNF-κB経路のネットワークの構造は分子ダイナミクスに大きな影響をあたえるが,細胞における分子の局在もまた,ダイナミクスの形成に重要な役割をはたす.細胞における局在は分子の濃度を局所的に高め,分子のあいだの反応の効率をあげたり,シグナルのオンオフ応答をひき出したりする効果がある.たとえば,PHドメインをもつアダプタータンパク質はPI3Kの活性により細胞膜に生じるPIP3に結合して局在し,その代表例であるGabやIRSはPI3K,RasGAP,SHP2などのタンパク質をひきつけることによりこれらのあいだの反応の効率をあげる.GabおよびIRSなどのアダプタータンパク質はシグナルの活性化の役割をもつとともに,シグナルが上昇した場合には負のフィードバックを介してシグナルを停止する役割も担う.これらのシグナルのオンオフのしくみは数理モデルにより明確に説明されている37).
また,NF-κB経路におけるCARMA1 38) やERK経路におけるKSR 35) は,それぞれTAK-IKK経路やRaf-MEK-ERK経路などのキナーゼ反応の足場タンパク質としてはたらき,これらのタンパク質を集合させてカスケード反応の特異性および効率をあげたり,シグナルのスイッチ応答をひき起こしたりする39).しかし,これらの反応効率の上昇やスイッチ応答は足場タンパク質とそれと結合するキナーゼとの比率に大きく依存し,この比率が至適でない場合には逆にキナーゼ反応の効率を下げることもある40).また,さきに述べたNF-κB経路の例にあるように,細胞質と核という異なるコンパートメントのあいだの移行もまた,反応の特異性の上昇に貢献する.このような細胞における局在制御の興味深い例として,最近,報告された細胞周期に関連するタンパク質の細胞質と核のあいだの移行と正のフィードバックとの連動がある41).ここでは,なぜ細胞分裂は不可逆反応なのかということに注目し,そのことがシステム生物学的に証明された.すなわち,間期から分裂期にかかる細胞周期においてCdk1-Cyclin B1複合体は細胞質から核へと移行するが,このとき,Cyclin B1のリン酸化を介して細胞質のCdk1-Cyclin B1複合体をさらに核へと取り込む正のフィードバック制御がはたらくことがわかった.この正のフィードバック制御の高い協同性により,ヒステリシス(くわえる力を最初の状態のときと同じにしても,状態は完全にはもどらないこと)という反応の不可逆性が生じ細胞分裂に突入するのである.このように,細胞の動態あるいはネットワークだけではなく,細胞における局在もまた,反応の特異性の発揮に大きく貢献することが徐々にわかってきている.
8.ボトムアップからトップダウンへ
これまで述べてきたように,ボトムアップ型のモデリングのアプローチは,さまざまな制御の機構を明らかにすることにより,細胞の制御の機構には一定の規則性のあることを示してきた.しかし,これらのモデリングの研究には多くの事前知識が必要である.一方で,近年のオミクス技術の発展により,トランスクリプトーム,プロテオーム,メタボローム,エピジェネティクスなどの解析が比較的容易に行えるようになり,これらのトップダウン型の研究とボトムアップ型の研究とを融合し,仮説のない状態から分子の機能を特定したり,細胞の形質を決定する分子マーカーを見い出したりしようという研究が増加している.もともと,トップダウン型のオミクス研究は米国においてさかんで,ボトムアップ型のモデリング研究は欧州においてさかんという傾向があったが,最近では,欧州においてもオミクス研究が広く行われるようになり,これらの方法論の統合は標準になりつつある.現在,このような研究はさまざまながんを対象としているが,最近では,免疫学などのほかの分野においてもオミクス研究とモデリング研究とをあわせた研究が増加してきている.制御性T細胞におけるトランスクリプトームのデータから転写因子Foxp3のネットワークのデジタルな制御をモデリングにより同定したのは,そういった例のひとつである42).
今後,こういったオミクス研究とモデリング研究との統合解析のいくさきは,ゲノムワイド関連解析(genome-wide association study:GWAS)などゲノムコホート研究43) との融合であろう(図5).たとえば,ある疾病の発症の有無,あるいは,発症するときの時期は,各人で大きく異なる.発症の有無は各人にとっては0か1かのデジタルな応答と同義である.また,発症する時期は遺伝子だけでなく環境の影響により大きく異なるが,このことはこれらのパラメーターに対する個人ごとの違いとしてとらえることができるだろう.乳がんをはじめとしたヒトの大規模ゲノム解析の結果から,がんの遺伝子の異常は,PIK3CA,PIK3R1,PTEN,AKT1,MAP3K1,KRAS,BRAF,EGFRなどのEGF受容体あるいはErbBのかかわる膜受容体からのシグナル伝達系に極端にかたよっていることが報告されている44).しかし,おのおのの患者のレベルにおいてどのような遺伝子異常の組合せがあるのか,また,その異常度と予後との関係などについてはいまだ明らかにされていない.このような入力と出力との関係の予測はネットワークのモデリングにより可能になるかもしれない.たとえば,遺伝子の増幅はタンパク質の初期の濃度の上昇,タンパク質の変異は分子間相互作用や酵素-基質間の結合親和性の変化などのパラメーターとして表わすことができる.さらに,これにほかの遺伝子の相互作用やフィードバック制御などのネットワーク構造がくわわると,個体レベルでの発症は非線形になるはずである.このような系では,1遺伝子のわずかな発現量の違いやタンパク質のあいだの親和性の違いが,個体のあいだの決定的な違いを生みだしてもおかしくはない.また一方で,個人ごとに生活習慣などを変えて遺伝子の活性化のパラメーターを制御し発症を未然に防ぐという,個別化予防医学のようなことも可能になるのではないかと想像している.
おわりに
このレビューでは,ヒトゲノムの研究と連動して発展したシグナル伝達系のシステム生物学的な研究について,ここ15年間の動向を駆け足で説明した.筆者が実験を中心に研究を進めていることから実験系の成果をおもにとりあげたが,ここでは説明しなかった理論的な研究も多くある45-48).ここで紹介したネットワーク制御の理論は,シグナル伝達系だけでなく転写因子あるいは細胞間の制御にも応用できる汎用性の高い考え方である.2012年,Mycoplasma菌の全細胞モデルが発表され49),大きな話題になった.まだ,1細菌のモデルではある.しかし,このことに触発された多くのグループは,このさきにヒトの全ゲノムモデルをみすえているのはまちがいないだろう.欧米のシステム生物学の研究の現場ではすでに臨床研究者との連携が急速に進んでおり,いまの時点ではありえないと思うかもしれないが,ヒトゲノムにもとづいた疾患の発症予測モデルが発表される日もそう遠くないような気がしている.ただ,こういった大規模モデルを構築する際に,ただ漫然と大きなモデルを構築するのではなく,なんらかの理論にもとづいたモデルを構築したほうが高い予測精度をもつことが,いままでのシグナル伝達のモデリングの研究の結果からも示唆されている.いずれにしても,システム生物学の強みは“予測”である.多くの実験研究者がモデルやシミュレーションをひとつの実験ツールと考えられるよう,今後,わが国においてもさまざまな研究の連携の進むことが望まれる.
文 献
- Ferrell, J. E. Jr. & Machleder, E. M.: The biochemical basis of an all-or-none cell fate switch in Xenopus oocytes. Science, 280, 895-898 (1998)[PubMed]
- Hoffmann, A., Levchenko, A., Scott, M. L. et al.: The IκB-NF-κB signaling module: temporal control and selective gene activation. Science, 298, 1241-125 (2002)[PubMed]
- Albert, R., Jeong, H. & Barabasi, A. L.: Error and attack tolerance of complex networks. Nature, 406, 378-382 (2000)[PubMed]
- Alon, U.: Network motifs: theory and experimental approaches. Nat. Rev. Genet., 8, 450-461 (2007)[PubMed]
- Heinrich, R., Neel, B. G. & Rapoport, T. A.: Mathematical models of protein kinase signal transduction. Mol. Cell, 9, 957-970 (2002)[PubMed]
- Kholodenko, B. N.: Cell-signalling dynamics in time and space. Nat. Rev. Mol. Cell Biol., 7, 165-176 (2006)[PubMed]
- Guix, M., Faber, A. C., Wang, S. E., et al.: Acquired resistance to EGFR tyrosine kinase inhibitors in cancer cells is mediated by loss of IGF-binding proteins. J. Clin. Invest., 118, 2609-2619 (2008)[PubMed]
- Arteaga, C. L. & Baselga, J.: Tyrosine kinase inhibitors: why does the current process of clinical development not apply to them? Cancer Cell, 5, 525-531 (2004)[PubMed]
- Chen, W. W., Schoeberl, B., Jasper, P. J. et al.: Input-output behavior of ErbB signaling pathways as revealed by a mass action model trained against dynamic data. Mol. Syst. Biol., 5, 239 (2009)[PubMed]
- Sachs, K., Perez, O., Pe'er, D. et al.: Causal protein-signaling networks derived from multiparameter single-cell data. Science, 308, 523-529 (2005)[PubMed]
- Nussinov, R. & Tsai, C. J.: Allostery in disease and in drug discovery. Cell, 153, 293-305 (2013)[PubMed]
- Machida, K., Thompson, C. M., Dierck, K. et al.: High-throughput phosphotyrosine profiling using SH2 domains. Mol. Cell, 26, 899-915 (2007)[PubMed]
- Scott, J. D. & Pawson, T.: Cell signaling in space and time: where proteins come together and when they're apart. Science, 326, 1220-1224 (2009)[PubMed]
- Santos, S. D., Verveer, P. J. & Bastiaens, P. I.: Growth factor-induced MAPK network topology shapes Erk response determining PC-12 cell fate. Nat. Cell Biol., 9, 324-330 (2007)[PubMed]
- McClean, M. N., Mody, A., Broach, J. R. et al.: Cross-talk and decision making in MAP kinase pathways. Nat. Genet., 39, 409-414 (2007)[PubMed]
- Nakakuki, T., Birtwistle, M. R., Saeki, Y. et al.: Ligand-specific c-Fos expression emerges from the spatiotemporal control of ErbB network dynamics. Cell, 141, 884-896 (2010)[PubMed]
- Markevich, N. I., Hoek, J. B. & Kholodenko, B. N.: Signaling switches and bistability arising from multisite phosphorylation in protein kinase cascades. J. Cell Biol., 164, 353-359 (2004)[PubMed]
- Roberts, P. J. & Der, C. J.: Targeting the Raf-MEK-ERK mitogen-activated protein kinase cascade for the treatment of cancer. Oncogene, 26, 3291-310 (2007)[PubMed]
- Kholodenko, B. N.: Negative feedback and ultrasensitivity can bring about oscillations in the mitogen-activated protein kinase cascades. Eur. J. Biochem., 267, 1583-1588 (2000)[PubMed]
- Shankaran, H., Ippolito, D. L., Chrisler, W. B. et al.: Rapid and sustained nuclear-cytoplasmic ERK oscillations induced by epidermal growth factor. Mol. Syst. Biol., 5, 332 (2009)[PubMed]
- Hiroshima, M., Saeki, Y., Okada-Hatakeyama, M. et al.: Dynamically varying interactions between heregulin and ErbB proteins detected by single-molecule analysis in living cells. Proc. Natl. Acad. Sci. USA, 109, 13984-13989 (2012)[PubMed]
- Sasagawa, S., Ozaki, Y., Fujita, K. et al.: Prediction and validation of the distinct dynamics of transient and sustained ERK activation. Nat. Cell Biol., 7, 365-373 (2005)[PubMed]
- Nagashima, T., Shimodaira, H., Ide, K. et al.: Quantitative transcriptional control of ErbB receptor signaling undergoes graded to biphasic response for cell differentiation. J. Biol. Chem., 282, 4045-4056 (2007)[PubMed]
- Murphy, L. O., Smith, S., Chen, R. H. et al.: Molecular interpretation of ERK signal duration by immediate early gene products. Nat. Cell Biol., 4, 556-564 (2002)[PubMed]
- Mackeigan, J. P., Murphy, L. O., Dimitri, C. A. et al.: Graded mitogen-activated protein kinase activity precedes switch-like c-Fos induction in mammalian cells. Mol. Cell. Biol., 25, 4676-4682 (2005)[PubMed]
- Bonizzi, G. & Karin, M.: The two NF-κB activation pathways and their role in innate and adaptive immunity. Trends Immunol., 25, 280-288 (2008)[PubMed]
- Oeckinghaus, A., Hayden, M. S., Ghosh, S.: Crosstalk in NF-κB signaling pathways. Nat. Immunol., 12, 695-708 (2011)[PubMed]
- Tay, S., Hughey, J. J., Lee, T. K. et al.: Single-cell NF-κB dynamics reveal digital activation and analogue information processing. Nature, 466, 267-271 (2010)[PubMed]
- Nelson, D. E., Ihekwaba, A. E., Elliott, M. et al.: Oscillations in NF-κB signaling control the dynamics of gene expression. Science, 306, 704-708 (2004)[PubMed]
- Shinohara, H. & Kurosaki, T.: Comprehending the complex connection between PKCβ, TAK1, and IKK in BCR signaling. Immunol. Rev., 232, 300-318 (2009)[PubMed]
- Polley, S., Huang, D. B., Hauenstein, A. V. et al.: A structural basis for IκB kinase 2 activation via oligomerization-dependent trans auto-phosphorylation. PLoS Biol., 11, e1001581 (2013)[PubMed]
- Avraham, R. & Yarden, Y.: Feedback regulation of EGFR signalling: decision making by early and delayed loops. Nat. Rev. Mol. Cell Biol., 12, 104-117 (2011)[PubMed]
- Novak, B. & Tyson, J. J.: Design principles of biochemical oscillators. Nat. Rev. Mol. Cell Biol., 9, 981-991 (2008)[PubMed]
- Aoki, K., Kumagai, Y., Sakurai, A. et al.: Stochastic ERK activation induced by noise and cell-to-cell propagation regulates cell density-dependent proliferation. Mol. Cell, 52, 529-540 (2013)[PubMed] [新着論文レビュー]
- Lim, W. A.: Designing customized cell signalling circuits. Nat. Rev. Mol. Cell Biol., 11, 393-403 (2010)[PubMed]
- O'Shaughnessy, E. C., Palani, S., Collins, J. J. et al.: Tunable signal processing in synthetic MAP kinase cascades. Cell, 144, 119-131 (2011)[PubMed]
- Kiyatkin, A., Aksamitiene, E., Markevich, N. I. et al.: Scaffolding protein Grb2-associated binder 1 sustains epidermal growth factor-induced mitogenic and survival signaling by multiple positive feedback loops. J. Biol. Chem., 281, 19925-19938 (2006)[PubMed]
- Qiao, Q., Yang, C., Zheng, C. et al.: Structural architecture of the CARMA1/Bcl10/MALT1 signalosome: nucleation-induced filamentous assembly. Mol. Cell, 51, 766-779 (2013)[PubMed]
- Good, M. C., Zalatan, J. G., Lim, W. A.: Scaffold proteins: hubs for controlling the flow of cellular information. Science, 332, 680-686 (2011)[PubMed]
- Levchenko, A,. Bruck, J., Sternberg, P. W.: Scaffold proteins may biphasically affect the levels of mitogen-activated protein kinase signaling and reduce its threshold properties. Proc. Natl. Acad. Sci. USA, 97, 5818-5823 (2000)[PubMed]
- Santos, S. D., Wollman, R., Meyer, T. et al.: Spatial positive feedback at the onset of mitosis. Cell, 149, 1500-1513 (2012)[PubMed]
- Fu, W., Ergun, A., Lu, T. at al.: A multiply redundant genetic switch 'locks in' the transcriptional signature of regulatory T cells. Nat. Immunol., 13, 972-980 (2012)[PubMed]
- Pleasance, E. D., Cheetham, R. K., Stephens, P. J. et al.: A comprehensive catalogue of somatic mutations from a human cancer genome. Nature, 463, 191-196 (2010)[PubMed]
- Cancer Genome Atlas Network: Comprehensive molecular portraits of human breast tumours. Nature, 490, 61-70 (2012)[PubMed]
- Tsai, T. Y., Choi, Y. S., Ma, W. et al.: Robust, tunable biological oscillations from interlinked positive and negative feedback loops. Science, 321, 126-129 (2008)[PubMed]
- Cheong, R., Rhee, A., Wang, C. J. et al.: Information transduction capacity of noisy biochemical signaling networks. Science, 334, 354-358 (2011)[PubMed]
- Thattai, M. & van Oudenaarden, A.: Intrinsic noise in gene regulatory networks. Proc. Natl. Acad. Sci. USA, 98, 8614-8619 (2001)[PubMed]
- Kholodenko, B. N., Kiyatkin, A., Bruggeman, F. J. et al.: Untangling the wires: a strategy to trace functional interactions in signaling and gene networks. Proc. Natl. Acad. Sci. USA, 99, 12841-12846 (2002)[PubMed]
- Karr, J. R., Sanghvi, J. C., Macklin, D. N. et al.: A whole-cell computational model predicts phenotype from genotype. Cell, 150, 389-401 (2012)[PubMed]
参考図書
江口至洋: 細胞のシステム生物学. 共立出版 (2008)
Uri Alon (著), 倉田博之, 宮野 悟 (訳): システム生物学入門: 生物回路の設計原理. 共立出版 (2008)
Alberts, B., Johnson, A., Lewis, J. et al: Molecular Biology of the Cell, 5th Ed, Chapter 7 & Chapter 15. Garland Science, New York (2008)
Voit, E. O: A First Course in Systems Biology. Garland Science, New York (2012)
Uri Alon’s Systems Biology Lecture-YouTube. http://www.youtube.com/watch?v=Z__BHVFP0Lk
著者プロフィール
略歴:2003年 東京農工大学大学院連合農学研究科にて博士号取得,理化学研究所ゲノム科学総合研究センター 研究員,上級研究員,チームリーダーを経て,2009年より同 免疫アレルギー科学総合研究センター(現 統合生命医科学研究センター)チームリーダー.
研究テーマ:シグナル転写ネットワークのシステム生物学.
抱負:いまは,細胞に含まれる異なる階層をつなげること.将来は,また創薬に貢献したい.
© 2014 岡田 眞里子 Licensed under CC 表示 2.1 日本