野外の環境における生物の環境応答の理解にむけて:トランスクリプトームデータと気象データの統合
2014/07/22
永野 惇・工藤 洋
(京都大学生態学研究センター)
email:永野 惇
領域融合レビュー, 3, e009 (2014) DOI: 10.7875/leading.author.3.e009
Atsushi J. Nagano & Hiroshi Kudoh: Integration of transcriptomics and meteorology.

分子生物学では,通常,実験は均質な実験室の環境において行われる.一方,生物の本来の生育場所は複雑に変動する野外の環境である.そのような複雑な環境における遺伝子の発現を解析するための方法として,気象データを組み合わせた統計モデリングがある.統計モデリングによる解析により,ハクサンハタザオの花成制御遺伝子であるAhgFLC遺伝子の発現は過去6週間の気温により説明できることがわかった.また,イネのトランスクリプトームの変動は概日時計と気温により大部分が説明できることがわかった.気温や日長など環境情報から季節を知るために必要となるしくみをシグナル-ノイズ比の観点から検討したところ,それらは統計モデリングにより得られた野外の環境における遺伝子の発現にみられた特徴と一致していた.さらに,その特徴は生物気象学における研究の多くの結果とも一致していた.野外を中心とする生態学や生物気象学などの成果と,実験室を中心とする分子生物学の成果の統合的な理解のため,トランスクリプトームデータと気象データの統計モデリングは重要である.
いわゆる分子生物学では,通常,実験は均質な実験室の環境において,遺伝的に均一なモデル生物を用いて行われる.問題をなるべく単純化し,明瞭な結果が得られるように実験条件を整える.慎重に制御された遺伝的な背景および環境を用いて,研究の対象としている要因以外を極力そろえて実験を行う.その結果,これまでに生物のさまざまな形質の背後にある分子機構が明らかにされてきた.
一方,いうまでもなく,生物の本来の生育場所は実験室ではなく野外である.生物の適応形質は自然生息地における自然選択によりかたちづくられたものであるので,それらの機能は自然生育地の環境において評価されるべきであろう.また,作物はその大半が野外の圃場において栽培されるため,作物の生産性の向上を望むのであれば,実際に野外の圃場の環境において起こっていることを理解する必要があるだろう.このように,野外における生物の実態を理解したいという要求はごく自然なものである.しかしながら,この要求に対し,実験室における分子生物学の発想により応えようとすると困難につきあたる.そのひとつが,野外の環境の複雑さである.さきに述べたように,実験室では気温や光を一定にするなど,比較的単純な条件に制御して実験を行う.一方,野外では気温ひとつとっても刻一刻と激しく変動する.もちろん,変動するのは気温だけでなく,光条件や湿度などが複雑に関係しあいながら変動している.
このレビューでは,このように複雑な野外の環境における生物の環境応答を分子生物学的な視点から取り扱った研究を紹介する.まず,中心となる方法論として,野外の環境における遺伝子の発現と気象データとを組み合わせた統計モデリングについて解説する.つぎに,実際の研究事例として,野生植物であるハクサンハタザオ(Arabidopsis halleri subsp. gemmifera)についての研究,および,主要な作物であるイネ(Oryza sativa)についての研究を紹介する.さらに,野外の環境において研究を行うことにより可能になる特徴的な議論の例として,環境からのシグナルの検出とノイズに対する頑健性との関係をとりあげる.最後に,このような研究が分子生物学と生物気象学の蓄積を統合的に理解することに役だつことについて説明する.
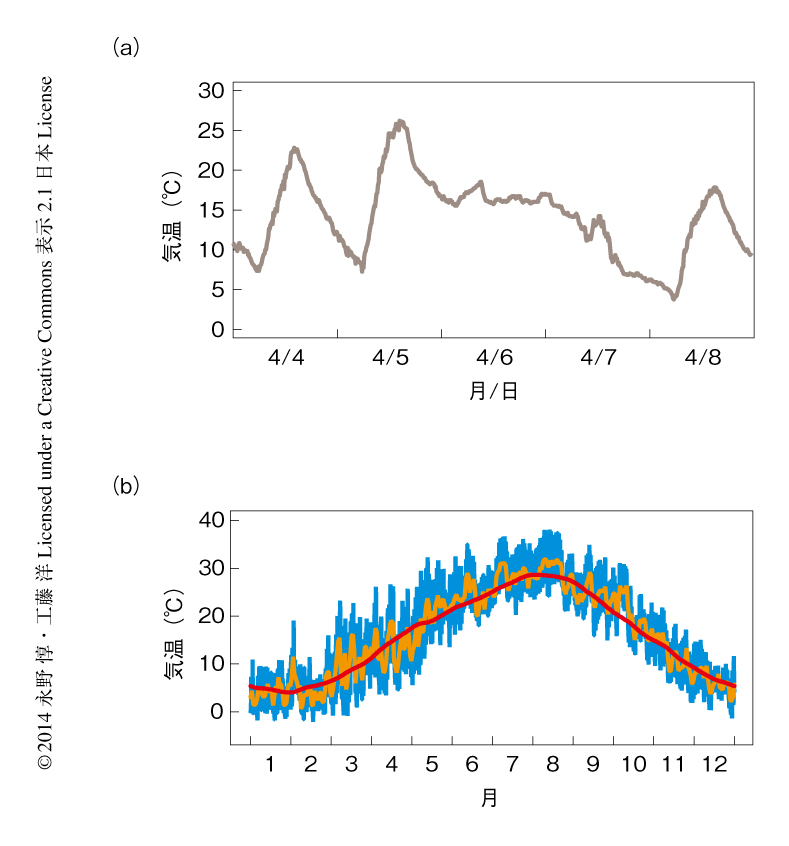
野外における環境変動の複雑さを京都市の気温を例としてみる.まず,日周変動がある(図1a).晴れた日には昼の気温が高く夜の気温は低い.これは雨の日には必ずしもあてはまらない.季節にもよるが,昼夜の気温差は10℃以上になることも多い.年周変動のようなより長いスパンの変動や(図1b),天候の変化などによるより短いスパンの変動も存在する.このように複雑に変動する野外の環境への生物の応答を,単純な環境に制御された実験室において明らかにすることは容易ではない.たとえば,高温ストレスの研究でしばしば使われる20℃で一定の条件から35℃で一定の条件へと一気に変化する状況は,野外では普通は起こらない.その代わり,気温は細かい変動をくり返しながら徐々に上昇する.気温の上昇が数分間で起こる場合と数時間かけて起こる場合とで,生物の応答は根本的に異なっているかもしれない.そう考えると,ひとくちに高温ストレスといっても条件設定の可能性は無数にあることがわかる.無数にある可能性のうち,どこからはじめるべきであろうか.その答えのひとつが,実際の野外の環境において生物の応答を調べることである.
では,そのために何を測定するべきだろうか.トランスクリプトームはもっとも魅力的な測定対象のひとつであろう.DNAマイクロアレイ法やRNA-Seq法によるトランスクリプトミクスは網羅性および定量性が高く,現状において生物システムのもっとも包括的な測定手法である.その網羅性ゆえに,トランスクリプトームのデータは直接的あるいは間接的にさまざまな生物現象の情報を内包すると期待できる.たとえば,これまでに分子生物学などにより明らかにされたさまざまな知見が遺伝子にひもづけられるかたちでデータベースに蓄積されており,それらの知見を用いることにより多様な生理状態について考察することが可能になる.また,遺伝子の発現は数分間から数時間で100倍以上も変動することがある1,2).この短時間で大きく変動しうる性質も,野外の環境における生物の環境応答を知るための測定対象として適している点であろう.また,野外の環境における栽培あるいは研究は,実験室で行う場合よりはるかに労力がかかる.くわえて,環境条件は自然まかせになるため時期が限られ,多くの場合,年に1回きりのチャンスにおいて可能なかぎりのことをすることになる.多数の項目をいちどに測定できるトランスクリプトミクスは,労力の面でも野外における研究に適している.
数は多くないものの,10年以上まえから野外の環境における遺伝子の発現の研究ははじまっていた3-6).近年では,イネ7-9),シロイヌナズナ10)(Arabidopsis thaliana),トウモロコシ11)(Zea mays),ビッグブルーステム12)(Andropogon gerardii),ヨーロッパヤマナラシ13)(Populus tremula),フタバガキ科の熱帯性植物14)(Shorea beccariana)など,モデル植物や作物を中心として野外の環境においてトランスクリプトームのデータが取得されるようになってきている.しかしながら,これらのうち気象データなどを用いて野外における環境変動との関係を解析している研究はわずかである.特定の現象の解析を目的としてデザインされた実験においては,気象条件の変動は単に結果を乱す要因と考えられることが多い.シロイヌナズナ10),ビッグブルーステム12),ヨーロッパヤマナラシ13) を用いた研究においては,気象データとの関係を解析している.直観的な解釈のむずかしい結果ではあるが,いずれの研究においても気温の影響が検出されるなど,一定の成果が得られている.しかしながら,サンプリングの時点数(日数)が少ないことや,データ解析手法における制約などから,それ以上にふみこんだ議論はできていない.
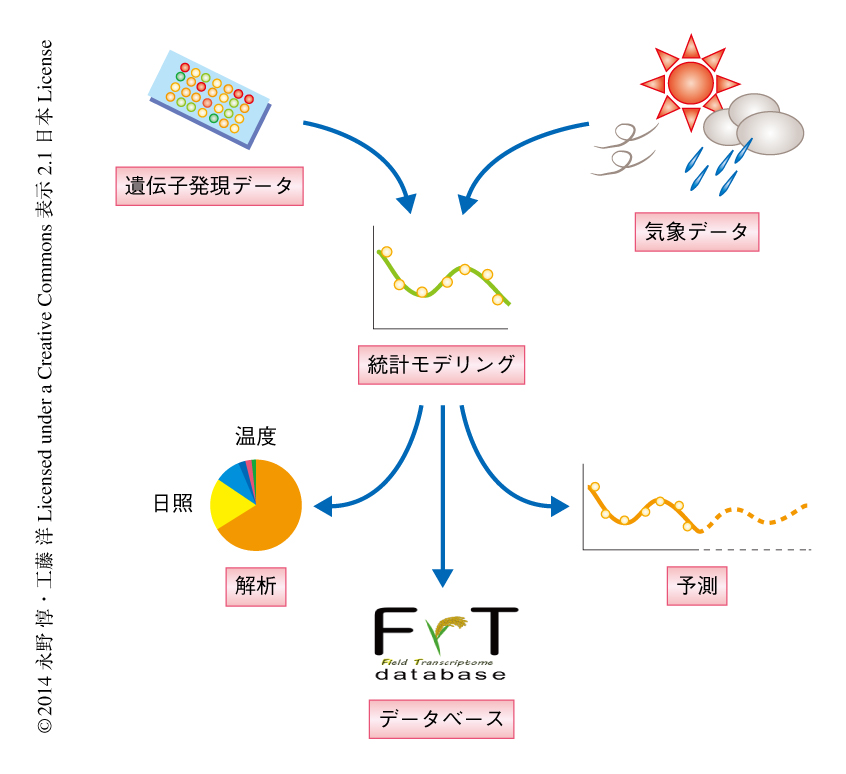
仮に,十分な量の気象データと野外におけるトランスクリプトームのデータが利用できるとするなら,どのように解析すれば変動する野外の環境とトランスクリプトームとの関係を明らかにできるであろうか.解析手法や切り口はいくつもあり,それぞれに一長一短がある.そこで,最終的に得たい情報にあわせた手法を選ぶことが重要になる.ここでは,統計モデリングとよばれるアプローチを採用する(図2).本来,統計モデリングという用語はかなり広い意味をもつが,ここでは,トランスクリプトームのデータ解析においてよく使われるクラスタリングと対比して,解析者が説明変数およびモデルをあたえる回帰をさすことにする.統計モデリングを用いる利点は,研究者が解釈したい切り口を仮定としてあたえて解析できるため,結果を解釈しやすい点である.統計モデリングでは,遺伝子の発現量のような説明される側のデータと,気温やサンプリングの時刻など説明する側のデータとの関係を数式で表わす必要がある.数式というとハードルが高いと感じられるかもしれないが,目的に応じて単純なものでもよい.たとえば,概日時計に由来するサンプリングの時刻に依存した発現変動として,単純な正弦関数を用いるなどである.また,生態学や作物学,生物気象学では,環境要因と開花時期,収量との関係など,これまでにさまざまなモデルが提案されている.それらの蓄積をうまく活用して解析を行うこともできる.
例として,ハクサンハタザオの自然集団におけるFLC遺伝子の発現の季節変動の解析について紹介する15).ハクサンハタザオはもっともよく研究されているモデル植物であるシロイヌナズナに近縁な種のひとつで,わが国に自生する.花成制御遺伝子であるFLC遺伝子はMADSボックス型の転写因子をコードしており,シロイヌナズナやその近縁の種において保存されている15-18).一年草および多年草のいずれにおいても,温度に依存的な花成の時期を制御する遺伝子としてはたらいていることが実験室において確認されている19).長期の低温によりFLC遺伝子の発現抑制が起こり,そののち,FLC遺伝子により発現の抑制されていたFT遺伝子の発現亢進が起こることで花成が導かれる.一年草であるシロイヌナズナのFLC遺伝子の発現抑制は低温処理の長さに対し量的に起こるが20),温度の上昇したのちの発現抑制は一生をおえるまでずっと維持される21).すなわち,この場合はFLC遺伝子の発現の季節的な制御は,葉をつくる時期である栄養成長相から花を咲かせる時期である繁殖成長相への移行をもって打ち切られていると考えられる.シロイヌナズナやハクサンハタザオに近縁の多年草であるArabis alpinaのもつFLC遺伝子のオーソログであるPEP1遺伝子の発現も,シロイヌナズナの場合と同様に継続的な低温により抑制され,この抑制はPEP1遺伝子座におけるヒストンH3のLys27のトリメチル化のレベルの上昇と相関している17).興味深いことに,PEP1遺伝子の発現の抑制は低温処理ののち温度を上昇させた場合には安定に維持されない.多年草はその生活史において,気温の季節変動に対しくり返し応答する必要がある.PEP1遺伝子の発現制御はそのための分子基盤の一端を担うと示唆される17).では,自然環境において,本当に多年草のもつFLC遺伝子のオーソログが気温の季節変動を認識するためのしくみとしてはたらいているのだろうか.
ハクサンハタザオの自然個体群を用いてFLC遺伝子のオーソログ(AhgFLC遺伝子)の発現の季節変動が調べられた15).自然生育地において自生している植物を用いたため,それぞれの個体は気温の長期にわたる季節変動だけでなく,大きな日周変動やより短期の変動にもさらされていた.2年間にわたり,週1回12時にそれぞれ6個体から葉をサンプリングし,定量PCR法によりAhgFLC遺伝子の発現量が測定された.AhgFLC遺伝子の発現は明瞭な季節変動を示した.発現は晩秋から冬にかけて徐々に低下していき,春になるとふたたび上昇しはじめ,夏前には最大値のレベルにまでもどった.このハクサンハタザオの自然集団では,2月末から3月初旬に抽台,4月末ごろに開花,5月から6月にかけてリバージョン(多年草で観察される,繁殖成長相から栄養成長相へと逆戻りする現象)が起こっていた.これらのイベントはAhgFLC遺伝子の発現の変動と密接に関係していると考えられた.
AhgFLC遺伝子の発現はノイズの大きな気温の変動から季節のパターンをうまく抽出していた(図3a).また,気温の季節パターンとAhgFLC遺伝子の発現の季節パターンとのあいだには遅れがみられた.この遅れのために,気温は上昇しているがAhgFLC遺伝子の発現の低い期間が生じ,そのタイミングで花成が起こっていた.つぎに,このようなAhgFLC遺伝子の発現パターンが気温によりどのように説明されるか,積算温度モデルを用いて解析された.積算温度は,ある一定の期間においてある閾値をうわまわった(あるいは,したまわった)気温の積算と定義される(図3b).積算温度は生態学や作物学において気温から開花日を予想するモデルなどに広く用いられている.実験室における解析から,シロイヌナズナのFLC遺伝子の発現は低温に影響されることが知られていたので,AhgFLC遺伝子の発現データの解析の際には閾値をしたまわった気温の積算が用いられた.解析の結果,2年間にわたるAhgFLC遺伝子の発現の変動をもっともよく説明したのは,過去42日間(6週間)の10.5℃以下の気温の積算であった15).驚くべきことに,このモデルは2年間の発現変動のうちの83%を説明することができた.閾値と比べ参照する過去の期間の長さはより重要で,6週間より長くても短くてもモデルの説明力はいちじるしく低下した.さらに,このモデルは培養装置を用いて人工的に気温を変化させた場合の応答も高い精度で予測することができた.
この研究の重要性のひとつとして強調したいのは,それが自然環境において遺伝的に不均一な複数の個体を用いて行われた点である.自然生育地では,しばしば気温以外の要因のほうがより大きく変動している.実際に,この研究に用いられたサンプルは,近傍の川の氾濫や積雪による被覆,および,乾燥ストレスにさらされていた.にもかかわらず,AhgFLC遺伝子の発現の変動はその大部分が過去の気温のみで説明することができた.この結果は,AhgFLC遺伝子の発現の制御機構は気温の季節変動を検出できるだけでなく,遺伝的な背景の不均一性や複雑な自然環境におけるノイズに対し,十分な頑健性をもつことを示した.本来,ハクサンハタザオは遺伝的な背景や微小な環境の異なる複数の個体のあいだで同じタイミングで開花する必要がある.この頑健性は,そのような開花の同調に重要な役割をはたしているのかもしれない.
ハクサンハタザオのAhgFLC遺伝子の例では,低温に応答することが既知であった1つの遺伝子のみを対象としていた.トランスクリプトームを考えると,当然,気温だけではなく光や降水などほかの環境条件が遺伝子の発現に影響する可能性もある.また,環境条件のほかに,日齢や体内時計なども遺伝子の発現に大きく影響するかもしれない.それらの影響の相対的な強さは遺伝子ごとに異なっていると考えられる.そこで,より包括的に,野外の環境におけるトランスクリプトームの変動に対しどのような要因が支配的に影響するのか,イネにおいて調べた例について紹介する22).
茨城県つくば市の実験圃場において,6月から9月にかけて500サンプル以上の葉が収集された.遺伝的な背景および葉の発生ステージの影響を極力うけないよう,系統としてはおもに日本晴を用い,もっとも若い完全展開葉に固定してサンプリングを行った.一方,時刻および気象の条件に関しては,豪雨が観測された日を含む9セットの2時間おき48時間のサンプルなど,さまざまなサンプルが含まれていた.これらのサンプルを用いてDNAマイクロアレイ解析が行われた.また,同時に解析に用いる気象データとして,圃場の近くに位置する気象庁の地上気象観測地点のデータが用いられた.全国に点在する地上気象観測地点では,気温,相対湿度,気圧,風向および風速,全天日射量,降水量などについて,1分ごとのデータが蓄積されている.統計モデリングにあたっては,日齢,体内時計,環境要因を考慮したモデルが用いられた.環境要因からの影響の部分については,さきに説明したような積算温度モデルを拡張したものが用いられた.個々の遺伝子がどのような環境要因の影響をうけるかが事前にわからないため,積算“温度”だけではなく,相対湿度,気圧,風速,全天日射量,降水量も含めた6通りの気象データから,遺伝子の発現をもっともよく説明するデータをおのおのの遺伝子について選ぶというアプローチがとられた.さらに,一日のうち特定の時間帯のみに環境応答を示す現象(ゲート効果)もモデルに組み込まれた.結果的に,遺伝子ごとに最大で17のパラメーターをもつモデルになった.このモデルと,約500サンプル分のトランスクリプトームのデータ,半年分の気象データを用いて,パラメーターを最適化する計算を行った.その結果,葉で発現のみられた遺伝子のうち96.7%について予測に利用することの可能なモデルパラメーターが得られた.このモデルが妥当な予測を行えることは,パラメーターの最適化に用いたものとは独立の野外サンプルのデータ,および,実験室において温度条件および光条件を変えた際のデータを用いて検証された.なお,個々の遺伝子の結果は,データベースFiT-DBとして公開されている(URL:http://fitdb.dna.affrc.go.jp/).
トランスクリプトームの全体にわたり結果のパラメーターを集計することにより,野外環境においてどのような要因が支配的かを知ることができた.たとえば,概日時計の影響がもっとも大きく,そのつぎに気温や光などへの環境応答が大きく,植物の日齢はそれほど影響しないことがわかった.また,環境応答を示す遺伝子の約8割は気温により説明されたことから,環境応答においては気温の影響がとくに大きいことがわかった.このほかにも,さまざまなことを結果のパラメーターから知ることができた.たとえば,環境応答を示す遺伝子の約3割でゲート効果のみられること明らかになった.また,これだけの量のデータを用い,かつ,統計モデリングを行ったことにより議論できるようになったことに,個体間の発現量のばらつきの大きさがある.概日時計や環境応答などの決定論的な変動では説明できない個体間の確率的なばらつきの大きさが遺伝子ごとに異なっており,リボソームやヒストンなど基本的な細胞機能にかかわる遺伝子では小さく,病害応答などの外的な刺激にかかわる遺伝子では大きいことが見い出された.さらに,得られたモデルを用いれば,任意の条件での予測が可能となる.ただし,モデルの最適化に用いたデータのとられた地点の野外環境から大きく外れた条件では予測精度が低下すると考えられるため,その利用には慎重な検討が必要である.
野外の環境において研究を行うことにより可能となる特徴的な議論として,環境からのシグナルの検出とノイズに対する頑健性との関係をとりあげる.シグナルとノイズの相対的な大きさは一般にシグナル/ノイズ比(S/N比)として知られている.高いシグナル/ノイズ比はシグナルの大きさがノイズの大きさより十分に大きいことを意味しており,その場合,目的とするシグナルは明瞭に検出できる.
まず例として,イネの光に対する応答を考える.光に対する応答を考慮したほうがよい遺伝子について,光に対する応答の閾値のパラメーターを集計してみると,そのほとんどがごく低い値になっていた22).つまり,真っ暗か否かの2値的な応答をしていることになる.このことは,生化学的にはファイトクロムなどの光受容体の感度がきわめて高いことに対応していると思われる23).なぜ,光に対しこのように過剰なまでに高い感度で応答するのだろうか.そのひとつの解釈が,環境からのシグナルの検出とノイズに対する頑健性の観点から得られる.シグナルとノイズといった場合,何がシグナルで何がノイズなのかは,当然,対象により異なる.ここで注目するのは日長の変化である.イネは日長の変化に鋭敏に応答し花成を行う24).そのため,日長の変化を正確に測定しシグナルとして用いることは重要であろう.このとき,日々の天候や周囲の植生などに起因する日長測定の誤差がノイズとみなされる.もし仮に,より感度の低い光受容体を用いて日長を測定すると,日々の天候により主観的な日の長さが大きく変動してしまうだろう.つまり,朝夕に雲が多く暗い日には日が短く感じられてしまう.逆に,非常に高感度な光受容体を用いて日長を測定すれば,曇りであろうがほとんど影響をうけずに日長を測定することができる.実際に,光に対するさまざまな閾値でのシグナル/ノイズ比を計算すると,閾値が低いほどシグナル/ノイズ比は高い値となる22).以上の議論から,光に対する高感度での応答は日長に対し正確に応答するために有用だと考えられる.じつは,この結論は生物の光周性において古くから知られている,光に対する閾値は低く光周性は天候によらないという事実と同じものである25).
気温の季節シグナルとしての特徴から,気温をもとに季節を知るためのしくみに必要な特徴を明らかにする.赤道の付近を除き,日の出から日の入りまでの時間(日長)は規則的な年周期の変動を示し,その変動は緯度から予測が可能である25,26).この変動はそもそも地球の地軸が23.4℃傾いていることに起因する.日長は夏至で最長,冬至で最短になる(北半球では,夏至はだいたい6月22日,冬至はだいたい12月22日).また,日射量の年周期の変動は季節をかたちづくる27,28).日長と気温の年間の変化は,温帯域より高緯度では植物の季節応答の主要な決定因子として十分な大きさがある29).ここで例として,京都市の気温のデータから検討する(図1).ある1地点のデータを解析した結果ではあるものの,以下の大半の議論は地球上のほかの任意の地点,少なくとも,温帯域の地点において成り立つ27,28).気温は明瞭な季節変動を示し,そのピークは日長の変化から1カ月から1.5カ月の遅れをもつ27,30).気温の季節変動は数カ月のスケールのパターンである(図1b).季節のパターンがあるのと同時に,気温はさまざまな時間スケールで激しく変動している.日平均気温の変化をみると,日々の変動やより長期の変動のほうが大きいことがわかる(図1b).たとえば,春は長期的には徐々に気温が上がっていく傾向があるものの,前日より涼しい日や,前の週より涼しい週がくることも珍しいことではない.さらに,気温の日中変動をみると,1日のなかでの気温の変化の幅は,しばしば1~2カ月以上の期間の気温の変化の幅に相当することがわかる(図1b).すなわち,気温は日周変動,日間変動,さらには,より短期あるいは長期の変動を含み,季節シグナルの抽出においてはそれらが深刻なノイズになる.すなわち,気温の季節変動はそのままではシグナル/ノイズ比の低い情報であることがわかる.一般に,シグナル/ノイズ比の低い情報を用いる場合,より長い期間の測定(より多くの測定)が信頼性の高いシグナルの検出のため必要となる.
では,どの程度の長さの測定が必要なのだろうか.ここでは議論を簡単にするため,移動平均を用いて検討する.たとえば,3日間の気温の移動平均であれば,過去3日分の気温の平均値がその時点での値になる.京都市の気温のデータを用いて,移動平均期間を1日間,7日間,42日間,270日間としたときの移動平均を計算した(図4).移動平均による平滑化により短期のノイズは取り除かれ,滑らかな季節変動のパターンが得られた.移動平均期間を長くとるにしたがってノイズはめだたなくなっていき,季節シグナルがはっきりとしてくることがわかる.移動平均期間が42日間の場合にはノイズはほとんど無視できる程度になっている.より長い移動平均期間をとることによりシグナル/ノイズ比はさらに向上するものの,別の2つの影響も現われる.ひとつはシグナルの絶対的な大きさの減少であり,もうひとつは季節シグナルの遅れである.このため,実際には参照する過去の期間が長すぎるのもよくない.以上の議論から,過去4~6週間程度の気温を参照するしくみがあれば,短期のノイズを無視し季節シグナルをとらえることができると考えられる.
この“過去4~6週間程度の気温を参照するしくみ”は,気温の気象データのみから推定されたものであった.一方,さきに述べたハクサンハタザオにおけるAhgFLC遺伝子の発現の変動においては,気温を参照する過去の期間は6週間であった.興味深いことに,移動平均と積算との違いはあるが,両者が気温の情報を利用する期間はよく一致している.つまり,AhgFLC遺伝子の発現の制御は,自然環境におけるノイズの大きい気温の情報から効率よく季節のシグナルを抽出するためのしくみとして最適化されていると考えられる.
この6週間という長い期間の気温を積算していくための分子機構の一端が,実験室におけるシロイヌナズナのFLC遺伝子の詳細な研究により明らかにされている.4℃などで一定にする低温処理により,2~3週間程度でFLC遺伝子の発現は最低のレベルまで抑制される31).しかしながら,FLC遺伝子の発現抑制の状態が完全に確立し,低温処理が終了したのちまで発現抑制を維持させるためには,4~8週間の低温処理が必要である20,32).FLC遺伝子の抑制が完全に確立した状態では,FLC遺伝子を含むゲノム領域がヒストン修飾を介してヘテロクロマチン化され,それが遺伝子の発現抑制の記憶としてはたらく33-36).FLC遺伝子の発現抑制の完全な確立には低温の期間の長さにしたがい,非コードRNAであるCOOLAIR 31) やCOLDAIR,Polycomb抑制複合体2を構成するタンパク質としてVRN2,SWN,FIE,MSI1 34),PHDファミリータンパク質としてVIN3,VRN5,VIL2 34,37-39) などがはたらく,多段階の過程により達成される40).これらの分子機構が実際の自然環境において低温の記憶としてはたらいているのか,また,さきに述べたハクサンハタザオのような多年草ではどのように制御されているのかについては,今後の研究が待たれる.
ハクサンハタザオにおいてみられたような気温の記憶と花成の制御との関係は,どのくらい一般的なのだろうか.さまざまなノイズを含む気温の情報から効率的に季節シグナルを抽出するしくみについての議論は,特定の種あるいは地域に依存したものではないので幅広く成り立つ可能性がある.生物気象学では,気温などの環境条件と開花など生物の季節応答との関係が長年にわたり調べられてきた.過去数十年ものあいだ,初開花日が記録されデータの解析が行われてきたのである41-43).初開花日は年にいちどしかないため,環境要因がどのように開花日の年ごとの変動に影響するかを統計的に解析するためには,20年分かそれ以上のデータが必要になる.実際の解析では,年ごとの気温の変動を反映する指標と初開花日とのあいだの相関を調べる.生物の季節現象に対する気候変動の影響はこれまでもしばしば解析されており,地球の温暖化は春の開花や展葉を有意に早めていることが報告されている44-47).
ここでみなおしたいのは,過去数週間から数カ月間の環境要因のデータにもとづいて初開花日を予測しようとする研究である.これらの研究におけるおもな問題設定は,どの環境要因のどのくらいの期間の過去の値が初開花日の変動に影響するのかということである.たとえば,英国中部における36年間(1954年~1989年)にわたる243種の顕花植物の初開花日が調べられている48).初開花日には温度が大きく影響することが見い出され,243種のうち219種において過去数カ月の気温から初開花日のばらつきの大半を説明することができた.早期(1~4月)に開花する種の初開花日は過去1~2カ月の気温の影響の強いものが多く,夏季(5月以降)に開花する種では最大で過去4カ月の気温が重要であった.また,イタリア中部における57種の顕花植物について,過去の気温と降水の情報を用いた23年間(1960年~1982年)の初開花日のモデリングが成功している49).2月から5月に開花する種では過去20~38日間の環境の影響を考慮することにより初開花日をもっともよく説明でき,6月から9月に開花する種では過去41~83日間(多くは,60日以下)の環境を考慮することにより初開花日をもっともよく説明することができた.初開花日をモデリングしたほかの研究においても,その多くで過去1~2カ月間の気温がもっとも重要な説明変数であった50-53).
種や地域による違いはあるものの,おおむね過去3~10週間の気温が年ごとの初開花日のばらつきを説明する効果が大きかった.初開花日に影響する過去の気温の期間は,温度に依存的な発生の過程の期間を反映しているというのが伝統的な解釈のひとつである.しかしながら,さきに述べたように,ノイズの多い自然環境において季節シグナルを抽出するという観点からみたとき,参照すべき過去の期間と生物気象学においてこれまで報告されてきた期間との一致が見い出される.このことから,年ごとの開花日のばらつきを,単に発生の過程にかかる時間が気温により受動的に変化した結果だと無条件にみなすべきではないと考えられる.自然生育地における大きなノイズを含む気温の変動のもとで花成の時期を制御するために進化した,より積極的な応答が含まれているのかもしれない.
遺伝子の発現と気象データの統計モデリングを切り口として,野外の環境における生物の環境応答を考える試みを紹介した.自然生育地や圃場といった,より現実的な条件における解析と解釈は実用において重要であるだけでなく,実験室における分子生物学の研究にも新たな視点を提供するだろう54).ここまで述べてきたような,シグナル/ノイズ比に注目した議論はそのひとつの例である.ほかにもたとえば,複数のストレスが同時にかかる状況やストレスの強度が時間的に変化する状況において何が起こるのかを,実験室における分子生物学の研究によりていねいに解析することは価値があるだろう.その際,無限にある条件設定の可能性から注目すべきものを絞り込むため,野外の環境における生物の応答を解析した結果が役にたつと考えられる.また,生態学や生物気象学など野外を中心に研究が進められてきた分野の知見と,実験室における分子生物学により蓄積された知見の統合的な理解を進めるうえでも,トランスクリプトームのデータと気象データの統計モデリングは重要になるだろう(図5).
略歴:2009年 京都大学大学院理学研究科 修了,同年 農業生物資源研究所 特別研究員を経て,2011年より京都大学生態学研究センター 特別研究員.2012年より科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:複雑な環境における生物のシステム動態の理解,予測,制御.
工藤 洋(Hiroshi Kudoh)
京都大学生態学研究センター 教授.
© 2014 永野 惇・工藤 洋 Licensed under CC 表示 2.1 日本
(京都大学生態学研究センター)
email:永野 惇
領域融合レビュー, 3, e009 (2014) DOI: 10.7875/leading.author.3.e009
Atsushi J. Nagano & Hiroshi Kudoh: Integration of transcriptomics and meteorology.
要 約
分子生物学では,通常,実験は均質な実験室の環境において行われる.一方,生物の本来の生育場所は複雑に変動する野外の環境である.そのような複雑な環境における遺伝子の発現を解析するための方法として,気象データを組み合わせた統計モデリングがある.統計モデリングによる解析により,ハクサンハタザオの花成制御遺伝子であるAhgFLC遺伝子の発現は過去6週間の気温により説明できることがわかった.また,イネのトランスクリプトームの変動は概日時計と気温により大部分が説明できることがわかった.気温や日長など環境情報から季節を知るために必要となるしくみをシグナル-ノイズ比の観点から検討したところ,それらは統計モデリングにより得られた野外の環境における遺伝子の発現にみられた特徴と一致していた.さらに,その特徴は生物気象学における研究の多くの結果とも一致していた.野外を中心とする生態学や生物気象学などの成果と,実験室を中心とする分子生物学の成果の統合的な理解のため,トランスクリプトームデータと気象データの統計モデリングは重要である.
はじめに
いわゆる分子生物学では,通常,実験は均質な実験室の環境において,遺伝的に均一なモデル生物を用いて行われる.問題をなるべく単純化し,明瞭な結果が得られるように実験条件を整える.慎重に制御された遺伝的な背景および環境を用いて,研究の対象としている要因以外を極力そろえて実験を行う.その結果,これまでに生物のさまざまな形質の背後にある分子機構が明らかにされてきた.
一方,いうまでもなく,生物の本来の生育場所は実験室ではなく野外である.生物の適応形質は自然生息地における自然選択によりかたちづくられたものであるので,それらの機能は自然生育地の環境において評価されるべきであろう.また,作物はその大半が野外の圃場において栽培されるため,作物の生産性の向上を望むのであれば,実際に野外の圃場の環境において起こっていることを理解する必要があるだろう.このように,野外における生物の実態を理解したいという要求はごく自然なものである.しかしながら,この要求に対し,実験室における分子生物学の発想により応えようとすると困難につきあたる.そのひとつが,野外の環境の複雑さである.さきに述べたように,実験室では気温や光を一定にするなど,比較的単純な条件に制御して実験を行う.一方,野外では気温ひとつとっても刻一刻と激しく変動する.もちろん,変動するのは気温だけでなく,光条件や湿度などが複雑に関係しあいながら変動している.
このレビューでは,このように複雑な野外の環境における生物の環境応答を分子生物学的な視点から取り扱った研究を紹介する.まず,中心となる方法論として,野外の環境における遺伝子の発現と気象データとを組み合わせた統計モデリングについて解説する.つぎに,実際の研究事例として,野生植物であるハクサンハタザオ(Arabidopsis halleri subsp. gemmifera)についての研究,および,主要な作物であるイネ(Oryza sativa)についての研究を紹介する.さらに,野外の環境において研究を行うことにより可能になる特徴的な議論の例として,環境からのシグナルの検出とノイズに対する頑健性との関係をとりあげる.最後に,このような研究が分子生物学と生物気象学の蓄積を統合的に理解することに役だつことについて説明する.
1.トランスクリプトミクスと統計モデリングによる複雑な環境における生物の応答の理解
野外における環境変動の複雑さを京都市の気温を例としてみる.まず,日周変動がある(図1a).晴れた日には昼の気温が高く夜の気温は低い.これは雨の日には必ずしもあてはまらない.季節にもよるが,昼夜の気温差は10℃以上になることも多い.年周変動のようなより長いスパンの変動や(図1b),天候の変化などによるより短いスパンの変動も存在する.このように複雑に変動する野外の環境への生物の応答を,単純な環境に制御された実験室において明らかにすることは容易ではない.たとえば,高温ストレスの研究でしばしば使われる20℃で一定の条件から35℃で一定の条件へと一気に変化する状況は,野外では普通は起こらない.その代わり,気温は細かい変動をくり返しながら徐々に上昇する.気温の上昇が数分間で起こる場合と数時間かけて起こる場合とで,生物の応答は根本的に異なっているかもしれない.そう考えると,ひとくちに高温ストレスといっても条件設定の可能性は無数にあることがわかる.無数にある可能性のうち,どこからはじめるべきであろうか.その答えのひとつが,実際の野外の環境において生物の応答を調べることである.
では,そのために何を測定するべきだろうか.トランスクリプトームはもっとも魅力的な測定対象のひとつであろう.DNAマイクロアレイ法やRNA-Seq法によるトランスクリプトミクスは網羅性および定量性が高く,現状において生物システムのもっとも包括的な測定手法である.その網羅性ゆえに,トランスクリプトームのデータは直接的あるいは間接的にさまざまな生物現象の情報を内包すると期待できる.たとえば,これまでに分子生物学などにより明らかにされたさまざまな知見が遺伝子にひもづけられるかたちでデータベースに蓄積されており,それらの知見を用いることにより多様な生理状態について考察することが可能になる.また,遺伝子の発現は数分間から数時間で100倍以上も変動することがある1,2).この短時間で大きく変動しうる性質も,野外の環境における生物の環境応答を知るための測定対象として適している点であろう.また,野外の環境における栽培あるいは研究は,実験室で行う場合よりはるかに労力がかかる.くわえて,環境条件は自然まかせになるため時期が限られ,多くの場合,年に1回きりのチャンスにおいて可能なかぎりのことをすることになる.多数の項目をいちどに測定できるトランスクリプトミクスは,労力の面でも野外における研究に適している.
数は多くないものの,10年以上まえから野外の環境における遺伝子の発現の研究ははじまっていた3-6).近年では,イネ7-9),シロイヌナズナ10)(Arabidopsis thaliana),トウモロコシ11)(Zea mays),ビッグブルーステム12)(Andropogon gerardii),ヨーロッパヤマナラシ13)(Populus tremula),フタバガキ科の熱帯性植物14)(Shorea beccariana)など,モデル植物や作物を中心として野外の環境においてトランスクリプトームのデータが取得されるようになってきている.しかしながら,これらのうち気象データなどを用いて野外における環境変動との関係を解析している研究はわずかである.特定の現象の解析を目的としてデザインされた実験においては,気象条件の変動は単に結果を乱す要因と考えられることが多い.シロイヌナズナ10),ビッグブルーステム12),ヨーロッパヤマナラシ13) を用いた研究においては,気象データとの関係を解析している.直観的な解釈のむずかしい結果ではあるが,いずれの研究においても気温の影響が検出されるなど,一定の成果が得られている.しかしながら,サンプリングの時点数(日数)が少ないことや,データ解析手法における制約などから,それ以上にふみこんだ議論はできていない.
仮に,十分な量の気象データと野外におけるトランスクリプトームのデータが利用できるとするなら,どのように解析すれば変動する野外の環境とトランスクリプトームとの関係を明らかにできるであろうか.解析手法や切り口はいくつもあり,それぞれに一長一短がある.そこで,最終的に得たい情報にあわせた手法を選ぶことが重要になる.ここでは,統計モデリングとよばれるアプローチを採用する(図2).本来,統計モデリングという用語はかなり広い意味をもつが,ここでは,トランスクリプトームのデータ解析においてよく使われるクラスタリングと対比して,解析者が説明変数およびモデルをあたえる回帰をさすことにする.統計モデリングを用いる利点は,研究者が解釈したい切り口を仮定としてあたえて解析できるため,結果を解釈しやすい点である.統計モデリングでは,遺伝子の発現量のような説明される側のデータと,気温やサンプリングの時刻など説明する側のデータとの関係を数式で表わす必要がある.数式というとハードルが高いと感じられるかもしれないが,目的に応じて単純なものでもよい.たとえば,概日時計に由来するサンプリングの時刻に依存した発現変動として,単純な正弦関数を用いるなどである.また,生態学や作物学,生物気象学では,環境要因と開花時期,収量との関係など,これまでにさまざまなモデルが提案されている.それらの蓄積をうまく活用して解析を行うこともできる.
2.ハクサンハタザオのFLC遺伝子における長期の低温応答
例として,ハクサンハタザオの自然集団におけるFLC遺伝子の発現の季節変動の解析について紹介する15).ハクサンハタザオはもっともよく研究されているモデル植物であるシロイヌナズナに近縁な種のひとつで,わが国に自生する.花成制御遺伝子であるFLC遺伝子はMADSボックス型の転写因子をコードしており,シロイヌナズナやその近縁の種において保存されている15-18).一年草および多年草のいずれにおいても,温度に依存的な花成の時期を制御する遺伝子としてはたらいていることが実験室において確認されている19).長期の低温によりFLC遺伝子の発現抑制が起こり,そののち,FLC遺伝子により発現の抑制されていたFT遺伝子の発現亢進が起こることで花成が導かれる.一年草であるシロイヌナズナのFLC遺伝子の発現抑制は低温処理の長さに対し量的に起こるが20),温度の上昇したのちの発現抑制は一生をおえるまでずっと維持される21).すなわち,この場合はFLC遺伝子の発現の季節的な制御は,葉をつくる時期である栄養成長相から花を咲かせる時期である繁殖成長相への移行をもって打ち切られていると考えられる.シロイヌナズナやハクサンハタザオに近縁の多年草であるArabis alpinaのもつFLC遺伝子のオーソログであるPEP1遺伝子の発現も,シロイヌナズナの場合と同様に継続的な低温により抑制され,この抑制はPEP1遺伝子座におけるヒストンH3のLys27のトリメチル化のレベルの上昇と相関している17).興味深いことに,PEP1遺伝子の発現の抑制は低温処理ののち温度を上昇させた場合には安定に維持されない.多年草はその生活史において,気温の季節変動に対しくり返し応答する必要がある.PEP1遺伝子の発現制御はそのための分子基盤の一端を担うと示唆される17).では,自然環境において,本当に多年草のもつFLC遺伝子のオーソログが気温の季節変動を認識するためのしくみとしてはたらいているのだろうか.
ハクサンハタザオの自然個体群を用いてFLC遺伝子のオーソログ(AhgFLC遺伝子)の発現の季節変動が調べられた15).自然生育地において自生している植物を用いたため,それぞれの個体は気温の長期にわたる季節変動だけでなく,大きな日周変動やより短期の変動にもさらされていた.2年間にわたり,週1回12時にそれぞれ6個体から葉をサンプリングし,定量PCR法によりAhgFLC遺伝子の発現量が測定された.AhgFLC遺伝子の発現は明瞭な季節変動を示した.発現は晩秋から冬にかけて徐々に低下していき,春になるとふたたび上昇しはじめ,夏前には最大値のレベルにまでもどった.このハクサンハタザオの自然集団では,2月末から3月初旬に抽台,4月末ごろに開花,5月から6月にかけてリバージョン(多年草で観察される,繁殖成長相から栄養成長相へと逆戻りする現象)が起こっていた.これらのイベントはAhgFLC遺伝子の発現の変動と密接に関係していると考えられた.
AhgFLC遺伝子の発現はノイズの大きな気温の変動から季節のパターンをうまく抽出していた(図3a).また,気温の季節パターンとAhgFLC遺伝子の発現の季節パターンとのあいだには遅れがみられた.この遅れのために,気温は上昇しているがAhgFLC遺伝子の発現の低い期間が生じ,そのタイミングで花成が起こっていた.つぎに,このようなAhgFLC遺伝子の発現パターンが気温によりどのように説明されるか,積算温度モデルを用いて解析された.積算温度は,ある一定の期間においてある閾値をうわまわった(あるいは,したまわった)気温の積算と定義される(図3b).積算温度は生態学や作物学において気温から開花日を予想するモデルなどに広く用いられている.実験室における解析から,シロイヌナズナのFLC遺伝子の発現は低温に影響されることが知られていたので,AhgFLC遺伝子の発現データの解析の際には閾値をしたまわった気温の積算が用いられた.解析の結果,2年間にわたるAhgFLC遺伝子の発現の変動をもっともよく説明したのは,過去42日間(6週間)の10.5℃以下の気温の積算であった15).驚くべきことに,このモデルは2年間の発現変動のうちの83%を説明することができた.閾値と比べ参照する過去の期間の長さはより重要で,6週間より長くても短くてもモデルの説明力はいちじるしく低下した.さらに,このモデルは培養装置を用いて人工的に気温を変化させた場合の応答も高い精度で予測することができた.
この研究の重要性のひとつとして強調したいのは,それが自然環境において遺伝的に不均一な複数の個体を用いて行われた点である.自然生育地では,しばしば気温以外の要因のほうがより大きく変動している.実際に,この研究に用いられたサンプルは,近傍の川の氾濫や積雪による被覆,および,乾燥ストレスにさらされていた.にもかかわらず,AhgFLC遺伝子の発現の変動はその大部分が過去の気温のみで説明することができた.この結果は,AhgFLC遺伝子の発現の制御機構は気温の季節変動を検出できるだけでなく,遺伝的な背景の不均一性や複雑な自然環境におけるノイズに対し,十分な頑健性をもつことを示した.本来,ハクサンハタザオは遺伝的な背景や微小な環境の異なる複数の個体のあいだで同じタイミングで開花する必要がある.この頑健性は,そのような開花の同調に重要な役割をはたしているのかもしれない.
3.圃場におけるイネのトランスクリプトームのデータの統計モデリング
ハクサンハタザオのAhgFLC遺伝子の例では,低温に応答することが既知であった1つの遺伝子のみを対象としていた.トランスクリプトームを考えると,当然,気温だけではなく光や降水などほかの環境条件が遺伝子の発現に影響する可能性もある.また,環境条件のほかに,日齢や体内時計なども遺伝子の発現に大きく影響するかもしれない.それらの影響の相対的な強さは遺伝子ごとに異なっていると考えられる.そこで,より包括的に,野外の環境におけるトランスクリプトームの変動に対しどのような要因が支配的に影響するのか,イネにおいて調べた例について紹介する22).
茨城県つくば市の実験圃場において,6月から9月にかけて500サンプル以上の葉が収集された.遺伝的な背景および葉の発生ステージの影響を極力うけないよう,系統としてはおもに日本晴を用い,もっとも若い完全展開葉に固定してサンプリングを行った.一方,時刻および気象の条件に関しては,豪雨が観測された日を含む9セットの2時間おき48時間のサンプルなど,さまざまなサンプルが含まれていた.これらのサンプルを用いてDNAマイクロアレイ解析が行われた.また,同時に解析に用いる気象データとして,圃場の近くに位置する気象庁の地上気象観測地点のデータが用いられた.全国に点在する地上気象観測地点では,気温,相対湿度,気圧,風向および風速,全天日射量,降水量などについて,1分ごとのデータが蓄積されている.統計モデリングにあたっては,日齢,体内時計,環境要因を考慮したモデルが用いられた.環境要因からの影響の部分については,さきに説明したような積算温度モデルを拡張したものが用いられた.個々の遺伝子がどのような環境要因の影響をうけるかが事前にわからないため,積算“温度”だけではなく,相対湿度,気圧,風速,全天日射量,降水量も含めた6通りの気象データから,遺伝子の発現をもっともよく説明するデータをおのおのの遺伝子について選ぶというアプローチがとられた.さらに,一日のうち特定の時間帯のみに環境応答を示す現象(ゲート効果)もモデルに組み込まれた.結果的に,遺伝子ごとに最大で17のパラメーターをもつモデルになった.このモデルと,約500サンプル分のトランスクリプトームのデータ,半年分の気象データを用いて,パラメーターを最適化する計算を行った.その結果,葉で発現のみられた遺伝子のうち96.7%について予測に利用することの可能なモデルパラメーターが得られた.このモデルが妥当な予測を行えることは,パラメーターの最適化に用いたものとは独立の野外サンプルのデータ,および,実験室において温度条件および光条件を変えた際のデータを用いて検証された.なお,個々の遺伝子の結果は,データベースFiT-DBとして公開されている(URL:http://fitdb.dna.affrc.go.jp/).
トランスクリプトームの全体にわたり結果のパラメーターを集計することにより,野外環境においてどのような要因が支配的かを知ることができた.たとえば,概日時計の影響がもっとも大きく,そのつぎに気温や光などへの環境応答が大きく,植物の日齢はそれほど影響しないことがわかった.また,環境応答を示す遺伝子の約8割は気温により説明されたことから,環境応答においては気温の影響がとくに大きいことがわかった.このほかにも,さまざまなことを結果のパラメーターから知ることができた.たとえば,環境応答を示す遺伝子の約3割でゲート効果のみられること明らかになった.また,これだけの量のデータを用い,かつ,統計モデリングを行ったことにより議論できるようになったことに,個体間の発現量のばらつきの大きさがある.概日時計や環境応答などの決定論的な変動では説明できない個体間の確率的なばらつきの大きさが遺伝子ごとに異なっており,リボソームやヒストンなど基本的な細胞機能にかかわる遺伝子では小さく,病害応答などの外的な刺激にかかわる遺伝子では大きいことが見い出された.さらに,得られたモデルを用いれば,任意の条件での予測が可能となる.ただし,モデルの最適化に用いたデータのとられた地点の野外環境から大きく外れた条件では予測精度が低下すると考えられるため,その利用には慎重な検討が必要である.
4.光受容体の感度と日長の測定の精度
野外の環境において研究を行うことにより可能となる特徴的な議論として,環境からのシグナルの検出とノイズに対する頑健性との関係をとりあげる.シグナルとノイズの相対的な大きさは一般にシグナル/ノイズ比(S/N比)として知られている.高いシグナル/ノイズ比はシグナルの大きさがノイズの大きさより十分に大きいことを意味しており,その場合,目的とするシグナルは明瞭に検出できる.
まず例として,イネの光に対する応答を考える.光に対する応答を考慮したほうがよい遺伝子について,光に対する応答の閾値のパラメーターを集計してみると,そのほとんどがごく低い値になっていた22).つまり,真っ暗か否かの2値的な応答をしていることになる.このことは,生化学的にはファイトクロムなどの光受容体の感度がきわめて高いことに対応していると思われる23).なぜ,光に対しこのように過剰なまでに高い感度で応答するのだろうか.そのひとつの解釈が,環境からのシグナルの検出とノイズに対する頑健性の観点から得られる.シグナルとノイズといった場合,何がシグナルで何がノイズなのかは,当然,対象により異なる.ここで注目するのは日長の変化である.イネは日長の変化に鋭敏に応答し花成を行う24).そのため,日長の変化を正確に測定しシグナルとして用いることは重要であろう.このとき,日々の天候や周囲の植生などに起因する日長測定の誤差がノイズとみなされる.もし仮に,より感度の低い光受容体を用いて日長を測定すると,日々の天候により主観的な日の長さが大きく変動してしまうだろう.つまり,朝夕に雲が多く暗い日には日が短く感じられてしまう.逆に,非常に高感度な光受容体を用いて日長を測定すれば,曇りであろうがほとんど影響をうけずに日長を測定することができる.実際に,光に対するさまざまな閾値でのシグナル/ノイズ比を計算すると,閾値が低いほどシグナル/ノイズ比は高い値となる22).以上の議論から,光に対する高感度での応答は日長に対し正確に応答するために有用だと考えられる.じつは,この結論は生物の光周性において古くから知られている,光に対する閾値は低く光周性は天候によらないという事実と同じものである25).
5.気温の季節変動の特徴とシグナルを抽出するしくみ
気温の季節シグナルとしての特徴から,気温をもとに季節を知るためのしくみに必要な特徴を明らかにする.赤道の付近を除き,日の出から日の入りまでの時間(日長)は規則的な年周期の変動を示し,その変動は緯度から予測が可能である25,26).この変動はそもそも地球の地軸が23.4℃傾いていることに起因する.日長は夏至で最長,冬至で最短になる(北半球では,夏至はだいたい6月22日,冬至はだいたい12月22日).また,日射量の年周期の変動は季節をかたちづくる27,28).日長と気温の年間の変化は,温帯域より高緯度では植物の季節応答の主要な決定因子として十分な大きさがある29).ここで例として,京都市の気温のデータから検討する(図1).ある1地点のデータを解析した結果ではあるものの,以下の大半の議論は地球上のほかの任意の地点,少なくとも,温帯域の地点において成り立つ27,28).気温は明瞭な季節変動を示し,そのピークは日長の変化から1カ月から1.5カ月の遅れをもつ27,30).気温の季節変動は数カ月のスケールのパターンである(図1b).季節のパターンがあるのと同時に,気温はさまざまな時間スケールで激しく変動している.日平均気温の変化をみると,日々の変動やより長期の変動のほうが大きいことがわかる(図1b).たとえば,春は長期的には徐々に気温が上がっていく傾向があるものの,前日より涼しい日や,前の週より涼しい週がくることも珍しいことではない.さらに,気温の日中変動をみると,1日のなかでの気温の変化の幅は,しばしば1~2カ月以上の期間の気温の変化の幅に相当することがわかる(図1b).すなわち,気温は日周変動,日間変動,さらには,より短期あるいは長期の変動を含み,季節シグナルの抽出においてはそれらが深刻なノイズになる.すなわち,気温の季節変動はそのままではシグナル/ノイズ比の低い情報であることがわかる.一般に,シグナル/ノイズ比の低い情報を用いる場合,より長い期間の測定(より多くの測定)が信頼性の高いシグナルの検出のため必要となる.
では,どの程度の長さの測定が必要なのだろうか.ここでは議論を簡単にするため,移動平均を用いて検討する.たとえば,3日間の気温の移動平均であれば,過去3日分の気温の平均値がその時点での値になる.京都市の気温のデータを用いて,移動平均期間を1日間,7日間,42日間,270日間としたときの移動平均を計算した(図4).移動平均による平滑化により短期のノイズは取り除かれ,滑らかな季節変動のパターンが得られた.移動平均期間を長くとるにしたがってノイズはめだたなくなっていき,季節シグナルがはっきりとしてくることがわかる.移動平均期間が42日間の場合にはノイズはほとんど無視できる程度になっている.より長い移動平均期間をとることによりシグナル/ノイズ比はさらに向上するものの,別の2つの影響も現われる.ひとつはシグナルの絶対的な大きさの減少であり,もうひとつは季節シグナルの遅れである.このため,実際には参照する過去の期間が長すぎるのもよくない.以上の議論から,過去4~6週間程度の気温を参照するしくみがあれば,短期のノイズを無視し季節シグナルをとらえることができると考えられる.
この“過去4~6週間程度の気温を参照するしくみ”は,気温の気象データのみから推定されたものであった.一方,さきに述べたハクサンハタザオにおけるAhgFLC遺伝子の発現の変動においては,気温を参照する過去の期間は6週間であった.興味深いことに,移動平均と積算との違いはあるが,両者が気温の情報を利用する期間はよく一致している.つまり,AhgFLC遺伝子の発現の制御は,自然環境におけるノイズの大きい気温の情報から効率よく季節のシグナルを抽出するためのしくみとして最適化されていると考えられる.
この6週間という長い期間の気温を積算していくための分子機構の一端が,実験室におけるシロイヌナズナのFLC遺伝子の詳細な研究により明らかにされている.4℃などで一定にする低温処理により,2~3週間程度でFLC遺伝子の発現は最低のレベルまで抑制される31).しかしながら,FLC遺伝子の発現抑制の状態が完全に確立し,低温処理が終了したのちまで発現抑制を維持させるためには,4~8週間の低温処理が必要である20,32).FLC遺伝子の抑制が完全に確立した状態では,FLC遺伝子を含むゲノム領域がヒストン修飾を介してヘテロクロマチン化され,それが遺伝子の発現抑制の記憶としてはたらく33-36).FLC遺伝子の発現抑制の完全な確立には低温の期間の長さにしたがい,非コードRNAであるCOOLAIR 31) やCOLDAIR,Polycomb抑制複合体2を構成するタンパク質としてVRN2,SWN,FIE,MSI1 34),PHDファミリータンパク質としてVIN3,VRN5,VIL2 34,37-39) などがはたらく,多段階の過程により達成される40).これらの分子機構が実際の自然環境において低温の記憶としてはたらいているのか,また,さきに述べたハクサンハタザオのような多年草ではどのように制御されているのかについては,今後の研究が待たれる.
6.生物気象学の蓄積をみなおす
ハクサンハタザオにおいてみられたような気温の記憶と花成の制御との関係は,どのくらい一般的なのだろうか.さまざまなノイズを含む気温の情報から効率的に季節シグナルを抽出するしくみについての議論は,特定の種あるいは地域に依存したものではないので幅広く成り立つ可能性がある.生物気象学では,気温などの環境条件と開花など生物の季節応答との関係が長年にわたり調べられてきた.過去数十年ものあいだ,初開花日が記録されデータの解析が行われてきたのである41-43).初開花日は年にいちどしかないため,環境要因がどのように開花日の年ごとの変動に影響するかを統計的に解析するためには,20年分かそれ以上のデータが必要になる.実際の解析では,年ごとの気温の変動を反映する指標と初開花日とのあいだの相関を調べる.生物の季節現象に対する気候変動の影響はこれまでもしばしば解析されており,地球の温暖化は春の開花や展葉を有意に早めていることが報告されている44-47).
ここでみなおしたいのは,過去数週間から数カ月間の環境要因のデータにもとづいて初開花日を予測しようとする研究である.これらの研究におけるおもな問題設定は,どの環境要因のどのくらいの期間の過去の値が初開花日の変動に影響するのかということである.たとえば,英国中部における36年間(1954年~1989年)にわたる243種の顕花植物の初開花日が調べられている48).初開花日には温度が大きく影響することが見い出され,243種のうち219種において過去数カ月の気温から初開花日のばらつきの大半を説明することができた.早期(1~4月)に開花する種の初開花日は過去1~2カ月の気温の影響の強いものが多く,夏季(5月以降)に開花する種では最大で過去4カ月の気温が重要であった.また,イタリア中部における57種の顕花植物について,過去の気温と降水の情報を用いた23年間(1960年~1982年)の初開花日のモデリングが成功している49).2月から5月に開花する種では過去20~38日間の環境の影響を考慮することにより初開花日をもっともよく説明でき,6月から9月に開花する種では過去41~83日間(多くは,60日以下)の環境を考慮することにより初開花日をもっともよく説明することができた.初開花日をモデリングしたほかの研究においても,その多くで過去1~2カ月間の気温がもっとも重要な説明変数であった50-53).
種や地域による違いはあるものの,おおむね過去3~10週間の気温が年ごとの初開花日のばらつきを説明する効果が大きかった.初開花日に影響する過去の気温の期間は,温度に依存的な発生の過程の期間を反映しているというのが伝統的な解釈のひとつである.しかしながら,さきに述べたように,ノイズの多い自然環境において季節シグナルを抽出するという観点からみたとき,参照すべき過去の期間と生物気象学においてこれまで報告されてきた期間との一致が見い出される.このことから,年ごとの開花日のばらつきを,単に発生の過程にかかる時間が気温により受動的に変化した結果だと無条件にみなすべきではないと考えられる.自然生育地における大きなノイズを含む気温の変動のもとで花成の時期を制御するために進化した,より積極的な応答が含まれているのかもしれない.
おわりに
遺伝子の発現と気象データの統計モデリングを切り口として,野外の環境における生物の環境応答を考える試みを紹介した.自然生育地や圃場といった,より現実的な条件における解析と解釈は実用において重要であるだけでなく,実験室における分子生物学の研究にも新たな視点を提供するだろう54).ここまで述べてきたような,シグナル/ノイズ比に注目した議論はそのひとつの例である.ほかにもたとえば,複数のストレスが同時にかかる状況やストレスの強度が時間的に変化する状況において何が起こるのかを,実験室における分子生物学の研究によりていねいに解析することは価値があるだろう.その際,無限にある条件設定の可能性から注目すべきものを絞り込むため,野外の環境における生物の応答を解析した結果が役にたつと考えられる.また,生態学や生物気象学など野外を中心に研究が進められてきた分野の知見と,実験室における分子生物学により蓄積された知見の統合的な理解を進めるうえでも,トランスクリプトームのデータと気象データの統計モデリングは重要になるだろう(図5).
文 献
- Goda, H., Sasaki, E., Akiyama, K. et al.: The AtGenExpress hormone and chemical treatment data set: experimental design, data evaluation, model data analysis and data access. Plant J. 55, 526-542 (2008)[PubMed]
- Kilian, J., Whitehead, D., Horak, J. et al.: The AtGenExpress global stress expression data set: protocols, evaluation and model data analysis of UV-B light, drought and cold stress responses. Plant J. 50, 347-363 (2007)[PubMed]
- Merquiol, E., Pnueli, L., Cohen, M. et al.: Seasonal and diurnal variations in gene expression in the desert legume Retam raetam. Plant Cell Environ., 25, 1627-1638 (2002)
- Anderson, J. V., Gesch, R. W., Jia, Y. et al.: Seasonal shifts in dormancy status, carbohydrate metabolism, and related gene expression in crown buds of leafy spurge. Plant Cell Environ., 28, 1567-1578 (2005)
- Mayrhofer, S., Teuber, M., Zimmer, I. et al.: Diurnal and seasonal variation of isoprene biosynthesis-related genes in grey poplar leaves. Plant Physiol., 139, 474-484 (2005)[PubMed]
- Manitasevic, S., Dunderski, J., Matic, G. et al.: Seasonal variation in heat shock proteins Hsp70 and Hsp90 expression in an exposed and a shaded habitat of Iris pumila. Plant Cell Environ., 30, 1-11 (2007)[PubMed]
- Sato, Y., Antonio, B., Namiki, N. et al.: Field transcriptome revealed critical developmental and physiological transitions involved in the expression of growth potential in japonica rice. BMC Plant Biol., 11, 10 (2011)[PubMed]
- Fukayama, H., Sugino, M., Fukuda, T. et al.: Gene expression profiling of rice grown in free air CO2 enrichment (FACE) and elevated soil temperature. Field Crops Res., 121, 195-199 (2010)
- Izawa, T., Mihara, M., Suzuki, Y. et al.: Os-GIGANTEA confers robust diurnal rhythms on the global transcriptome of rice in the field. Plant Cell, 23, 1741-1755 (2011)[PubMed]
- Richards, C. L., Rosas, U., Banta, J. et al.: Genome-wide patterns of Arabidopsis gene expression in nature. PLoS Genet., 8, e1002662 (2012)[PubMed]
- Hayes, K. R., Beatty, M., Meng, X. et al.: Maize global transcriptomics reveals pervasive leaf diurnal rhythms but rhythms in developing ears are largely limited to the core oscillator. PLoS One, 5, e12887 (2010)[PubMed]
- Travers, S. E., Tang, Z., Caragea, D. et al.: Variation in gene expression of Andropogon gerardii in response to altered environmental conditions associated with climate change. J. Ecol., 98, 374-383 (2010)
- Sjodin, A., Wissel, K., Bylesjo, M. et al.: Global expression profiling in leaves of free-growing aspen. BMC Plant Biol., 8, 61 (2008)[PubMed]
- Kobayashi, M. J., Takeuchi, Y., Kenta, T. et al.: Mass flowering of the tropical tree Shorea beccariana was preceded by expression changes in flowering and drought-responsive genes. Mol. Ecol., 22, 4767-4782 (2013)[PubMed]
- Aikawa, S., Kobayashi, M. J., Satake, A. et al.: Robust control of the seasonal expression of the Arabidopsis FLC gene in a fluctuating environment. Proc. Natl. Acad. Sci. USA, 107, 11632-11637 (2010)[PubMed]
- Lin, S., Wang, J., Poon, S. et al.: Differential regulation of expression by vernalization FLOWERING LOCUS C in cabbage and Arabidopsis. Plant Physiol., 137, 1037-1048 (2005)[PubMed]
- Wang, R., Farrona, S., Vincent, C. et al.: PEP1 regulates perennial flowering in Arabis alpina. Nature, 459, 423-427 (2009)[PubMed]
- Kemi, U., Niittyvuopio, A., Toivainen, T. et al.: Role of vernalization and of duplicated FLOWERING LOCUS C in the perennial Arabidopsis lyrata. New Phytol., 197, 323-335 (2013)[PubMed]
- Albani, M. C. & Coupland, G.: Comparative analysis of flowering in annual and perennial plants. Curr. Top Dev. Biol., 91, 323-348 (2010)[PubMed]
- Sheldon, C. C., Rouse, D. T., Finnegan, E. et al.: The molecular basis of vernalization: the central role of FLOWERING LOCUS C (FLC). Proc. Natl. Acad. Sci. USA, 97, 3753-3758 (2000)[PubMed]
- Choi, J., Hyun, Y., Kang, M. J. et al.: Resetting and regulation of FLOWERING LOCUS C expression during Arabidopsis reproductive development. Plant J., 57, 918-931 (2009)[PubMed]
- Nagano, A. J., Sato, Y., Mihara, M. et al.: Deciphering and prediction of transcriptome dynamics under fluctuating field conditions. Cell, 51, 1358-1369 (2012)[PubMed]
- Franklin, K. A. & Quail, P. H.: Phytochrome functions in Arabidopsis development. J. Exp. Bot., 61, 11-24 (2010)[PubMed]
- Itoh, H., Nonoue, Y., Yano, M. et al.: A pair of floral regulators sets critical day length for Hd3a florigen expression in rice. Nat. Genet. 42, 635-638 (2010)[PubMed]
- Withrow, R. B.: A kinetic analysis of photoperiodism. in Photoperiodism and Related Phenomena in Plants and Animals (Withrow, R. B. ed.), pp. 439-471, American Association for Advancement of Science, Washington DC (1959)
- Lee, R.: Latitude and photoperiodism. Arch. Met. Geoph. Biokl. Ser. B, 18, 325-332 (1970)
- Trenberth, K. E.: What are the seasons? Bull. Am. Meteorol. Soc., 64, 1276-1282 (1983)
- Jones, P. D., New, M., Parker, D. E. et al.: Surface air temperature and its changes over the past 150 years. Rev. Geophys., 37, 173-199 (1999)
- Battey, N. H.: Aspects of seasonality. J. Exp. Bot., 51, 1769-1780 (2000)[PubMed]
- Prescott, J. A. & Collins, J. A.: The lag of temperature behind solar radiation. Q. J. Roy. Meteorol. Soc., 77, 121-126 (1951)
- Swiezewski, S., Liu, F. & Magusin, A.: Cold-induced silencing by long antisense transcripts of an Arabidopsis Polycomb target. Nature, 462, 799-802 (2009)[PubMed]
- Shindo, C., Lister, C. & Crevillen, P.: Variation in the epigenetic silencing of FLC contributes to natural variation in Arabidopsis vernalization response. Genes Dev., 20, 3079-3083 (2006)[PubMed]
- Bastow, R., Mylne, J. S., Lister, C. et al. et al.: Vernalization requires epigenetic silencing of FLC by histone methylation. Nature, 427, 164-167 (2004)[PubMed]
- De Lucia, F., Crevillen, P., Jones, A. M. E. et al.: A PHD-polycomb repressive complex 2 triggers the epigenetic silencing of FLC during vernalization. Proc. Natl. Acad. Sci. USA, 105, 16831-16836 (2008)[PubMed]
- Schmitz, R. J., Sung, S. & Amasino, R. M.: Histone arginine methylation is required for vernalization-induced epigenetic silencing of FLC in winter-annual Arabidopsis thaliana. Proc. Natl. Acad. Sci. USA, 105, 411-416 (2008)[PubMed]
- Angel, A., Song, J., Dean, C. et al.: A Polycomb-based switch underlying quantitative epigenetic memory. Nature, 476, 105-108 (2011)[PubMed]
- Sung, S. & Amasino, R. M.: Vernalization in Arabidopsis thaliana is mediated by the PHD finger protein VIN3. Nature, 427, 159-164 (2004)[PubMed]
- Wood, C. C., Robertson, M., Tanner, G. et al.: The Arabidopsis thaliana vernalization response requires a polycomb-like protein complex that also includes VERNALIZATION INSENSITIVE 3. Proc. Natl. Acad. Sci. USA, 103, 14631-14636 (2006)[PubMed]
- Greb, T., Mylne, J. S., Crevillen, P. et al.: The PHD finger protein VRN5 functions in the epigenetic silencing of Arabidopsis FLC. Curr. Biol, 17, 73-78 (2007)[PubMed]
- Song, J., Angel, A., Howard, M. et al.: Vernalization: a cold-induced epigenetic switch. J. Cell Sci., 125, 3723-3731 (2012)[PubMed]
- Fitter, A. H. & Peat, H. J.: The ecological flora database. J. Ecol., 82, 415-425 (1994)
- Tooke, F. & Battey, N. H.: Temperate flowering phenology. J. Exp. Bot., 61, 2853-2862 (2010)[PubMed]
- Schwartz, M. D.: Phenology: an Integrative Environmental Science. Kluwer, Dordrecht (2003)
- Bradley, N. L., Leopold, A. C., Ross, J. et al.: Phenological changes reflect climate change in Winsconsin. Proc. Natl. Acad. Sci. USA, 96, 9701-9704 (1999)[PubMed]
- Penulelas, J. & Filella, I.: Responses to a warming world. Science, 294, 793-795 (2001)[PubMed]
- Walther, G. R., Post, E., Convey, P. et al.: Ecological response to recent climate change. Nature, 416, 389-395 (2002)[PubMed]
- Root, T. L., Price, J. T., Hall, K. R. et al.: Fingerprints of global warming on wild animals and plants. Nature, 421, 57-60 (2003)[PubMed]
- Fitter, A. H., Fitter, R. S. R., Harris, I. T. B. et al.: Relationships between first flowering date and temperature in the flora of a locality in central England. Funct. Ecol., 9, 55-60 (1995)
- Censi, C. A. & Ceschia, M.: Forecasting of the flowering time for wild species observed at Guidonia, central Italy. Int. J. Biometeorol., 44, 88-96 (2000)[PubMed]
- Marletto, V., Branzi, G. P. & Sirotti, M.: Forecasting flowering dates of lawn species with air temperature: application boundaries of the linear approach. Aerobiologia, 8, 75-83 (1992)
- Rodriguez-Rajo, F. J., Frenguelli, G. & Jato, M. V.: Effect of air temperature on forecasting the start of the Betula pollen season at two contrasting sites in the south of Europe (1995-2001). Int. J. Biometeorol., 47, 117-125 (2003)[PubMed]
- Roberts, A. M. I.: Exploring relationships between phenological and weather data using smoothing. Int. J. Biometeorol., 53, 463-470 (2008)[PubMed]
- Luedeling, E., Kunz, A. & Blanke, M. M.: Identification of chilling and heat requirements of cherry trees: a statistical approach. Int. J. Biometeorol., 57, 679-689 (2013)[PubMed]
- Kudoh, H. & Nagano, A. J.: Memory of temperature in the seasonal control of flowering time: an unexplored link between meteorology and molecular biology. in Evolutionary Biology: Exobiology and Evolutionary Mechanisms (Pontarotti, P. ed.), pp. 195-215, Springer, Berlin (2013)
著者プロフィール
略歴:2009年 京都大学大学院理学研究科 修了,同年 農業生物資源研究所 特別研究員を経て,2011年より京都大学生態学研究センター 特別研究員.2012年より科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:複雑な環境における生物のシステム動態の理解,予測,制御.
工藤 洋(Hiroshi Kudoh)
京都大学生態学研究センター 教授.
© 2014 永野 惇・工藤 洋 Licensed under CC 表示 2.1 日本