プロテアソームの作動機構と生理作用の新しい理解
2014/10/16
村田 茂穂
(東京大学大学院薬学系研究科 蛋白質代謝学教室)
email:村田茂穂
領域融合レビュー, 3, e011 (2014) DOI: 10.7875/leading.author.3.e011
Shigeo Murata: A new understanding of action mechanism, physiological roles and regulation of the proteasome.

26Sプロテアソームは約2.5 MDa,66個のサブユニットから構成される,真核生物に必須の巨大なタンパク質複合体である.なぜこのように複雑な構造をもっているのか,どのようにタンパク質の分解を実行しているのか,いまだよくわかっていない.しかし近年,クライオ電子顕微鏡を用いた高い分解能での構造解析から大きな進展がみられている.また,プロテアソームの量の多寡とがん,神経変性,老化などヒトの疾患とのかかわりについての報告も蓄積してきた.プロテアソームの発現がどのように制御されているのかについてもほとんど未解明であったが,細胞の内外の栄養環境やストレスに応答してプロテアソームの発現が制御され,寿命の延長,ストレス応答,幹細胞の機能維持に重要な役割をはたしていることがわかってきた.
細胞は恒常性の維持,ストレス応答,機能変換などを実行するための手段のひとつとして,細胞内タンパク質分解を利用している.細胞において役割をおえた(おえるべき)タンパク質や不良なタンパク質の大半は,分解の目印としてユビキチンを付加されたのち,プロテアソームとよばれる巨大なタンパク質複合体により短いペプチド断片にまで消化される.ユビキチン-プロテアソーム系は真核生物において必須の細胞内タンパク質分解系であるが,その要点は,標的となるタンパク質の幅広さと高い選択性の両立,および,無秩序ではなく厳密に制御された分解にある.筆者が大学院生だった1990年代後半はユビキチン-プロテアソーム系の具体的な重要性がわかりはじめたばかりのころであったが,そののちの研究の進展はめざましく,細胞周期,遺伝子発現,シグナル伝達をはじめとした幅広い生命現象において必須の制御機構としてはたらいていることが明らかになり,近年の生命科学を理解するうえで必須の研究分野である.
ユビキチン-プロテアソーム系においていまだ大きな謎につつまれているのが,タンパク質の分解を実行するプロテアソームである.プロテアソームは多数のサブユニットから構築されるきわめて複雑なタンパク質複合体であるが,ここ数年の構造解析の進展により,その詳細な作動機構がわかってきた.また,プロテアソームの機能と老化との関連や,その発現の新しい制御機構も明らかになりつつある.このレビューでは,とくにここ数年で明らかになってきたプロテアソームの作動機構と生理作用を中心に解説する.
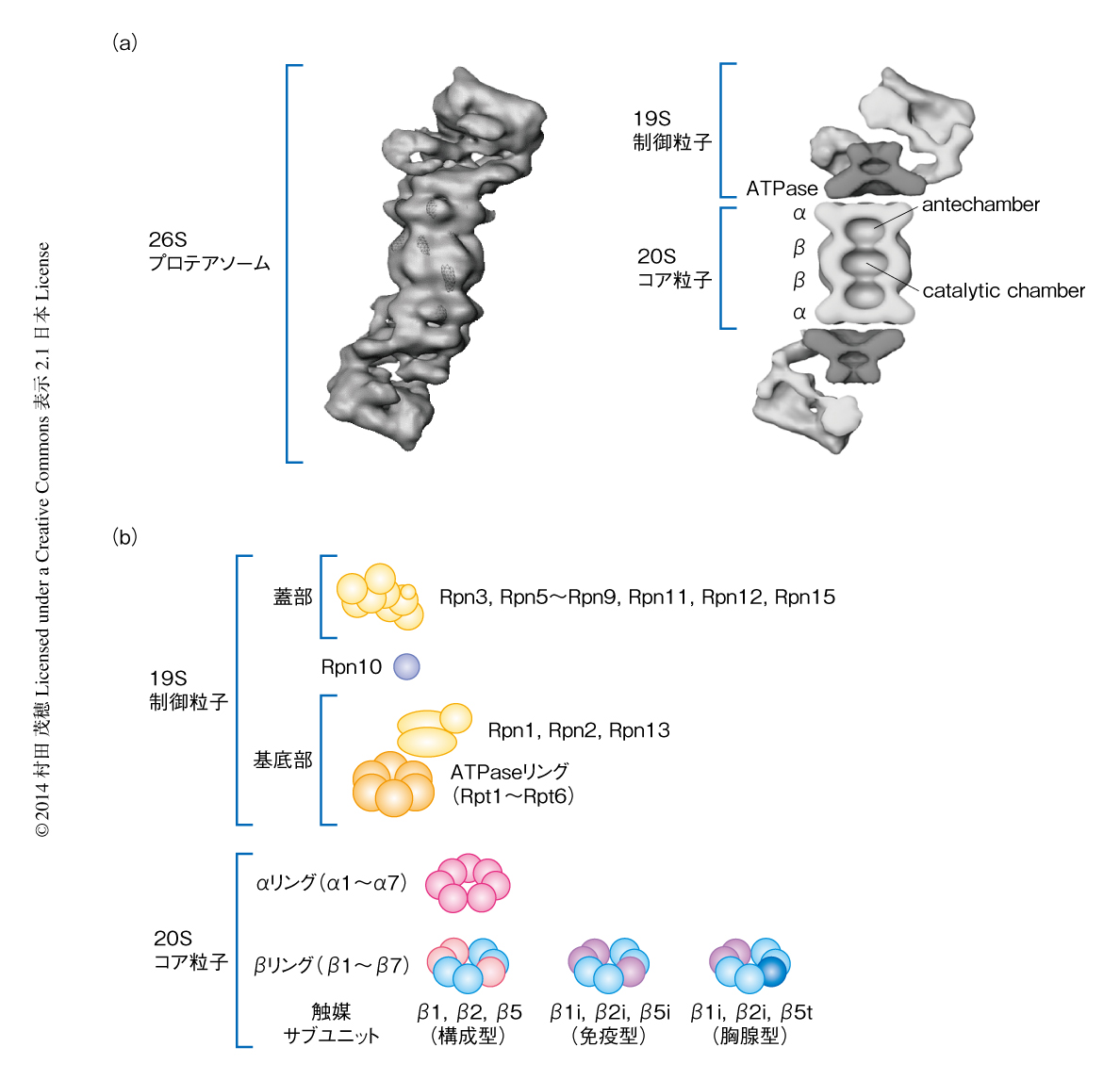
プロテアソームは酵母から哺乳類にいたるすべての真核生物において高度に保存された構造をもつタンパク質複合体である.ユビキチン化されたタンパク質を分解するのは,プロテアーゼ活性をもつ20Sコア粒子(CP,あるいは,20Sプロテアソームともよぶ)の両端または片側に19S制御粒子(RPともよぶ)が会合した26Sプロテアソームである.これは,長さ44 nm,最大直径20 nm,2.5 MDaにも及ぶ巨大な酵素である(図1).
20Sコア粒子はX線結晶構造解析がなされており,構造の詳細が判明している1).7種類のサブユニットα1~α7からなるαリングと,7種類のサブユニットβ1~β7からなるβリングがαββαの順に積み重なった中空の樽状の形状をしており,βリングの内壁にプロテアーゼ活性中心が露出している.β1,β2,β5のみがプロテアーゼ活性をもつ.脊椎動物においては,酵母から保存されている構成的に発現するタイプの20Sコア粒子のほか,インターフェロンγに応答して発現が誘導されるβ1i,β2i,β5iというプロテアーゼ活性をもつ異なるサブユニットが組み込まれた免疫型20Sコア粒子,この免疫型20Sコア粒子からさらにβ5iが胸腺皮質に特異的に発現するサブユニットβ5tと置換した胸腺型20Sコア粒子が存在し,これらはMHCクラスIを介した免疫応答においてきわめて重要なはたらきをしていることが知られているが,詳細についてはほかにゆずる2).
20Sコア粒子が単独のときには入り口となるαリングの中央は閉じており,開口しても13Åの狭いチャネルとなり,解きほぐされた(アンフォールディングされた)タンパク質のみが通過できる.通過した基質タンパク質はαリングとβリングから形成されるantechamber(控えの間)とよばれる空間において解きほぐされた状態で維持され,さらに,2つのβリングにかこまれたcatalytic chamber(触媒空間)に移動して分解される.
20Sコア粒子における分解のために基質タンパク質の前処理を行うのが19S制御粒子である.ATPase活性をもつ6種類のサブユニットRpt1~Rpt6からなるATPaseリングに, ATPase活性をもたない13種類のサブユニットRpn1~Rpn3,Rpn5~Rpn13,Rpn15が会合したタンパク質複合体であり,生化学的な性質やその形成機構から,Rpt1~Rpt6,Rpn1,Rpn2,Rpn13からなる基底部(base)と,Rpn3,Rpn5~Rpn9,Rpn11,Rpn12,Rpn15からなる蓋部(lid)の2つに区分することができる3)(図1).基底部と蓋部は独立して形成されたのちに会合して19S制御粒子ができあがるが,Rpn10は完成した19S制御粒子に会合する.
これまで生化学的および遺伝学的な解析から,いくつかのサブユニットについてはユビキチン化されたタンパク質の分解における役割が知られている.ATPaseサブユニットは基質タンパク質の解きほぐし,20Sコア粒子のαリングの開口,20Sコア粒子への基質タンパク質の送り込みを行う.Rpn10およびRpn13はユビキチン鎖を直接に捕捉する“ユビキチン受容体”としてはたらく.Rpn11はZn2+メタロプロテアーゼであり,ユビキチン鎖の根元を切断することにより基質タンパク質からユビキチン鎖を切り離し分解を促進する.
これらの多数のサブユニットがどのように有機的に連携してユビキチン化されたタンパク質の分解を可能にしているのかを理解するため,19S制御粒子の構造解析がながらく待望されていたが,構造の複雑さ,不均一さから困難をきわめていた.しかし,ここ数年のクライオ電子顕微鏡による単粒子解析技術の進歩により,26Sプロテアソーム全体の高次構造が7~10Åの高分解能で明らかになり,プロテアソームの作動機構の理解が飛躍的に進展した4-11).
クライオ電子顕微鏡による解析を中心に,個別のサブユニットや類似したタンパク質の結晶構造解析の情報やこれまでの生化学的な解析の蓄積を参照することにより,19S制御粒子におけるおのおののサブユニットの配置や活性中心の位置,ATP加水分解にともなう構造変化をはじめ,19S制御粒子が非常に巧妙なしくみでユビキチン化されたタンパク質を処理している機構がわかってきた.
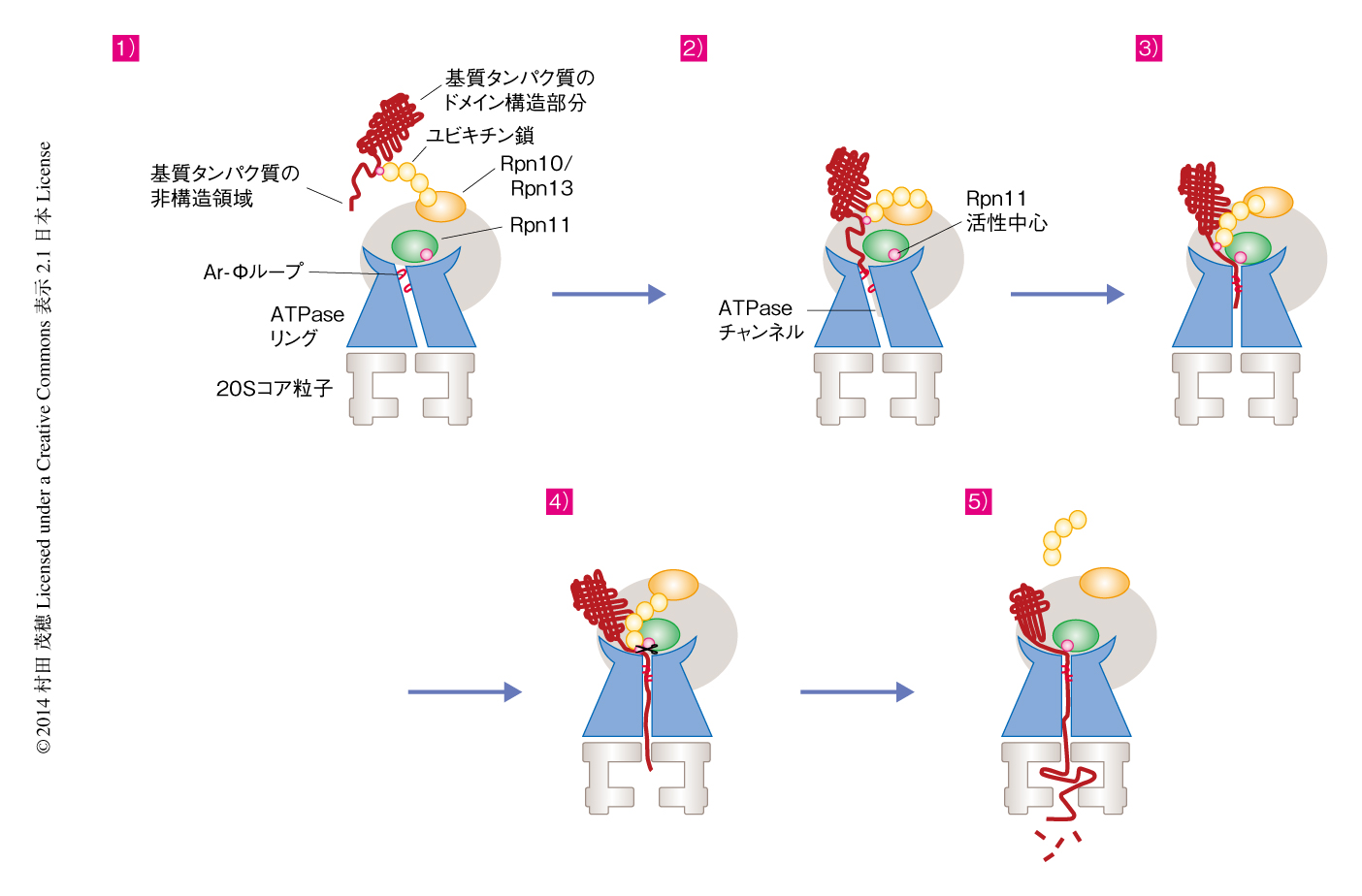
ATPaseサブユニットはRpt1-Rpt2-Rpt6-Rpt3-Rpt4-Rpt5の順にならんでリング構造を形成し,その中央にはチャネルが形成される.ほかのAAA+アンフォールダーゼと同様に,基質を捕捉するAr-Φループ(Ar-Φ:aromatic hydrophobic,芳香性および疎水性)とよばれる疎水性に富んだ領域がおのおののサブユニットからチャネルへむけて突き出している.ATPの存在下(基質タンパク質と会合していない状態を模倣)では,Ar-ΦループはRpt3を頂点,Rpt2を底とし,Rpt6がそのあいだを連結するらせん階段状に配列し,さらに,ATPaseリングのチャネルの軸は20Sコア粒子のチャネルの軸から傾いている.しかし,非加水分解性のATPアナログであるATPγSの存在下(基質タンパク質と会合した状態を模倣)では,おのおののAr-Φループはほぼ同一の平面にそろい,ATPaseリングのチャネルの軸と20Sコア粒子のチャネルの軸が一致する5)(図2).このように,ATPの加水分解サイクルにともなう立体構造の変化により,基質タンパク質の高次構造を解きほぐしながら20Sコア粒子へと送り込んでいる機構がわかってきた.
ATPaseリングは20Sコア粒子のαリングと直接に会合する.Rpt2,Rpt3,Rpt5のC末端に存在するHbYX配列(疎水性アミノ酸-チロシン-任意のアミノ酸からなる配列)がαサブユニットのあいだにあるポケットに突き刺さることにより,αリングに構造変化が起こり開口し,解きほぐされた基質タンパク質が20Sコア粒子へと送り込まれる.
ユビキチン化されたタンパク質の捕捉はRpn10およびRpn13により行われる.これら“ユビキチン受容体”はアンテナのように19S制御粒子の頂上の周辺部に互いに90Å離れて位置している(図3).これまでの生化学的な解析から,4つ以上のユビキチンが連なったテトラユビキチン鎖で修飾されていることが基質タンパク質がプロテアソームにより効率的に分解されるために必要であることが知られている.2つのユビキチン受容体の距離はちょうどテトラユビキチン鎖の長さに相当することから,それらの協調により高い親和性でユビキチン鎖を捕捉している可能性が示唆されている.
しかし,ユビキチン鎖のみではプロテアソームによる分解には十分ではなく,基質タンパク質が30アミノ酸残基以上の長さの非構造領域をもつことが必要であり,この領域がATPaseチャネルに入り込みAr-Φループにより捕捉されることにより,はじめて分解への運命が決定づけられる12).
脱ユビキチン化酵素Rpn11はユビキチン鎖の根元を基質タンパク質から切り離す.このはたらきはユビキチン化されたタンパク質の分解に必須の過程であるが,ATPの加水分解に依存的に基質タンパク質の解きほぐしが開始されてはじめて実行される.すなわち,ATPaseリングの立体構造の変化は19S制御粒子の全体の構造変化をともない,基質タンパク質の解きほぐしのときには,あたかも解きほぐされつつあるポリペプチド鎖を間近で見張るかのように,Rpn11の活性中心はATPaseリングのチャネルの入り口の10Å直上に位置するようになる7,9,10).基質タンパク質がユビキチン化されている部位まで解きほぐしが進み,チャネルの入り口の付近にユビキチン鎖がくると,これを切り離すことによりユビキチン鎖がATPaseチャネルに詰まることをふせいでいる(図2).実際に,N末端のみがユビキチン化される球状タンパク質のC末端に解きほぐしの開始を導く非構造領域を融合させ,この球状タンパク質にRpn11活性を欠失させたプロテアソームを作用させると,C末端から解きほぐしがはじまり,ひき続いて球状タンパク質の解きほぐしまでは起こるが,N末端のユビキチン鎖のところで解きほぐしは停止する7).
脱ユビキチン化酵素Rpn11の活性中心とユビキチン受容体Rpn10およびRpn13のユビキチン鎖結合領域との距離はそれぞれ75Åであり,テトラユビキチン鎖であればRpn10あるいはRpn13がユビキチン鎖を捕捉しつつRpn11が脱ユビキチン化を行うことのできる距離である.このことは,テトラユビキチン鎖が効率的な分解シグナルとなる機構と関係しているかもしれない.
基底部はRpt1~Rpt6のATPaseリングにRpn1,Rpn2,Rpn13が会合して形成される.Rpn1およびRpn2はユビキチン化されたタンパク質の分解を制御するタンパク質の足場としてはたらく.Rpn1はユビキチン様ドメインとの結合能をもち,ユビキチン化されたタンパク質を26SプロテアソームまでリクルートするhHR23やUbiquilinといったシャトルタンパク質や,ユビキチン鎖をトリミングして分解を遅延させる脱ユビキチン化酵素Usp14/Ubp6,プロテアソームにおいてユビキチン鎖の伸長反応を行うユビキチンリガーゼなどをリクルートする.一方,Rpn2はユビキチン受容体Rpn13の結合サブユニットとしてはたらいており,このRpn2およびRpn13は26Sプロテアソームの頂上の周辺部に位置している4,11)(図3).Rpn13にはさらに脱ユビキチン化酵素であるUCH37が会合する.このように,基底部においてはユビキチン化されたタンパク質をリクルートしつつ,そのユビキチン鎖の長さを制御するタンパク質が会合する,いわばタンパク質分解の制御の場となっている可能性が考えられる.
蓋部という名称は,プロテアソームの低解像度の電子顕微鏡像において“ふた”のようにみえる部分に相当するだろうとの予想から命名されたものが,クライオ電子顕微鏡により判明した蓋部の構造は意外なものであった.Rpn9,Rpn5,Rpn6,Rpn7,Rpn3,Rpn12は,互いのC末端側のPCIドメインを介して順に蹄鉄状に会合し,そこからおのおののN末端側を指を広げたように放射状に伸ばしている6)(図3).MPNドメインをもつRpn8とRpn11はヘテロ二量体を形成し,蓋部を構成するサブユニットのC末端に存在するヘリックスを束ねるようにして会合している.蓋部と基底部はおのおの独立して形成されたのちに会合して19S制御粒子になるが,蓋部は基底部にふたのようにおおいかぶさるのではなく,基底部の側面をおおうように張りつき,Rpn11を適切な場所に配置するとともに,Rpn5およびRpn6のN末端が20Sコア粒子のα1およびα2と直接に相互作用することにより,20Sコア粒子と19S制御粒子との結合を補強していることが明らかになった4,11)(図3).
以上のように,おのおののサブユニットのはたらきの分子機構が明らかになりつつあり,今後,プロテアソームを標的とした創薬や,機能未知の多くのサブユニットの役割について,大きなヒントをあたえるきっかけになると考えられる.今後は,さらに詳細な構造を明らかにするとともに,プロテアソームと会合するタンパク質の作用機構を明らかにするため,それらと26Sプロテアソームとの複合体の構造の解明が待たれる.
プロテアソームの作動機構は明らかになりつつあるが,プロテアソームの量や機能がどのように制御されているのかについてはほとんど解明されていないのが実情である.しかし,プロテアソームの量や活性の増減と老化あるいはヒトの疾患との関連が知られはじめ,プロテアソームの量的な制御と生理および疾患との関連が重要になってきた.
老化において観察される現象として,ゲノムの不安定性,エピジェネティックな変化,栄養感知の異常,ミトコンドリアの機能異常,幹細胞の機能低下などがあるが,異常タンパク質の蓄積も老化の主要な特徴のひとつである.実際に,タンパク質の凝集は神経変性疾患をはじめとした加齢に随伴する多くの疾患に観察される13).
いうまでもなく,プロテアソームは細胞におけるタンパク質の恒常性の維持において主要な役割をはたしており,老化への関与は十分に予想されるところである.実際に,さまざまな生物においてプロテアソームの機能が加齢とともに低下することが観察されており,プロテアソームの機能を人為的に低下させると,加齢にともなうタンパク質の凝集の出現頻度が上昇し,神経変性や代謝異常を生じることも報告されている14).一方,げっ歯類としては異例に長い寿命をもつハダカデバネズミは高いプロテアソーム活性を持つこと,ヒトに由来する健康長寿の線維芽細胞が若年者なみのプロテアソーム活性を保持していることなどから,プロテアソームの機能と老化との関連性は強く示唆される15).それでは,人為的にプロテアソームの活性を上昇させれば寿命は延びるのだろうか? これまで実際に,出芽酵母,線虫,ショウジョウバエ,ヒト細胞において,この推測が正しいことを支持する結果が得られている.
出芽酵母では転写因子Rpn4(もともと,プロテアソームのサブユニットと誤認されて同定されたので,この名がついている)がプロテアソームのすべてのサブユニットの遺伝子を一斉に転写することが知られているが,このRpn4を増加させた出芽酵母では分裂寿命が1.5倍以上も延長する16).Rpn4はプロテアソームの遺伝子のみならずほかの遺伝子の転写も亢進するが,寿命延長へ関与の知られるTorあるいはSir2や抗ストレス転写因子であるYap1はRpn4の増加による寿命延長には関与しておらず,プロテアソームの量の増大が必要十分であることが示されている.
多細胞生物においてはプロテアソームの増大が寿命を延長させることが直接的に示されている.ショウジョウバエではRpn11を単独で過剰に発現させることによりプロテアソーム活性が上昇し,その結果,寿命が延長するとともに加齢にともなう変性タンパク質の蓄積による神経細胞死も抑制された17).プロテアソームは33種類のサブユニットが集合して形成されているので,単一のサブユニットの過剰発現によりプロテアソーム活性が上昇することは不思議であるが,少なくとも,Rpn11がプロテアソームの外ではたらいているという報告はなく,プロテアソームの形成をなんらかの機構により促進していると推測される.プロテアソームの単一サブユニットの過剰発現によるプロテアソーム活性の増加と寿命延長は,線虫においても観察されている.線虫ではFOXOファミリー転写因子であるDAF-16に依存的なRpn6の発現上昇が寿命延長にはたらいていることが示された.Rpn6単独の過剰発現でもプロテアソーム活性が上昇して異常タンパク質の分解が亢進し,酸化ストレスや熱ストレスなどタンパク質ストレスのもとでの寿命延長をもたらす18).
ショウジョウバエや線虫において,なぜ単一のサブユニットの過剰発現がプロテアソームの機能を上昇させるのか,その機構は不明であるが,単一のサブユニットやプロテアソーム関連遺伝子の高発現によりプロテアソームの機能が亢進する例はほかにも知られている.マウスの受精初期胚では1細胞期から4細胞期の期間だけにZPACというプロテアソーム形成シャペロンを発現してプロテアソームを増産することによりタンパク質レベルでの母性-胚性転移を促進していること19),ヒトの骨髄間質細胞ではβ5を過剰発現させることによりプロテアソーム活性を上昇させ多分化能を維持していること20),ES細胞およびiPS細胞は多能性を維持するためFOXO4を介してRpn6の発現を上昇させていること21),が報告されており,細胞や組織の性質によりプロテアソームの形成における律速段階など,プロテアソームの増産の戦略が異なっているのかもしれない.
プロテアソームが正常に組み立てられて機能するためには,33種類すべてのサブユニット,および,それらの集合を助ける特異的な分子シャペロンが協調して発現する必要がある.
さきに述べたとおり,出芽酵母ではRpn4がプロテアソームの遺伝子の転写を活性化することが知られている.Rpn4はプロテアソームの機能が正常のときはユビキチン化に依存的および非依存的な経路によりたえず分解されているが,プロテアソームの機能が低下すると分解をまぬがれ,PACE配列を上流にもつプロテアソーム関連遺伝子の転写を一斉に誘導する.Rpn4を欠損した出芽酵母では野生型と比べプロテアソームの量がいちじるしく低下することから,Rpn4は誘導性の発現とともに基礎発現量も制御していることがわかる.
高等動物においても同様のフィードバック機構の存在することが知られている.1次配列においてRpn4と相同なタンパク質はみつかっていないが,酸化ストレス応答にはたらく転写活性化タンパク質として知られるNrf1は,プロテアソームの遺伝子の誘導性の発現にもはたらいていることが明らかになった22).Nrf1はN末端側に膜貫通領域をもつ小胞体膜タンパク質であり,通常は小胞体関連分解により恒常的にプロテアソームにより分解され転写活性を発揮しないようにしている.しかし,プロテアソームの機能が低下したり,ある種のストレスに細胞がさらされたりすると,Nrf1の分解は阻害されプロセシングをうけて核へと移行することによりプロテアソームの遺伝子の発現を誘導する23).このNrf1のプロセシング酵素はプロテアソーム自体であるとされている24).Nrf1を欠損した細胞ではプロテアソームの基礎発現量はほぼ正常であり,Nrf1は主としてプロテアソームの量のフィードバック制御にはたらいており,基礎的な発現を制御するほかの分子機構の存在が考えられる.しかし,ニューロンに特異的なNrf1ノックアウトマウスはプロテアソームの機能の低下を介して変性タンパク質の蓄積と神経変性をひき起こすことから,Nrf1によるプロテアソームの遺伝子の転写制御は個体の恒常性の維持において重要なはたらきをしていることがわかる25,26).近年,抗がん剤としてプロテアソーム阻害剤が使用されているが,腫瘍細胞はNrf1の活性化によるフィードバック機構によりプロテアソームの発現を亢進させることがプロテアソーム阻害剤に対する耐性の原因になっていると考えられている.Nrf1の誘導性の発現を同時に制御することができれば,この耐性機構の克服が可能になると期待される.
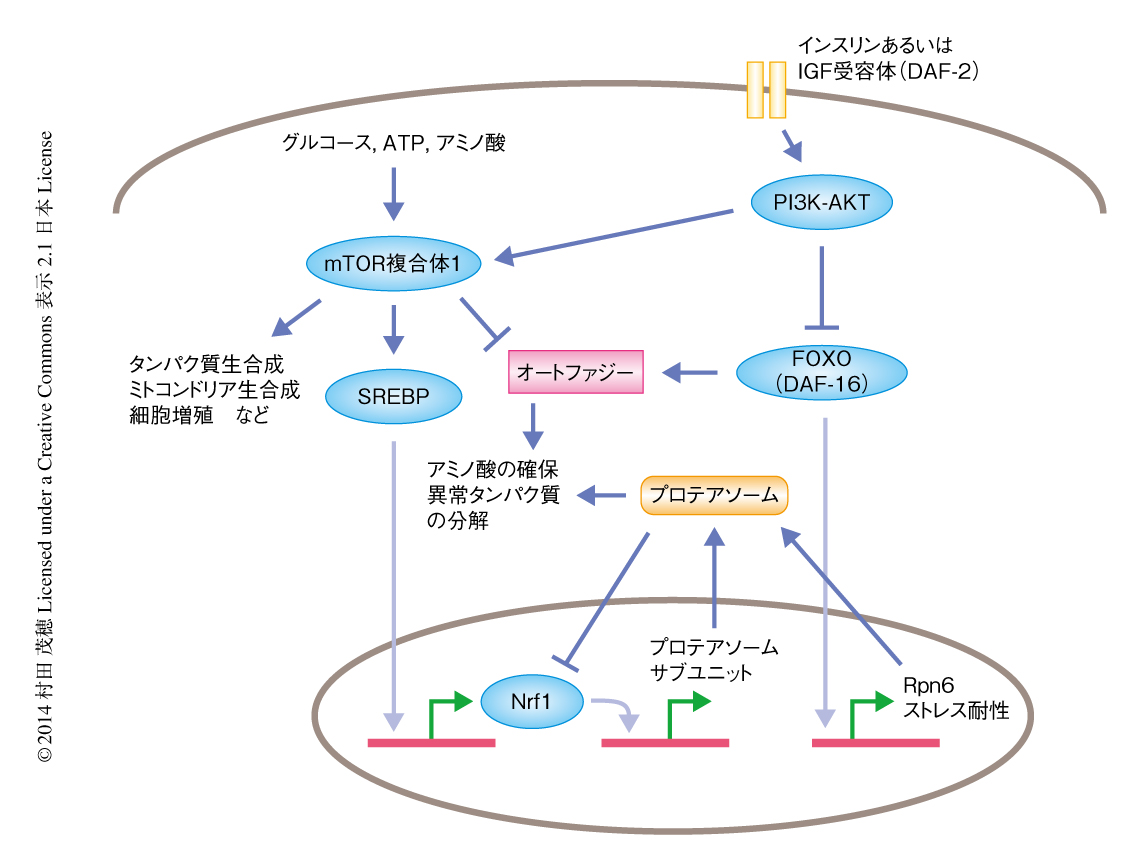
細胞環境,とくに,栄養状態に応答したプロテアソームの発現の制御機構についてもわかりはじめた.mTOR複合体1は富栄養の状態や成長因子に応答して活性化され,タンパク質や脂質の生合成をはじめとした代謝の同化作用を促進し細胞の成長や増殖を正に制御するマスター制御タンパク質として知られている.しかし同時に,プロテアソームを増加させることによりタンパク質分解を促進し,細胞におけるアミノ酸プールを増大させていることがわかった27).mTOR複合体1が脂質生合成のマスター転写因子であるSREBPを活性化させることは以前より知られていたが,SREBPはNrf1の転写を亢進させることによりプロテアソームの遺伝子の転写を上昇させている(図4).プロテアソームによるタンパク質分解が細胞におけるアミノ酸プールの維持に重要であることは以前から示されているが28,29),飢餓の際にアミノ酸の確保を行うタンパク質分解系であるオートファジーがmTOR複合体1により抑制されることを考えると,細胞における2大分解系の使い分けの対比が興味深い.
PI3K-AKTシグナル伝達経路により正に制御されるmTOR複合体1とは反対に,FOXOはPI3K-AKTシグナル伝達経路により負に制御される転写因子であり,栄養飢餓やさまざまなストレスに対応して恒常性の維持にはたらき,オートファジーをはじめとした異化作用やタンパク質恒常性の維持機構を発動させる.さきに述べたように,線虫およびES細胞ではDAF16あるいはFOXO4を介してRpn6の転写を上昇させることによりプロテアソームの機能を亢進させ,異常タンパク質の出現による毒性を軽減させている18,21)(図4).
mTOR複合体1は老化の促進および細胞増殖の促進に作用し,FOXOはその逆に作用するにもかかわらず,どちらもプロテアソームの発現を誘導することはいっけん奇異にうつる.しかし,プロテアソームがユビキチン化の目印にしたがってどのタンパク質を分解するかが細胞機能を制御する要点であると考えると,いずれの場合も,プロテアソームとともに誘導されているユビキチンリガーゼとその特異的な基質タンパク質が生理作用の理解の鍵になるであろう.筋萎縮の際にFOXO3により誘導されるユビキチンリガーゼMuRF1やMAFbx/atrogin-1などが好例である.富栄養のときおよび細胞増殖のときに活発に分解されてアミノ酸の供給源となるタンパク質とはいったい何であるのか,興味深い問題である.
プロテアソームについて,ここ数年で大きな進展をみせた領域にしぼり解説した.そのほかの重要なトピックスであるプロテアソームの多様性,プロテアソームと会合してプロテアソームの機能を制御するタンパク質,プロテアソームの分子集合については,ほかを参照されたい30).
20Sコア粒子の活性部位を阻害するプロテアソーム阻害剤が多発性骨髄腫に対する効果的な抗がん剤として用いられるようになって以来,プロテアソームは有望な創薬標的と考えられるようになってきた.プロテアソームの動作機構が明らかになるにつれて,新しい作用点をもつ阻害剤も開発されつつある.たとえば,Rpn13によるユビキチン鎖の認識に対する阻害剤が抗がん剤として有望であることも報告されている31).その反対に,プロテアソーム会合型の脱ユビキチン化酵素Usp14を阻害してプロテアソームによる分解を亢進させることにより,神経変性疾患の治療として細胞における異常タンパク質を分解しようとする試みもなされている32).
発見から25年を経過するいまなお,プロテアソームの新しい生理作用が発見されつづけており,その関与する領域は拡大をつづけている.今後も,老化,がん,免疫をキーワードに,プロテアソームの重要性はいっそう増してくると考えられる.
略歴:2000年 東京大学大学院医学系研究科 修了,東京都臨床医学総合研究所 研究員,科学技術振興機構 さきがけ研究員を経て,2007年より東京大学大学院薬学系研究科 教授.
研究テーマ:プロテアソームの機能の異常と疾患,ユビキチン-プロテアソーム系によるタンパク質恒常性の維持機構,胸腺プロテアソームによるT細胞の選択機構.
抱負:プロテアソームの機能を健全に保つことによる健康長寿は可能か? に挑戦したい.
研究室URL:http://www.f.u-tokyo.ac.jp/~tanpaku/
© 2014 村田 茂穂 Licensed under CC 表示 2.1 日本
(東京大学大学院薬学系研究科 蛋白質代謝学教室)
email:村田茂穂
領域融合レビュー, 3, e011 (2014) DOI: 10.7875/leading.author.3.e011
Shigeo Murata: A new understanding of action mechanism, physiological roles and regulation of the proteasome.
要 約
26Sプロテアソームは約2.5 MDa,66個のサブユニットから構成される,真核生物に必須の巨大なタンパク質複合体である.なぜこのように複雑な構造をもっているのか,どのようにタンパク質の分解を実行しているのか,いまだよくわかっていない.しかし近年,クライオ電子顕微鏡を用いた高い分解能での構造解析から大きな進展がみられている.また,プロテアソームの量の多寡とがん,神経変性,老化などヒトの疾患とのかかわりについての報告も蓄積してきた.プロテアソームの発現がどのように制御されているのかについてもほとんど未解明であったが,細胞の内外の栄養環境やストレスに応答してプロテアソームの発現が制御され,寿命の延長,ストレス応答,幹細胞の機能維持に重要な役割をはたしていることがわかってきた.
はじめに
細胞は恒常性の維持,ストレス応答,機能変換などを実行するための手段のひとつとして,細胞内タンパク質分解を利用している.細胞において役割をおえた(おえるべき)タンパク質や不良なタンパク質の大半は,分解の目印としてユビキチンを付加されたのち,プロテアソームとよばれる巨大なタンパク質複合体により短いペプチド断片にまで消化される.ユビキチン-プロテアソーム系は真核生物において必須の細胞内タンパク質分解系であるが,その要点は,標的となるタンパク質の幅広さと高い選択性の両立,および,無秩序ではなく厳密に制御された分解にある.筆者が大学院生だった1990年代後半はユビキチン-プロテアソーム系の具体的な重要性がわかりはじめたばかりのころであったが,そののちの研究の進展はめざましく,細胞周期,遺伝子発現,シグナル伝達をはじめとした幅広い生命現象において必須の制御機構としてはたらいていることが明らかになり,近年の生命科学を理解するうえで必須の研究分野である.
ユビキチン-プロテアソーム系においていまだ大きな謎につつまれているのが,タンパク質の分解を実行するプロテアソームである.プロテアソームは多数のサブユニットから構築されるきわめて複雑なタンパク質複合体であるが,ここ数年の構造解析の進展により,その詳細な作動機構がわかってきた.また,プロテアソームの機能と老化との関連や,その発現の新しい制御機構も明らかになりつつある.このレビューでは,とくにここ数年で明らかになってきたプロテアソームの作動機構と生理作用を中心に解説する.
1.プロテアソームの基礎知識
プロテアソームは酵母から哺乳類にいたるすべての真核生物において高度に保存された構造をもつタンパク質複合体である.ユビキチン化されたタンパク質を分解するのは,プロテアーゼ活性をもつ20Sコア粒子(CP,あるいは,20Sプロテアソームともよぶ)の両端または片側に19S制御粒子(RPともよぶ)が会合した26Sプロテアソームである.これは,長さ44 nm,最大直径20 nm,2.5 MDaにも及ぶ巨大な酵素である(図1).
20Sコア粒子はX線結晶構造解析がなされており,構造の詳細が判明している1).7種類のサブユニットα1~α7からなるαリングと,7種類のサブユニットβ1~β7からなるβリングがαββαの順に積み重なった中空の樽状の形状をしており,βリングの内壁にプロテアーゼ活性中心が露出している.β1,β2,β5のみがプロテアーゼ活性をもつ.脊椎動物においては,酵母から保存されている構成的に発現するタイプの20Sコア粒子のほか,インターフェロンγに応答して発現が誘導されるβ1i,β2i,β5iというプロテアーゼ活性をもつ異なるサブユニットが組み込まれた免疫型20Sコア粒子,この免疫型20Sコア粒子からさらにβ5iが胸腺皮質に特異的に発現するサブユニットβ5tと置換した胸腺型20Sコア粒子が存在し,これらはMHCクラスIを介した免疫応答においてきわめて重要なはたらきをしていることが知られているが,詳細についてはほかにゆずる2).
20Sコア粒子が単独のときには入り口となるαリングの中央は閉じており,開口しても13Åの狭いチャネルとなり,解きほぐされた(アンフォールディングされた)タンパク質のみが通過できる.通過した基質タンパク質はαリングとβリングから形成されるantechamber(控えの間)とよばれる空間において解きほぐされた状態で維持され,さらに,2つのβリングにかこまれたcatalytic chamber(触媒空間)に移動して分解される.
20Sコア粒子における分解のために基質タンパク質の前処理を行うのが19S制御粒子である.ATPase活性をもつ6種類のサブユニットRpt1~Rpt6からなるATPaseリングに, ATPase活性をもたない13種類のサブユニットRpn1~Rpn3,Rpn5~Rpn13,Rpn15が会合したタンパク質複合体であり,生化学的な性質やその形成機構から,Rpt1~Rpt6,Rpn1,Rpn2,Rpn13からなる基底部(base)と,Rpn3,Rpn5~Rpn9,Rpn11,Rpn12,Rpn15からなる蓋部(lid)の2つに区分することができる3)(図1).基底部と蓋部は独立して形成されたのちに会合して19S制御粒子ができあがるが,Rpn10は完成した19S制御粒子に会合する.
これまで生化学的および遺伝学的な解析から,いくつかのサブユニットについてはユビキチン化されたタンパク質の分解における役割が知られている.ATPaseサブユニットは基質タンパク質の解きほぐし,20Sコア粒子のαリングの開口,20Sコア粒子への基質タンパク質の送り込みを行う.Rpn10およびRpn13はユビキチン鎖を直接に捕捉する“ユビキチン受容体”としてはたらく.Rpn11はZn2+メタロプロテアーゼであり,ユビキチン鎖の根元を切断することにより基質タンパク質からユビキチン鎖を切り離し分解を促進する.
これらの多数のサブユニットがどのように有機的に連携してユビキチン化されたタンパク質の分解を可能にしているのかを理解するため,19S制御粒子の構造解析がながらく待望されていたが,構造の複雑さ,不均一さから困難をきわめていた.しかし,ここ数年のクライオ電子顕微鏡による単粒子解析技術の進歩により,26Sプロテアソーム全体の高次構造が7~10Åの高分解能で明らかになり,プロテアソームの作動機構の理解が飛躍的に進展した4-11).
2.プロテアソームの作動の分子機構
クライオ電子顕微鏡による解析を中心に,個別のサブユニットや類似したタンパク質の結晶構造解析の情報やこれまでの生化学的な解析の蓄積を参照することにより,19S制御粒子におけるおのおののサブユニットの配置や活性中心の位置,ATP加水分解にともなう構造変化をはじめ,19S制御粒子が非常に巧妙なしくみでユビキチン化されたタンパク質を処理している機構がわかってきた.
ATPaseサブユニットはRpt1-Rpt2-Rpt6-Rpt3-Rpt4-Rpt5の順にならんでリング構造を形成し,その中央にはチャネルが形成される.ほかのAAA+アンフォールダーゼと同様に,基質を捕捉するAr-Φループ(Ar-Φ:aromatic hydrophobic,芳香性および疎水性)とよばれる疎水性に富んだ領域がおのおののサブユニットからチャネルへむけて突き出している.ATPの存在下(基質タンパク質と会合していない状態を模倣)では,Ar-ΦループはRpt3を頂点,Rpt2を底とし,Rpt6がそのあいだを連結するらせん階段状に配列し,さらに,ATPaseリングのチャネルの軸は20Sコア粒子のチャネルの軸から傾いている.しかし,非加水分解性のATPアナログであるATPγSの存在下(基質タンパク質と会合した状態を模倣)では,おのおののAr-Φループはほぼ同一の平面にそろい,ATPaseリングのチャネルの軸と20Sコア粒子のチャネルの軸が一致する5)(図2).このように,ATPの加水分解サイクルにともなう立体構造の変化により,基質タンパク質の高次構造を解きほぐしながら20Sコア粒子へと送り込んでいる機構がわかってきた.
ATPaseリングは20Sコア粒子のαリングと直接に会合する.Rpt2,Rpt3,Rpt5のC末端に存在するHbYX配列(疎水性アミノ酸-チロシン-任意のアミノ酸からなる配列)がαサブユニットのあいだにあるポケットに突き刺さることにより,αリングに構造変化が起こり開口し,解きほぐされた基質タンパク質が20Sコア粒子へと送り込まれる.
ユビキチン化されたタンパク質の捕捉はRpn10およびRpn13により行われる.これら“ユビキチン受容体”はアンテナのように19S制御粒子の頂上の周辺部に互いに90Å離れて位置している(図3).これまでの生化学的な解析から,4つ以上のユビキチンが連なったテトラユビキチン鎖で修飾されていることが基質タンパク質がプロテアソームにより効率的に分解されるために必要であることが知られている.2つのユビキチン受容体の距離はちょうどテトラユビキチン鎖の長さに相当することから,それらの協調により高い親和性でユビキチン鎖を捕捉している可能性が示唆されている.
しかし,ユビキチン鎖のみではプロテアソームによる分解には十分ではなく,基質タンパク質が30アミノ酸残基以上の長さの非構造領域をもつことが必要であり,この領域がATPaseチャネルに入り込みAr-Φループにより捕捉されることにより,はじめて分解への運命が決定づけられる12).
脱ユビキチン化酵素Rpn11はユビキチン鎖の根元を基質タンパク質から切り離す.このはたらきはユビキチン化されたタンパク質の分解に必須の過程であるが,ATPの加水分解に依存的に基質タンパク質の解きほぐしが開始されてはじめて実行される.すなわち,ATPaseリングの立体構造の変化は19S制御粒子の全体の構造変化をともない,基質タンパク質の解きほぐしのときには,あたかも解きほぐされつつあるポリペプチド鎖を間近で見張るかのように,Rpn11の活性中心はATPaseリングのチャネルの入り口の10Å直上に位置するようになる7,9,10).基質タンパク質がユビキチン化されている部位まで解きほぐしが進み,チャネルの入り口の付近にユビキチン鎖がくると,これを切り離すことによりユビキチン鎖がATPaseチャネルに詰まることをふせいでいる(図2).実際に,N末端のみがユビキチン化される球状タンパク質のC末端に解きほぐしの開始を導く非構造領域を融合させ,この球状タンパク質にRpn11活性を欠失させたプロテアソームを作用させると,C末端から解きほぐしがはじまり,ひき続いて球状タンパク質の解きほぐしまでは起こるが,N末端のユビキチン鎖のところで解きほぐしは停止する7).
脱ユビキチン化酵素Rpn11の活性中心とユビキチン受容体Rpn10およびRpn13のユビキチン鎖結合領域との距離はそれぞれ75Åであり,テトラユビキチン鎖であればRpn10あるいはRpn13がユビキチン鎖を捕捉しつつRpn11が脱ユビキチン化を行うことのできる距離である.このことは,テトラユビキチン鎖が効率的な分解シグナルとなる機構と関係しているかもしれない.
基底部はRpt1~Rpt6のATPaseリングにRpn1,Rpn2,Rpn13が会合して形成される.Rpn1およびRpn2はユビキチン化されたタンパク質の分解を制御するタンパク質の足場としてはたらく.Rpn1はユビキチン様ドメインとの結合能をもち,ユビキチン化されたタンパク質を26SプロテアソームまでリクルートするhHR23やUbiquilinといったシャトルタンパク質や,ユビキチン鎖をトリミングして分解を遅延させる脱ユビキチン化酵素Usp14/Ubp6,プロテアソームにおいてユビキチン鎖の伸長反応を行うユビキチンリガーゼなどをリクルートする.一方,Rpn2はユビキチン受容体Rpn13の結合サブユニットとしてはたらいており,このRpn2およびRpn13は26Sプロテアソームの頂上の周辺部に位置している4,11)(図3).Rpn13にはさらに脱ユビキチン化酵素であるUCH37が会合する.このように,基底部においてはユビキチン化されたタンパク質をリクルートしつつ,そのユビキチン鎖の長さを制御するタンパク質が会合する,いわばタンパク質分解の制御の場となっている可能性が考えられる.
蓋部という名称は,プロテアソームの低解像度の電子顕微鏡像において“ふた”のようにみえる部分に相当するだろうとの予想から命名されたものが,クライオ電子顕微鏡により判明した蓋部の構造は意外なものであった.Rpn9,Rpn5,Rpn6,Rpn7,Rpn3,Rpn12は,互いのC末端側のPCIドメインを介して順に蹄鉄状に会合し,そこからおのおののN末端側を指を広げたように放射状に伸ばしている6)(図3).MPNドメインをもつRpn8とRpn11はヘテロ二量体を形成し,蓋部を構成するサブユニットのC末端に存在するヘリックスを束ねるようにして会合している.蓋部と基底部はおのおの独立して形成されたのちに会合して19S制御粒子になるが,蓋部は基底部にふたのようにおおいかぶさるのではなく,基底部の側面をおおうように張りつき,Rpn11を適切な場所に配置するとともに,Rpn5およびRpn6のN末端が20Sコア粒子のα1およびα2と直接に相互作用することにより,20Sコア粒子と19S制御粒子との結合を補強していることが明らかになった4,11)(図3).
以上のように,おのおののサブユニットのはたらきの分子機構が明らかになりつつあり,今後,プロテアソームを標的とした創薬や,機能未知の多くのサブユニットの役割について,大きなヒントをあたえるきっかけになると考えられる.今後は,さらに詳細な構造を明らかにするとともに,プロテアソームと会合するタンパク質の作用機構を明らかにするため,それらと26Sプロテアソームとの複合体の構造の解明が待たれる.
3.プロテアソームと老化および寿命
プロテアソームの作動機構は明らかになりつつあるが,プロテアソームの量や機能がどのように制御されているのかについてはほとんど解明されていないのが実情である.しかし,プロテアソームの量や活性の増減と老化あるいはヒトの疾患との関連が知られはじめ,プロテアソームの量的な制御と生理および疾患との関連が重要になってきた.
老化において観察される現象として,ゲノムの不安定性,エピジェネティックな変化,栄養感知の異常,ミトコンドリアの機能異常,幹細胞の機能低下などがあるが,異常タンパク質の蓄積も老化の主要な特徴のひとつである.実際に,タンパク質の凝集は神経変性疾患をはじめとした加齢に随伴する多くの疾患に観察される13).
いうまでもなく,プロテアソームは細胞におけるタンパク質の恒常性の維持において主要な役割をはたしており,老化への関与は十分に予想されるところである.実際に,さまざまな生物においてプロテアソームの機能が加齢とともに低下することが観察されており,プロテアソームの機能を人為的に低下させると,加齢にともなうタンパク質の凝集の出現頻度が上昇し,神経変性や代謝異常を生じることも報告されている14).一方,げっ歯類としては異例に長い寿命をもつハダカデバネズミは高いプロテアソーム活性を持つこと,ヒトに由来する健康長寿の線維芽細胞が若年者なみのプロテアソーム活性を保持していることなどから,プロテアソームの機能と老化との関連性は強く示唆される15).それでは,人為的にプロテアソームの活性を上昇させれば寿命は延びるのだろうか? これまで実際に,出芽酵母,線虫,ショウジョウバエ,ヒト細胞において,この推測が正しいことを支持する結果が得られている.
出芽酵母では転写因子Rpn4(もともと,プロテアソームのサブユニットと誤認されて同定されたので,この名がついている)がプロテアソームのすべてのサブユニットの遺伝子を一斉に転写することが知られているが,このRpn4を増加させた出芽酵母では分裂寿命が1.5倍以上も延長する16).Rpn4はプロテアソームの遺伝子のみならずほかの遺伝子の転写も亢進するが,寿命延長へ関与の知られるTorあるいはSir2や抗ストレス転写因子であるYap1はRpn4の増加による寿命延長には関与しておらず,プロテアソームの量の増大が必要十分であることが示されている.
多細胞生物においてはプロテアソームの増大が寿命を延長させることが直接的に示されている.ショウジョウバエではRpn11を単独で過剰に発現させることによりプロテアソーム活性が上昇し,その結果,寿命が延長するとともに加齢にともなう変性タンパク質の蓄積による神経細胞死も抑制された17).プロテアソームは33種類のサブユニットが集合して形成されているので,単一のサブユニットの過剰発現によりプロテアソーム活性が上昇することは不思議であるが,少なくとも,Rpn11がプロテアソームの外ではたらいているという報告はなく,プロテアソームの形成をなんらかの機構により促進していると推測される.プロテアソームの単一サブユニットの過剰発現によるプロテアソーム活性の増加と寿命延長は,線虫においても観察されている.線虫ではFOXOファミリー転写因子であるDAF-16に依存的なRpn6の発現上昇が寿命延長にはたらいていることが示された.Rpn6単独の過剰発現でもプロテアソーム活性が上昇して異常タンパク質の分解が亢進し,酸化ストレスや熱ストレスなどタンパク質ストレスのもとでの寿命延長をもたらす18).
ショウジョウバエや線虫において,なぜ単一のサブユニットの過剰発現がプロテアソームの機能を上昇させるのか,その機構は不明であるが,単一のサブユニットやプロテアソーム関連遺伝子の高発現によりプロテアソームの機能が亢進する例はほかにも知られている.マウスの受精初期胚では1細胞期から4細胞期の期間だけにZPACというプロテアソーム形成シャペロンを発現してプロテアソームを増産することによりタンパク質レベルでの母性-胚性転移を促進していること19),ヒトの骨髄間質細胞ではβ5を過剰発現させることによりプロテアソーム活性を上昇させ多分化能を維持していること20),ES細胞およびiPS細胞は多能性を維持するためFOXO4を介してRpn6の発現を上昇させていること21),が報告されており,細胞や組織の性質によりプロテアソームの形成における律速段階など,プロテアソームの増産の戦略が異なっているのかもしれない.
4.プロテアソームの発現の制御機構
プロテアソームが正常に組み立てられて機能するためには,33種類すべてのサブユニット,および,それらの集合を助ける特異的な分子シャペロンが協調して発現する必要がある.
さきに述べたとおり,出芽酵母ではRpn4がプロテアソームの遺伝子の転写を活性化することが知られている.Rpn4はプロテアソームの機能が正常のときはユビキチン化に依存的および非依存的な経路によりたえず分解されているが,プロテアソームの機能が低下すると分解をまぬがれ,PACE配列を上流にもつプロテアソーム関連遺伝子の転写を一斉に誘導する.Rpn4を欠損した出芽酵母では野生型と比べプロテアソームの量がいちじるしく低下することから,Rpn4は誘導性の発現とともに基礎発現量も制御していることがわかる.
高等動物においても同様のフィードバック機構の存在することが知られている.1次配列においてRpn4と相同なタンパク質はみつかっていないが,酸化ストレス応答にはたらく転写活性化タンパク質として知られるNrf1は,プロテアソームの遺伝子の誘導性の発現にもはたらいていることが明らかになった22).Nrf1はN末端側に膜貫通領域をもつ小胞体膜タンパク質であり,通常は小胞体関連分解により恒常的にプロテアソームにより分解され転写活性を発揮しないようにしている.しかし,プロテアソームの機能が低下したり,ある種のストレスに細胞がさらされたりすると,Nrf1の分解は阻害されプロセシングをうけて核へと移行することによりプロテアソームの遺伝子の発現を誘導する23).このNrf1のプロセシング酵素はプロテアソーム自体であるとされている24).Nrf1を欠損した細胞ではプロテアソームの基礎発現量はほぼ正常であり,Nrf1は主としてプロテアソームの量のフィードバック制御にはたらいており,基礎的な発現を制御するほかの分子機構の存在が考えられる.しかし,ニューロンに特異的なNrf1ノックアウトマウスはプロテアソームの機能の低下を介して変性タンパク質の蓄積と神経変性をひき起こすことから,Nrf1によるプロテアソームの遺伝子の転写制御は個体の恒常性の維持において重要なはたらきをしていることがわかる25,26).近年,抗がん剤としてプロテアソーム阻害剤が使用されているが,腫瘍細胞はNrf1の活性化によるフィードバック機構によりプロテアソームの発現を亢進させることがプロテアソーム阻害剤に対する耐性の原因になっていると考えられている.Nrf1の誘導性の発現を同時に制御することができれば,この耐性機構の克服が可能になると期待される.
細胞環境,とくに,栄養状態に応答したプロテアソームの発現の制御機構についてもわかりはじめた.mTOR複合体1は富栄養の状態や成長因子に応答して活性化され,タンパク質や脂質の生合成をはじめとした代謝の同化作用を促進し細胞の成長や増殖を正に制御するマスター制御タンパク質として知られている.しかし同時に,プロテアソームを増加させることによりタンパク質分解を促進し,細胞におけるアミノ酸プールを増大させていることがわかった27).mTOR複合体1が脂質生合成のマスター転写因子であるSREBPを活性化させることは以前より知られていたが,SREBPはNrf1の転写を亢進させることによりプロテアソームの遺伝子の転写を上昇させている(図4).プロテアソームによるタンパク質分解が細胞におけるアミノ酸プールの維持に重要であることは以前から示されているが28,29),飢餓の際にアミノ酸の確保を行うタンパク質分解系であるオートファジーがmTOR複合体1により抑制されることを考えると,細胞における2大分解系の使い分けの対比が興味深い.
PI3K-AKTシグナル伝達経路により正に制御されるmTOR複合体1とは反対に,FOXOはPI3K-AKTシグナル伝達経路により負に制御される転写因子であり,栄養飢餓やさまざまなストレスに対応して恒常性の維持にはたらき,オートファジーをはじめとした異化作用やタンパク質恒常性の維持機構を発動させる.さきに述べたように,線虫およびES細胞ではDAF16あるいはFOXO4を介してRpn6の転写を上昇させることによりプロテアソームの機能を亢進させ,異常タンパク質の出現による毒性を軽減させている18,21)(図4).
mTOR複合体1は老化の促進および細胞増殖の促進に作用し,FOXOはその逆に作用するにもかかわらず,どちらもプロテアソームの発現を誘導することはいっけん奇異にうつる.しかし,プロテアソームがユビキチン化の目印にしたがってどのタンパク質を分解するかが細胞機能を制御する要点であると考えると,いずれの場合も,プロテアソームとともに誘導されているユビキチンリガーゼとその特異的な基質タンパク質が生理作用の理解の鍵になるであろう.筋萎縮の際にFOXO3により誘導されるユビキチンリガーゼMuRF1やMAFbx/atrogin-1などが好例である.富栄養のときおよび細胞増殖のときに活発に分解されてアミノ酸の供給源となるタンパク質とはいったい何であるのか,興味深い問題である.
おわりに
プロテアソームについて,ここ数年で大きな進展をみせた領域にしぼり解説した.そのほかの重要なトピックスであるプロテアソームの多様性,プロテアソームと会合してプロテアソームの機能を制御するタンパク質,プロテアソームの分子集合については,ほかを参照されたい30).
20Sコア粒子の活性部位を阻害するプロテアソーム阻害剤が多発性骨髄腫に対する効果的な抗がん剤として用いられるようになって以来,プロテアソームは有望な創薬標的と考えられるようになってきた.プロテアソームの動作機構が明らかになるにつれて,新しい作用点をもつ阻害剤も開発されつつある.たとえば,Rpn13によるユビキチン鎖の認識に対する阻害剤が抗がん剤として有望であることも報告されている31).その反対に,プロテアソーム会合型の脱ユビキチン化酵素Usp14を阻害してプロテアソームによる分解を亢進させることにより,神経変性疾患の治療として細胞における異常タンパク質を分解しようとする試みもなされている32).
発見から25年を経過するいまなお,プロテアソームの新しい生理作用が発見されつづけており,その関与する領域は拡大をつづけている.今後も,老化,がん,免疫をキーワードに,プロテアソームの重要性はいっそう増してくると考えられる.
文 献
- Borissenko, L. & Groll, M.: 20S proteasome and its inhibitors: crystallographic knowledge for drug development. Chem. Rev., 107, 687-717 (2007)[PubMed]
- Kniepert, A. & Groettrup, M.: The unique functions of tissue-specific proteasomes. Trends Biochem. Sci., 39, 17-24 (2014)[PubMed]
- Murata, S., Yashiroda, H. & Tanaka, K.: Molecular mechanisms of proteasome assembly. Nat. Rev. Mol. Cell Biol., 10, 104-115 (2009)[PubMed]
- Beck, F., Unverdorben, P., Bohn, S. et al.: Near-atomic resolution structural model of the yeast 26S proteasome. Proc. Natl. Acad. Sci. USA, 109, 14870-14875 (2012)[PubMed]
- Beckwith, R., Estrin, E., Worden, E. J. et al.: Reconstitution of the 26S proteasome reveals functional asymmetries in its AAA+ unfoldase. Nat. Struct. Mol. Biol., 20, 1164-1172 (2013)[PubMed]
- Estrin, E., Lopez-Blanco, J. R., Chacon, P. et al.: Formation of an intricate helical bundle dictates the assembly of the 26S proteasome lid. Structure, 21, 1624-1635 (2013)[PubMed]
- Matyskiela, M. E., Lander, G. C. & Martin, A.: Conformational switching of the 26S proteasome enables substrate degradation. Nat. Struct. Mol. Biol., 20, 781-788 (2013)[PubMed]
- Pathare, G. R., Nagy, I., Sledz, P. et al.: Crystal structure of the proteasomal deubiquitylation module Rpn8-Rpn11. Proc. Natl. Acad. Sci. USA, 111, 2984-2989 (2014)[PubMed]
- Unverdorben, P., Beck, F., Sledz, P. et al.: Deep classification of a large cryo-EM dataset defines the conformational landscape of the 26S proteasome. Proc. Natl. Acad. Sci. USA, 111, 5544-5549 (2014)[PubMed]
- Worden, E. J., Padovani, C. & Martin, A.: Structure of the Rpn11-Rpn8 dimer reveals mechanisms of substrate deubiquitination during proteasomal degradation. Nat. Struct. Mol. Biol., 21, 220-227 (2014)[PubMed]
- Lander, G. C., Estrin, E., Matyskiela, M. E. et al.: Complete subunit architecture of the proteasome regulatory particle. Nature, 482, 186-191 (2012)[PubMed]
- Bhattacharyya, S., Yu, H., Mim, C. et al.: Regulated protein turnover: snapshots of the proteasome in action. Nat. Rev. Mol. Cell Biol., 15, 122-133 (2014)[PubMed]
- Lopez-Otin, C., Blasco, M. A., Partridge, L. et al.: The hallmarks of aging. Cell, 153, 1194-217 (2013)[PubMed]
- Tomaru, U., Takahashi, S., Ishizu, A. et al.: Decreased proteasomal activity causes age-related phenotypes and promotes the development of metabolic abnormalities. Am. J. Pathol., 180, 963-972 (2012)[PubMed]
- Rodriguez, K. A., Edrey, Y. H., Osmulski, P. et al.: Altered composition of liver proteasome assemblies contributes to enhanced proteasome activity in the exceptionally long-lived naked mole-rat. PLoS One, 7, e35890 (2012)[PubMed]
- Kruegel, U., Robison, B., Dange, T. et al.: Elevated proteasome capacity extends replicative lifespan in Saccharomyces cerevisiae. PLoS Genet., 7, e1002253 (2011)[PubMed]
- Tonoki, A., Kuranaga, E., Tomioka, T. et al.: Genetic evidence linking age-dependent attenuation of the 26S proteasome with the aging process. Mol. Cell. Biol., 29, 1095-1106 (2009)[PubMed]
- Vilchez, D., Morantte, I., Liu, Z. et al.: RPN-6 determines C. elegans longevity under proteotoxic stress conditions. Nature, 489, 263-268 (2012)[PubMed]
- Shin, S. -W., Shimizu, N., Tokoro, M. et al.: Mouse zygote-specific proteasome assembly chaperone important for maternal-to-zygotic transition. Biol. Open, 2, 170-182 (2013)[PubMed]
- Lu, L., Song, H. -F., Wei, J. -L. et al.: Ameliorating replicative senescence of human bone marrow stromal cells by PSMB5 overexpression. Biochem. Biophys. Res. Commun., 443, 1182-1188 (2014)[PubMed]
- Vilchez, D., Boyer, L., Morantte, I. et al.: Increased proteasome activity in human embryonic stem cells is regulated by PSMD11. Nature, 489, 304-308 (2012)[PubMed]
- Radhakrishnan, S. K., Lee, C. S., Young, P. et al.: Transcription factor Nrf1 mediates the proteasome recovery pathway after proteasome inhibition in mammalian cells. Mol. Cell, 38, 17-28 (2011)[PubMed]
- Steffen, J., Seeger, M., Koch, A. et al.: Proteasomal degradation is transcriptionally controlled by TCF11 via an ERAD-dependent feedback loop. Mol. Cell, 40, 147-158 (2010)[PubMed]
- Sha, Z. & Goldberg, A. L.: Proteasome-mediated processing of Nrf1 is essential for coordinate induction of all proteasome subunits and p97. Curr. Biol., 24, 1-11 (2014)[PubMed]
- Lee, C. S., Lee, C., Hu, T. et al.: Loss of nuclear factor E2-related factor 1 in the brain leads to dysregulation of proteasome gene expression and neurodegeneration. Proc. Natl. Acad. Sci. USA, 108, 8408-8413 (2011)[PubMed]
- Kobayashi, A., Tsukide, T., Miyasaka, T. et al.: Central nervous system-specific deletion of transcription factor Nrf1 causes progressive motor neuronal dysfunction. Genes Cells, 16, 692-703 (2011)[PubMed]
- Kwiatkowski, D. J., Manning, B. D., Zhang, Y. et al.: Coordinated regulation of protein synthesis and degradation by mTORC1. Nature, 513, 440-443 (2014)[PubMed]
- Vabulas, R. M. & Hartl, F. U.: Protein synthesis upon acute nutrient restriction relies on proteasome function. Science, 310, 1960-1963 (2005)[PubMed]
- Suraweera, A., Mu, C., Hanssum, A. et al.: Failure of amino acid homeostasis causes cell death following proteasome inhibition. Mol. Cell, 48, 242-253 (2012)[PubMed]
- 村田茂穂, 反町洋之 (編): タンパク質分解系による生体制御. 実験医学, 29, 1830-2072 (2011)
- Anchoori, R. K., Karanam, B., Peng, S. et al.: A bis-benzylidine piperidone targeting proteasome ubiquitin receptor RPN13/ADRM1 as a therapy for cancer. Cancer Cell, 24, 791-805 (2013)[PubMed]
- Lee, B. -H., Lee, M. J., Park, S. et al.: Enhancement of proteasome activity by a small-molecule inhibitor of USP14. Nature, 467, 179-184 (2010)[PubMed]
著者プロフィール
略歴:2000年 東京大学大学院医学系研究科 修了,東京都臨床医学総合研究所 研究員,科学技術振興機構 さきがけ研究員を経て,2007年より東京大学大学院薬学系研究科 教授.
研究テーマ:プロテアソームの機能の異常と疾患,ユビキチン-プロテアソーム系によるタンパク質恒常性の維持機構,胸腺プロテアソームによるT細胞の選択機構.
抱負:プロテアソームの機能を健全に保つことによる健康長寿は可能か? に挑戦したい.
研究室URL:http://www.f.u-tokyo.ac.jp/~tanpaku/
© 2014 村田 茂穂 Licensed under CC 表示 2.1 日本