花の発生における幹細胞の増殖および分化の制御
2014/12/11
伊藤 寿朗
(シンガポールTemasek Life Sciences Laboratory)
email:伊藤寿朗
領域融合レビュー, 3, e014 (2014) DOI: 10.7875/leading.author.3.e014
Toshiro Ito: Growth and differentiation control in Arabidopsis flower development.

花の器官形成におけるABCモデルが提唱されてから20年あまりたつ.シロイヌナズナを中心としたモデル植物における遺伝学的な解析からはじまった花の発生の研究は,のちの分子遺伝学およびゲノム科学の進歩により大きく進展してきた.Aクラス,Bクラス,Cクラスの遺伝子がクローニングされ,これらのコードする転写因子の上流および下流における遺伝子制御ネットワークが明らかにされつつある.それらの転写因子はゲノムにおいて数千をこえる部位に結合し,数百をこえる遺伝子の発現を制御している.それらには,生殖細胞の誘導,花の器官形成や成熟において鍵となる転写因子やクロマチン制御タンパク質,植物ホルモンの合成,シグナル伝達タンパク質などをコードする遺伝子が含まれる.それぞれの遺伝子は花の発生の時間軸にそって特異的な細胞において発現し機能している.最近のエピジェネティクスの研究に着目しながら,花の幹細胞の増殖および分化における時間的および空間的に特異的な制御機構について論じる.
種子植物は生殖器官として“花”をもち,有性生殖の結果として種子を形成する.人類は数百万年の歴史のなかで花の産物である実や種子が食用に適することを見い出し,果実や穀物の生産性の向上をめざしそれらの植物を培ってきた.また,花はその美しさゆえ,古代より人々に愛されてきた.古今東西の遺跡や壁画,紋章などにおいて,花の絵柄は普遍的にみられ,美や生命力の象徴ともいえる.同時に,移ろいゆく四季折々を彩る花は,われわれ日本人の無常と“もののあはれ”の価値観の象徴でもある.実際に,すべての植物において花を咲かすことは幹細胞としての活性を停止することであり,これは幹細胞の死を意味する.時が来たならば自らの増殖を止め,次世代の種子をつくることに専心する花の生きざまに,われわれ日本人は生命の美しさと儚さを重ね合せるのかもしれない.
現代の植物科学の研究者には,分子遺伝学およびゲノム科学という強力なツールがあたえられている.これにより,花の成り立ちの基本原理や生命の美しさそのものを分子の言葉で読み解くことが可能になった.このレビューでは,シロイヌナズナにおける花の発生の研究のなかでも,とくに過去10年間の進歩を,エピジェネティクスを介した幹細胞の増殖の制御機構に着目して論じる.
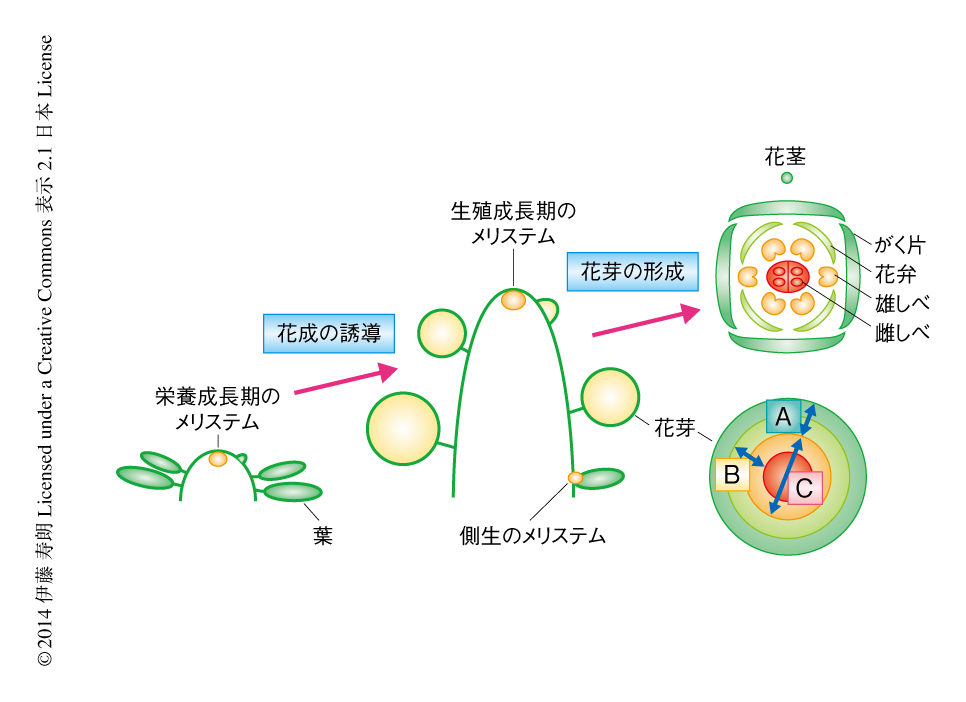
シロイヌナズナというモデル植物は過去30年間におよぶ遺伝学的な研究において多大な貢献をし,植物の発生における多くの基本原理を明らかにしてきた.植物は茎および根の先端部にあるメリステムとよばれる領域においてつねに幹細胞を維持しており,植物のかたちづくりは幹細胞の絶え間なくつづく増殖と分化にささえられている.栄養成長期には,茎のメリステムは自ら増殖しつつ,その側方に葉や茎を形成していく(図1).一方,環境からのシグナルあるいは内生するシグナルを感受すると,茎頂のメリステムにおいて栄養成長から生殖成長への転換が起こる.花成の誘導はその遺伝学的な解析から明らかにされた,光周期依存経路,春化依存経路,自律的経路,ジベレリン依存経路のほか,温度受容性経路,加齢経路という複数の独立した経路からのシグナルが統合され,最終的にFT,SOC1,AP1,LFYといった花成を促進する転写因子の発現が誘導される1-8)(図2).生殖成長がはじまると,まず,葉をともなう側生のメリステムが形成され,そののち,花芽が側方の器官として形成される(図1).花原基において,花成誘導遺伝子の下流の機能として花器官の分化の方向を決定するホメオティックタンパク質の発現が誘導される9)(図1,図2).ホメオティックタンパク質は自らのおかれた位置情報にもとづき花器官の分化を誘導する.花の幹細胞は発生の初期のみ維持され,ある一定数の花器官が形成されたのちその活性は抑制される.幹細胞の増殖の抑制もまたホメオティックタンパク質により担われている.
環境からあるいは内生するシグナルをうけてはたらく花成経路は,複数の転写因子およびクロマチン制御タンパク質からなる遺伝子発現制御のカスケードとして理解されるようになった.シグナルの感受から花器官の分化の方向を決定する遺伝子の発現制御にいたる花のかたちづくりは,時間軸および空間的な配置にそった特異的な遺伝子の発現制御ネットワークによりもたらされると考えることができる.とくに,花成経路および花器官の分化の方向を決定するタンパク質としては,MADSドメインをもつ転写因子が主要な役割をはたしている9,10)(図2).それらの転写因子は異なる植物種においても非常によく保存されており,花のかたちづくりの基本原理は植物種をこえて保存されている.
いまや高校の生物の教科書にも掲載されている花のABCモデルは,1991年に提唱された9-11).輪生状に形成されるがく片,花弁,雄しべ,雌しべの4つの花器官の分化の方向は,A,B,Cの3つのクラスのホメオティックタンパク質の組合せにより決定される(図1,図2).花の原基においてホメオティックタンパク質の機能がすべて消失した場合には,花器官は輪生状に形成される普通葉に変換する.Aクラス,Bクラス,Cクラスのホメオティックタンパク質はMADSドメインという共通したDNA結合ドメインをもつ転写因子として機能しており,そこには,やはりMADSドメインをもつEクラスのタンパク質も必要である12)(図2).すなわち,がく片への分化はAクラスのAP1とEクラスのSEPとの複合体により決定される.花弁への分化の方向を決定するスイッチとしてはAP1-AP3-PI-SEP複合体,雄しべへの分化の方向を決定するスイッチとしてはAG-AP3-PI-SEP複合体,雌しべへの分化の方向を決定するスイッチとしてはAG-SEP複合体が機能する13).これらのMADSドメインタンパク質複合体は,in vitroにおいては二量体としてCArGボックスとよばれる5’-CC(A/T)6GG-3’という回文配列に結合する.四量体の場合にはDNAにループ構造の形成を誘導し2カ所のCArGボックスに結合しうることが示されている14).
花弁への分化スイッチとなるAP1-AP3-PI-SEP複合体と,雄しべへの分化スイッチとなるAG-AP3-PI-SEP複合体との違いは,AP1とAGのみである(図2).植物体においてAP1とAGとのキメラタンパク質を発現させる実験により,その特異性はMADSドメインの内部のDNAとの結合には直接には関与しないC末端側,および,Iドメインにあり,さらに,C末端側のKドメインとCドメインはAP1とAGとで交換が可能であるとされている15).すなわち,花弁と雄しべの分化の方向を決定するスイッチの違いは,AP1とAGのDNA結合ドメインのC末端側に集約されるようである.また,雄しべと雌しべの分化のスイッチの違いはAP3およびPIの有無であり,その特異性はタンパク質の相互作用にかかわるとされるIドメインとKドメインにある15).
in vivoにおいては,SEP3は二量体の10倍以上の分子量である約670 kDaの複合体を形成している14).SEP3複合体,AP3複合体,AG複合体に共通して含まれるタンパク質として,SWI/SNFタイプのクロマチンリモデリングタンパク質であるBRM,ISWIタイプのクロマチンリモデリングタンパク質であるCHR4,CHR11,CHR17,ヒストン脱メチル化酵素であるREF6が同定されている14).さらに,SEP3複合体およびAG複合体に共通するタンパク質として,SWI/SNFタイプのクロマチンリモデリングタンパク質であるSYDがみつかっている.また,WD40ドメインをもつTPLはAP2と複合体を形成することが知られている16).すなわち,ホメオティックタンパク質は非常に大きな複合体を形成し,その複合体はクロマチンの制御にもかかわることが示唆される.しかし,花の発生において,ゲノムの特異的な部位にて時間軸にそってどのような複合体が形成されているかについては,まったくわかっていない.
ホメオティックタンパク質は,その変異により本来は機能する細胞が別のタイプの細胞に変換してしまうため,通常の遺伝学的な解析では下流の経路が解明できない.温度感受性変異体,あるいは,ステロイドホルモン受容体のリガンド結合部位と花のホメオティックタンパク質との融合タンパク質による活性誘導系,薬剤誘導遺伝子プロモーターとRNAi法により誘導の可能な機能破壊系のトランスジェニック植物を用いることにより,花のホメオティック遺伝子は花の発生の過程において継続的にさまざまな機能をもつことが明らかにされた.Aクラス,Bクラス,Cクラスのホメオティックタンパク質は,それぞれ4つの花器官の原基が形態的に明らかになるまえの未分化な細胞塊において,予定器官領域にてその発現がはじまる9).Aクラスのホメオティックタンパク質は花のメリステムの分化方向の決定にもかかわっているため,花原基の形成の直後から発現している.一方,BクラスおよびCクラスのホメオティックタンパク質は,がく片の原基の形成の際,花弁,雄しべ,雌しべの3つの器官の原基の区別がつかない花発生ステージ3にて17),それぞれ,花弁・雄しべ予定領域および雄しべ・雌しべ予定領域において発現が誘導される.Cクラスのホメオティックタンパク質であるAGは,花発生ステージ5から花発生ステージ6の花弁に運命づけられている器官を完全な雄しべに変換することができる18).一方,花発生ステージ7になってしまうとAGの発現を誘導しても花粉の形成は誘導できなくなるが,花弁の一部の形態を花糸のように中央部から基部にかけて細長くすることはできる.しかし,幹細胞の増殖の停止には花発生ステージ3からのAGの活性が必要である19).さらに,雄しべおよび雌しべの正常な形態形成には,花発生ステージ10から花発生ステージ13までにおよぶ継続的なAGの活性が必要であるとされている18,20).とくに,花の発生の中期よりのちにRNAi法によりAGの活性をノックダウンした場合,雌しべにおいて葉の特徴である枝分かれしたトライコームの形成が観察された20).すなわち,AGは雄しべおよび雌しべの分化を誘導するのみならず,葉のプログラムを抑制する機能ももつことが示された.また,花発生ステージ3から花発生ステージ4においてAP3をノックダウンすると雄しべから雌しべへの変換が起こるが,花発生ステージ5よりのちにAP3をノックダウンしても雄しべが雌しべに変換することはなく未成熟な雄しべが形成された21).すなわち,雄しべの分化方向の決定は花発生ステージ3からの早い時期に決められており,いったん雄しべに運命づけられた花器官は雌しべになることはない.一方で,花発生ステージ7から花発生ステージ8においてAP3をノックダウンすると花弁からがく片への変換がみられた.これらのことから,花弁の分化方向の決定は生殖器官よりも遅く,花の発生の中期まで決まっていないことが示唆された.以上,花器官の分化は花発生ステージに特異的に不可逆的に決定され,かつ,正常な機能をもつ花器官の形成にはホメオティックタンパク質による継続的な遺伝子の発現制御が必要であることが示された.
転写因子として機能するホメオティックタンパク質が実際にどれくらいの数のどのようなターゲット遺伝子を制御しているのかという問いへの答えは,2000年代からのゲノム科学の進展により明らかにされている.効率的な遺伝子発現の誘導系を活用して,マイクロアレイ法やRNA-seq法といったRNAプロファイリングを行うことにより,活性を誘導したのちの時間軸にそった包括的な遺伝子発現の解析が可能である.さらに,クロマチン免疫沈降法とそれにより濃縮された配列のゲノムタイリングアレイ法による解析あるいはハイスループットなシークエンシングにより,転写因子の結合する配列をゲノムワイドに解析することが可能である.これまでに,遺伝子発現およびゲノムワイドな結合にもとづき,Aクラス,Bクラス,Cクラス,Eクラスすべてのホメオティックタンパク質AP1,AP2,AP3-PI,AG,SEP3の下流のターゲット遺伝子が解析されている20-24).いずれのホメオティックタンパク質も細胞における発現量はほかの転写因子と比べ高く,ゲノムワイドな結合の解析では,AP3-PIが約1500,AP1,AP2,AGが約2000,SEP3が約4000と,数千にもおよぶ部位と結合していた.そのうち,遺伝子発現ともとの転写因子との相関関係がみられたものがそれぞれ数百個ほど同定された.また,AP1のターゲットとなる部位の半分以上はSEP3の結合部位でもあり,実際にMADSドメインタンパク質が複合体を形成して機能することがターゲット遺伝子の解析においても支持された23).
それぞれのターゲット遺伝子は花の発生の時間軸にそって特異的な細胞に分化する領域において発現が誘導される.たとえば,Cクラスのホメオティックタンパク質であるAGは,花発生ステージ6に,将来,雄しべの葯になる細胞において,別の転写因子であるSPL/NZZの発現を誘導することにより花粉の形成を誘導する25).ag変異体においてSPL/NZZ遺伝子の発現を異所的に誘導することにより,花弁の上部両端に花粉嚢の形成が誘導された.この器官は下等被子植物においてみられる葉的な雄しべの形態に似ていた.また,ag変異体において異所的な遺伝子プロモーターを用いてAG遺伝子の発現を誘導した場合にも,SPL/NZZ遺伝子は花発生ステージ5から花発生ステージ6のかぎられた花芽において,将来は葯となる特定の細胞においてのみ発現が誘導された.これは,SPL/NZZの発現の誘導には,AGのほかに発生の時期や細胞に特異的なタンパク質が関与していることを示唆した.
AGは花発生ステージ10の雄しべの花糸においてDAD1というジャスモン酸合成酵素の発現を誘導し,花粉の成熟,葯の開裂,花糸の伸長を協調的に制御している18).DAD1は異所的な遺伝子プロモーターを用いてAG遺伝子の発現を誘導したとき,成熟した花弁においても発現が誘導された.これは,すでにSPL/NZZの発現を誘導することができなくなり分化が花弁に方向づけられた花器官においてもDAD1の発現は誘導されうることを示した.実際に,花発生ステージ7よりのちにAGの発現を誘導したときにみられる花糸をもった花弁の形態がこれにより説明できる.一方,SPL/NZZの発現を誘導したのち継続的なAGの活性が得られない場合に観察された葉的な雄しべは,花粉の形成を誘導したのち,花糸や葯の形成に必要なタンパク質やDAD1の発現が誘導されなかった結果であると解釈される.
花器官は葉に由来する相同な器官であり,器官の配向を決める遺伝子や原基の形成にかかわる遺伝子などは葉と花で共通して発現している.興味深いことに,葉と花で共通に発現する原基の形成にかかわるJAG遺伝子は,花においてはAGの直接のターゲット遺伝子として制御されている20).Aクラス,Bクラス,Cクラスの遺伝子は花芽において発現量が高く,花に特異的な遺伝子の発現の誘導だけではなく,葉に特異的な遺伝子の発現の抑制や,葉と花に共通する遺伝子の花器官における発現の誘導まで,非常に多くの遺伝子の発現を制御している.花のホメオティックタンパク質としての機能をもつことの生物学的な意味の探求はこれからの課題である一方で,それぞれの遺伝子がどのように時間的および空間的に特異的な制御をうけているのかという問いへの挑戦もまた,ようやくはじまったところである.
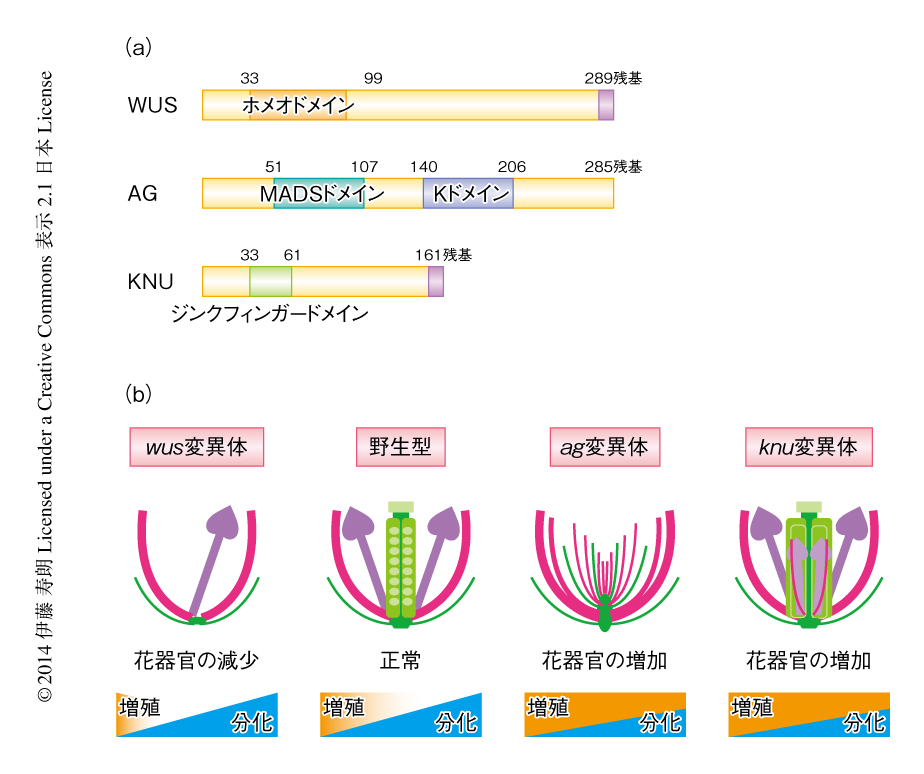
花においては,幹細胞の強い増殖活性が抑制されて種子が形成される.花の幹細胞の増殖および分化のマスター制御遺伝子は,Cクラスの遺伝子であるAG遺伝子である.AGの活性化には幹細胞の決定タンパク質であるWUSが直接的に機能している26,27)(図3).WUSはホメオドメインをもつ転写因子であり,その欠損により幹細胞は維持されなくなるため雌しべをもたない花になる.WUSはLFYなどの転写因子とともにAGの発現の誘導に直接的にかかわっている(図2).花発生ステージ3においてAGの発現が誘導されたのちにも,WUSは花の幹細胞の増殖を維持しつづける.WUSおよびAGは約2日間にわたり花原基の中心部において同時に機能する.そして,十分な幹細胞の増殖がなされた約2日のちに,AGがWUS遺伝子の発現を転写レベルで抑制することにより幹細胞の増殖を完全に停止する(図4).
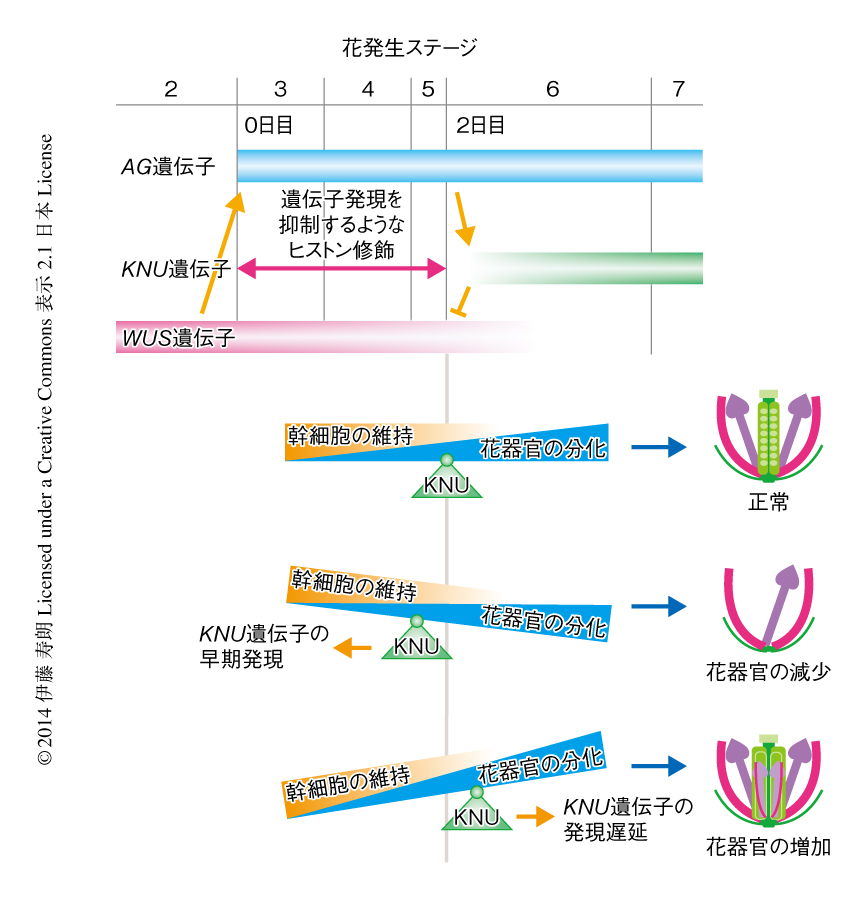
花の幹細胞の活性抑制のタイミングの制御においては,AGの直接のターゲット遺伝子であるKNU遺伝子が重要な役割をはたす19).KNUはジンクフィンガーをもつWUSの転写抑制タンパク質であり,花発生ステージ6に幹細胞の存在する領域において発現が誘導される(図3,図4).KNUの発現のタイミングは増殖と分化のバランスを直接的に制御するものであり,早期の発現により花器官の数は減少し,逆に,発現の遅延は花器官の数の増加につながる(図4).すなわち,KNUはWUSとAGによる時間的に隔てられた負のフィードバック経路の介在タンパク質として作用する.また,KNUの早期の発現はポリコームタンパク質により導入および維持されるヒストン修飾により抑制されている.
転写因子であるAGは花発生ステージ3において発現が誘導されたのち,花発生ステージ6においてKNUおよびSPL/NZZの発現を誘導する19,25).花発生ステージ6の花芽においてAGの発現を誘導すると,SPL/NZZの発現は花粉嚢の形成される領域において即座に誘導される.ところが,KNUの発現はAGの発現を誘導してから2日のちに誘導される.さらに,幹細胞の増殖の抑制には花発生ステージ3からはじまるAGの活性が必要であり,AGの発現を1日おくらせて花発生ステージ4において誘導した場合には,KNUの発現も通常よりも1日おくれて観察された19).すなわち,同じ時期に異なった領域において発現の誘導されるAGの2つの直接のターゲット遺伝子であるKNU遺伝子とSPL遺伝子は,それぞれ異なった機構により制御されていると推定される.AGはKNU遺伝子座に直接に結合するのにもかかわらず,発現の誘導に2日間のずれがあるのはなぜだろうか.
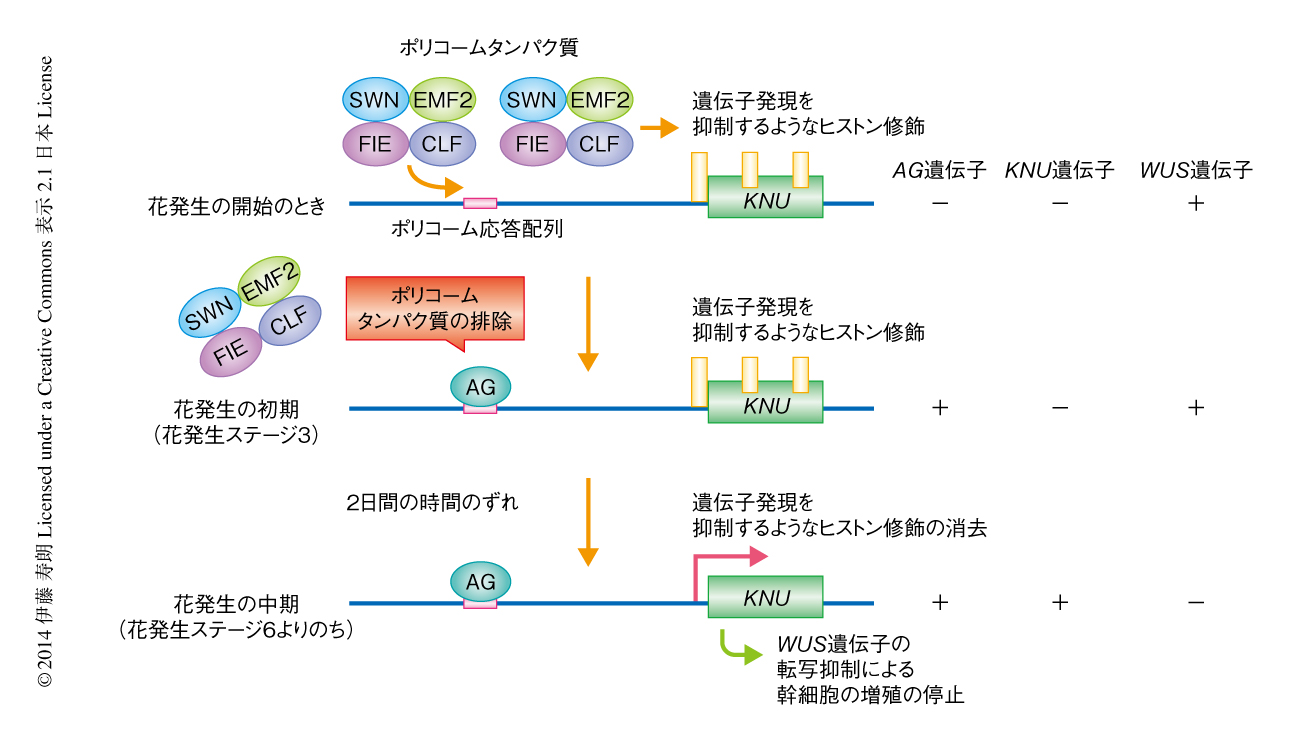
AGによる転写の誘導はポリコームタンパク質を排除することよりはじまる(図5).遺伝子発現を抑制するようなヒストン修飾の維持に必要なポリコームタンパク質の結合がAGにより阻害されると,細胞周期の進行にともない遺伝子発現の抑制状態の希釈が起こる.これにより,KNU遺伝子の発現が2日間という時間のずれをともない誘導されるというバイオタイマー機構が提唱されている28)(図5).これは,転写因子がポリコームタンパク質を排除し時間的な制御を細胞周期と連動させるはじめての例となった.
ポリコームタンパク質は発生の過程において,時間的および空間的に特異的な遺伝子発現の制御に重要な役割をはたす10).ポリコームタンパク質はほぼすべての細胞において発現しているが,特異的なゲノム領域への導入とそこからの排除により,時間的および空間的に特異的な遺伝子発現の制御がもたらされていると考えられる.ポリコームタンパク質をリクルートするのに必要なポリコーム応答配列はショウジョウバエにおいて最初に同定された29).KNU遺伝子の上流にはこのポリコーム応答配列があり,その約150 bpの短い配列はポリコームタンパク質を導入し,遺伝子発現を抑制するようなヒストン修飾を蓄積させることにより別の遺伝子プロモーターの活性を抑制するという機能をもつ28).花の発生の過程においてAGの発現が誘導されたのち,AGがKNU遺伝子の上流のポリコーム応答配列に結合すると,ポリコームタンパク質がKNU遺伝子座から排除され結合できなくなる.動物における実験では,遺伝子発現を抑制するようなヒストン修飾を維持するために,ポリコームタンパク質はS期のDNA複製フォークにもつねに結合していることが示されている30,31).すなわち,ポリコームタンパク質が排除されると,遺伝子発現を抑制するようなヒストン修飾は消去される(図5).また,ヒストン修飾のダイナミックな変化には細胞周期の進行が必要である32).実際に,KNU遺伝子の発現の誘導までのタイミングは細胞周期の進行に依存的である28).実験的には,細胞周期の進行を早めることにより1日から1日半でKNU遺伝子の発現は誘導され,逆に,細胞周期の阻害により2日間の時間のずれはさらに遅れた.さらに,人工的な遺伝子プロモーターを構築することによりポリコームタンパク質の排除による転写制御系を再現することもできた.
ショウジョウバエにおいて,ポリコーム応答配列の欠損によりヒストン修飾によって抑制されていたターゲット遺伝子の発現が,細胞周期の進行に依存して脱抑制されることが示されている33,34).植物の幹細胞においては,実際に,転写因子がポリコーム応答配列の活性を阻害することによりターゲット遺伝子の発現を脱抑制していると考えられた28).AGは花器官の分化および幹細胞の増殖の抑制を統合的に制御している.幹細胞の増殖の抑制が細胞周期にもとづくバイオタイマーにより制御されているのはなぜだろうか.ゲノムワイドな解析により,AGの下流において複数の細胞周期の制御タンパク質の発現が制御されていることが示されている.可能性のひとつとして,AGは花器官の分化の誘導と増殖の抑制のタイミングの最適化をはかるため,細胞周期を介して増殖と分化を微調節していることが考えられる.また,動物においては未分化な幹細胞は概日時計のリズムをもたないという報告がある35).花の幹細胞の発生の過程における時間的な制御において,細胞周期の進行にともなうバイオタイマー機構には生物学的な必然性のあることが予想される.
ABCモデルの発表から,花の形態形成の分子レベルでの解析は大きく発展してきた.この背景には,変異体から遺伝子をクローニングし既知の変異体との多重変異体を作製することにより,既知の遺伝子との関連を探していく遺伝学的な方法論が非常にうまく機能してきたことがある.さらに,過去十数年間のゲノム科学にもとづくマイクロアレイ法や次世代シークエンサーなどの技術進歩により,Aクラス,Bクラス,Cクラスのタンパク質の下流においてはたらくターゲット遺伝子の全体像が解明されつつある.同時に,鍵となる遺伝子のイメージングや数理生物学的な解析により,遺伝学的に同定された鍵となる遺伝子から表現型にいたるまでの過程を理解しようとするアプローチも活発に行われている.しかしながら,個々の遺伝子がどのようにして時間的および空間的に特異的に制御されているのかは,いまだ明らかになっていない.さらに,それぞれのホメオティックタンパク質により制御される非常に多くの遺伝子が,どのように協調的に作用して秩序をもった美しいかたちや機能がつくりだされるのかについても,ほとんどが未知である.このレビューにおいてその一例を示したように,これらの制御にはエピジェネティックなクロマチンの制御が重要な役割をはたしている.また,植物ホルモンも発生の調和的な制御において欠かせない役割を担っている.
今後の課題として,ゲノムワイドな解析において時間軸にそった空間的な特異性の解像度を上げていく必要があると考えられる.そのためには,これまでの生化学的な解析に用いてきた試料の均一性を上げていくことが肝要である.それには,特定の細胞が異常に増殖する変異体の活用や,特定の遺伝子レポーターを用いたセルソーティング,発現を指標とする精製系などの広い活用が期待される.また,解析手法それ自体の感度を上げることも必要である.今後のさらなる技術的な進歩により細胞に特異的な解析ができるようになれば,時間軸および空間的な配置にもとづく膨大な情報が得られるだろう.そして,それらを統合的に解析していく手法も必要になってくるであろう.そこには,変わりゆく環境に順応して変化する植物の高い可塑性,あるいは,環境適応力も組み込む必要がある.ホメオティック遺伝子から3次元の形態の構築にいたる遺伝子の動的な発現制御の全貌の解明は,現代の植物科学の研究者にとって使命であるといえる.
略歴:1997年 京都大学大学院理学研究科博士課程 修了,同年 米国California Institute of Technology研究員,2003年 同Senior Research Fellow,2005年 シンガポールTemasek Life Sciences LaboratoryにてPrincipal Investigatorを経て,2011年より同Senior Principal Investigator.
抱負:Things do always work out.
研究室URL:http://www.tll.org.sg/group-leaders/toshiro-ito/
© 2014 伊藤 寿朗 Licensed under CC 表示 2.1 日本
(シンガポールTemasek Life Sciences Laboratory)
email:伊藤寿朗
領域融合レビュー, 3, e014 (2014) DOI: 10.7875/leading.author.3.e014
Toshiro Ito: Growth and differentiation control in Arabidopsis flower development.
要 約
花の器官形成におけるABCモデルが提唱されてから20年あまりたつ.シロイヌナズナを中心としたモデル植物における遺伝学的な解析からはじまった花の発生の研究は,のちの分子遺伝学およびゲノム科学の進歩により大きく進展してきた.Aクラス,Bクラス,Cクラスの遺伝子がクローニングされ,これらのコードする転写因子の上流および下流における遺伝子制御ネットワークが明らかにされつつある.それらの転写因子はゲノムにおいて数千をこえる部位に結合し,数百をこえる遺伝子の発現を制御している.それらには,生殖細胞の誘導,花の器官形成や成熟において鍵となる転写因子やクロマチン制御タンパク質,植物ホルモンの合成,シグナル伝達タンパク質などをコードする遺伝子が含まれる.それぞれの遺伝子は花の発生の時間軸にそって特異的な細胞において発現し機能している.最近のエピジェネティクスの研究に着目しながら,花の幹細胞の増殖および分化における時間的および空間的に特異的な制御機構について論じる.
はじめに
種子植物は生殖器官として“花”をもち,有性生殖の結果として種子を形成する.人類は数百万年の歴史のなかで花の産物である実や種子が食用に適することを見い出し,果実や穀物の生産性の向上をめざしそれらの植物を培ってきた.また,花はその美しさゆえ,古代より人々に愛されてきた.古今東西の遺跡や壁画,紋章などにおいて,花の絵柄は普遍的にみられ,美や生命力の象徴ともいえる.同時に,移ろいゆく四季折々を彩る花は,われわれ日本人の無常と“もののあはれ”の価値観の象徴でもある.実際に,すべての植物において花を咲かすことは幹細胞としての活性を停止することであり,これは幹細胞の死を意味する.時が来たならば自らの増殖を止め,次世代の種子をつくることに専心する花の生きざまに,われわれ日本人は生命の美しさと儚さを重ね合せるのかもしれない.
現代の植物科学の研究者には,分子遺伝学およびゲノム科学という強力なツールがあたえられている.これにより,花の成り立ちの基本原理や生命の美しさそのものを分子の言葉で読み解くことが可能になった.このレビューでは,シロイヌナズナにおける花の発生の研究のなかでも,とくに過去10年間の進歩を,エピジェネティクスを介した幹細胞の増殖の制御機構に着目して論じる.
1.シロイヌナズナにおける花の発生の流れ
シロイヌナズナというモデル植物は過去30年間におよぶ遺伝学的な研究において多大な貢献をし,植物の発生における多くの基本原理を明らかにしてきた.植物は茎および根の先端部にあるメリステムとよばれる領域においてつねに幹細胞を維持しており,植物のかたちづくりは幹細胞の絶え間なくつづく増殖と分化にささえられている.栄養成長期には,茎のメリステムは自ら増殖しつつ,その側方に葉や茎を形成していく(図1).一方,環境からのシグナルあるいは内生するシグナルを感受すると,茎頂のメリステムにおいて栄養成長から生殖成長への転換が起こる.花成の誘導はその遺伝学的な解析から明らかにされた,光周期依存経路,春化依存経路,自律的経路,ジベレリン依存経路のほか,温度受容性経路,加齢経路という複数の独立した経路からのシグナルが統合され,最終的にFT,SOC1,AP1,LFYといった花成を促進する転写因子の発現が誘導される1-8)(図2).生殖成長がはじまると,まず,葉をともなう側生のメリステムが形成され,そののち,花芽が側方の器官として形成される(図1).花原基において,花成誘導遺伝子の下流の機能として花器官の分化の方向を決定するホメオティックタンパク質の発現が誘導される9)(図1,図2).ホメオティックタンパク質は自らのおかれた位置情報にもとづき花器官の分化を誘導する.花の幹細胞は発生の初期のみ維持され,ある一定数の花器官が形成されたのちその活性は抑制される.幹細胞の増殖の抑制もまたホメオティックタンパク質により担われている.
環境からあるいは内生するシグナルをうけてはたらく花成経路は,複数の転写因子およびクロマチン制御タンパク質からなる遺伝子発現制御のカスケードとして理解されるようになった.シグナルの感受から花器官の分化の方向を決定する遺伝子の発現制御にいたる花のかたちづくりは,時間軸および空間的な配置にそった特異的な遺伝子の発現制御ネットワークによりもたらされると考えることができる.とくに,花成経路および花器官の分化の方向を決定するタンパク質としては,MADSドメインをもつ転写因子が主要な役割をはたしている9,10)(図2).それらの転写因子は異なる植物種においても非常によく保存されており,花のかたちづくりの基本原理は植物種をこえて保存されている.
2.花の分化の方向の決定にかかわるホメオティックタンパク質の作用機構
いまや高校の生物の教科書にも掲載されている花のABCモデルは,1991年に提唱された9-11).輪生状に形成されるがく片,花弁,雄しべ,雌しべの4つの花器官の分化の方向は,A,B,Cの3つのクラスのホメオティックタンパク質の組合せにより決定される(図1,図2).花の原基においてホメオティックタンパク質の機能がすべて消失した場合には,花器官は輪生状に形成される普通葉に変換する.Aクラス,Bクラス,Cクラスのホメオティックタンパク質はMADSドメインという共通したDNA結合ドメインをもつ転写因子として機能しており,そこには,やはりMADSドメインをもつEクラスのタンパク質も必要である12)(図2).すなわち,がく片への分化はAクラスのAP1とEクラスのSEPとの複合体により決定される.花弁への分化の方向を決定するスイッチとしてはAP1-AP3-PI-SEP複合体,雄しべへの分化の方向を決定するスイッチとしてはAG-AP3-PI-SEP複合体,雌しべへの分化の方向を決定するスイッチとしてはAG-SEP複合体が機能する13).これらのMADSドメインタンパク質複合体は,in vitroにおいては二量体としてCArGボックスとよばれる5’-CC(A/T)6GG-3’という回文配列に結合する.四量体の場合にはDNAにループ構造の形成を誘導し2カ所のCArGボックスに結合しうることが示されている14).
花弁への分化スイッチとなるAP1-AP3-PI-SEP複合体と,雄しべへの分化スイッチとなるAG-AP3-PI-SEP複合体との違いは,AP1とAGのみである(図2).植物体においてAP1とAGとのキメラタンパク質を発現させる実験により,その特異性はMADSドメインの内部のDNAとの結合には直接には関与しないC末端側,および,Iドメインにあり,さらに,C末端側のKドメインとCドメインはAP1とAGとで交換が可能であるとされている15).すなわち,花弁と雄しべの分化の方向を決定するスイッチの違いは,AP1とAGのDNA結合ドメインのC末端側に集約されるようである.また,雄しべと雌しべの分化のスイッチの違いはAP3およびPIの有無であり,その特異性はタンパク質の相互作用にかかわるとされるIドメインとKドメインにある15).
in vivoにおいては,SEP3は二量体の10倍以上の分子量である約670 kDaの複合体を形成している14).SEP3複合体,AP3複合体,AG複合体に共通して含まれるタンパク質として,SWI/SNFタイプのクロマチンリモデリングタンパク質であるBRM,ISWIタイプのクロマチンリモデリングタンパク質であるCHR4,CHR11,CHR17,ヒストン脱メチル化酵素であるREF6が同定されている14).さらに,SEP3複合体およびAG複合体に共通するタンパク質として,SWI/SNFタイプのクロマチンリモデリングタンパク質であるSYDがみつかっている.また,WD40ドメインをもつTPLはAP2と複合体を形成することが知られている16).すなわち,ホメオティックタンパク質は非常に大きな複合体を形成し,その複合体はクロマチンの制御にもかかわることが示唆される.しかし,花の発生において,ゲノムの特異的な部位にて時間軸にそってどのような複合体が形成されているかについては,まったくわかっていない.
3.花のホメオティックタンパク質の発現および機能
ホメオティックタンパク質は,その変異により本来は機能する細胞が別のタイプの細胞に変換してしまうため,通常の遺伝学的な解析では下流の経路が解明できない.温度感受性変異体,あるいは,ステロイドホルモン受容体のリガンド結合部位と花のホメオティックタンパク質との融合タンパク質による活性誘導系,薬剤誘導遺伝子プロモーターとRNAi法により誘導の可能な機能破壊系のトランスジェニック植物を用いることにより,花のホメオティック遺伝子は花の発生の過程において継続的にさまざまな機能をもつことが明らかにされた.Aクラス,Bクラス,Cクラスのホメオティックタンパク質は,それぞれ4つの花器官の原基が形態的に明らかになるまえの未分化な細胞塊において,予定器官領域にてその発現がはじまる9).Aクラスのホメオティックタンパク質は花のメリステムの分化方向の決定にもかかわっているため,花原基の形成の直後から発現している.一方,BクラスおよびCクラスのホメオティックタンパク質は,がく片の原基の形成の際,花弁,雄しべ,雌しべの3つの器官の原基の区別がつかない花発生ステージ3にて17),それぞれ,花弁・雄しべ予定領域および雄しべ・雌しべ予定領域において発現が誘導される.Cクラスのホメオティックタンパク質であるAGは,花発生ステージ5から花発生ステージ6の花弁に運命づけられている器官を完全な雄しべに変換することができる18).一方,花発生ステージ7になってしまうとAGの発現を誘導しても花粉の形成は誘導できなくなるが,花弁の一部の形態を花糸のように中央部から基部にかけて細長くすることはできる.しかし,幹細胞の増殖の停止には花発生ステージ3からのAGの活性が必要である19).さらに,雄しべおよび雌しべの正常な形態形成には,花発生ステージ10から花発生ステージ13までにおよぶ継続的なAGの活性が必要であるとされている18,20).とくに,花の発生の中期よりのちにRNAi法によりAGの活性をノックダウンした場合,雌しべにおいて葉の特徴である枝分かれしたトライコームの形成が観察された20).すなわち,AGは雄しべおよび雌しべの分化を誘導するのみならず,葉のプログラムを抑制する機能ももつことが示された.また,花発生ステージ3から花発生ステージ4においてAP3をノックダウンすると雄しべから雌しべへの変換が起こるが,花発生ステージ5よりのちにAP3をノックダウンしても雄しべが雌しべに変換することはなく未成熟な雄しべが形成された21).すなわち,雄しべの分化方向の決定は花発生ステージ3からの早い時期に決められており,いったん雄しべに運命づけられた花器官は雌しべになることはない.一方で,花発生ステージ7から花発生ステージ8においてAP3をノックダウンすると花弁からがく片への変換がみられた.これらのことから,花弁の分化方向の決定は生殖器官よりも遅く,花の発生の中期まで決まっていないことが示唆された.以上,花器官の分化は花発生ステージに特異的に不可逆的に決定され,かつ,正常な機能をもつ花器官の形成にはホメオティックタンパク質による継続的な遺伝子の発現制御が必要であることが示された.
4.花のホメオティックタンパク質のターゲットとなる遺伝子
転写因子として機能するホメオティックタンパク質が実際にどれくらいの数のどのようなターゲット遺伝子を制御しているのかという問いへの答えは,2000年代からのゲノム科学の進展により明らかにされている.効率的な遺伝子発現の誘導系を活用して,マイクロアレイ法やRNA-seq法といったRNAプロファイリングを行うことにより,活性を誘導したのちの時間軸にそった包括的な遺伝子発現の解析が可能である.さらに,クロマチン免疫沈降法とそれにより濃縮された配列のゲノムタイリングアレイ法による解析あるいはハイスループットなシークエンシングにより,転写因子の結合する配列をゲノムワイドに解析することが可能である.これまでに,遺伝子発現およびゲノムワイドな結合にもとづき,Aクラス,Bクラス,Cクラス,Eクラスすべてのホメオティックタンパク質AP1,AP2,AP3-PI,AG,SEP3の下流のターゲット遺伝子が解析されている20-24).いずれのホメオティックタンパク質も細胞における発現量はほかの転写因子と比べ高く,ゲノムワイドな結合の解析では,AP3-PIが約1500,AP1,AP2,AGが約2000,SEP3が約4000と,数千にもおよぶ部位と結合していた.そのうち,遺伝子発現ともとの転写因子との相関関係がみられたものがそれぞれ数百個ほど同定された.また,AP1のターゲットとなる部位の半分以上はSEP3の結合部位でもあり,実際にMADSドメインタンパク質が複合体を形成して機能することがターゲット遺伝子の解析においても支持された23).
それぞれのターゲット遺伝子は花の発生の時間軸にそって特異的な細胞に分化する領域において発現が誘導される.たとえば,Cクラスのホメオティックタンパク質であるAGは,花発生ステージ6に,将来,雄しべの葯になる細胞において,別の転写因子であるSPL/NZZの発現を誘導することにより花粉の形成を誘導する25).ag変異体においてSPL/NZZ遺伝子の発現を異所的に誘導することにより,花弁の上部両端に花粉嚢の形成が誘導された.この器官は下等被子植物においてみられる葉的な雄しべの形態に似ていた.また,ag変異体において異所的な遺伝子プロモーターを用いてAG遺伝子の発現を誘導した場合にも,SPL/NZZ遺伝子は花発生ステージ5から花発生ステージ6のかぎられた花芽において,将来は葯となる特定の細胞においてのみ発現が誘導された.これは,SPL/NZZの発現の誘導には,AGのほかに発生の時期や細胞に特異的なタンパク質が関与していることを示唆した.
AGは花発生ステージ10の雄しべの花糸においてDAD1というジャスモン酸合成酵素の発現を誘導し,花粉の成熟,葯の開裂,花糸の伸長を協調的に制御している18).DAD1は異所的な遺伝子プロモーターを用いてAG遺伝子の発現を誘導したとき,成熟した花弁においても発現が誘導された.これは,すでにSPL/NZZの発現を誘導することができなくなり分化が花弁に方向づけられた花器官においてもDAD1の発現は誘導されうることを示した.実際に,花発生ステージ7よりのちにAGの発現を誘導したときにみられる花糸をもった花弁の形態がこれにより説明できる.一方,SPL/NZZの発現を誘導したのち継続的なAGの活性が得られない場合に観察された葉的な雄しべは,花粉の形成を誘導したのち,花糸や葯の形成に必要なタンパク質やDAD1の発現が誘導されなかった結果であると解釈される.
花器官は葉に由来する相同な器官であり,器官の配向を決める遺伝子や原基の形成にかかわる遺伝子などは葉と花で共通して発現している.興味深いことに,葉と花で共通に発現する原基の形成にかかわるJAG遺伝子は,花においてはAGの直接のターゲット遺伝子として制御されている20).Aクラス,Bクラス,Cクラスの遺伝子は花芽において発現量が高く,花に特異的な遺伝子の発現の誘導だけではなく,葉に特異的な遺伝子の発現の抑制や,葉と花に共通する遺伝子の花器官における発現の誘導まで,非常に多くの遺伝子の発現を制御している.花のホメオティックタンパク質としての機能をもつことの生物学的な意味の探求はこれからの課題である一方で,それぞれの遺伝子がどのように時間的および空間的に特異的な制御をうけているのかという問いへの挑戦もまた,ようやくはじまったところである.
5.花の幹細胞における増殖の抑制機構
花においては,幹細胞の強い増殖活性が抑制されて種子が形成される.花の幹細胞の増殖および分化のマスター制御遺伝子は,Cクラスの遺伝子であるAG遺伝子である.AGの活性化には幹細胞の決定タンパク質であるWUSが直接的に機能している26,27)(図3).WUSはホメオドメインをもつ転写因子であり,その欠損により幹細胞は維持されなくなるため雌しべをもたない花になる.WUSはLFYなどの転写因子とともにAGの発現の誘導に直接的にかかわっている(図2).花発生ステージ3においてAGの発現が誘導されたのちにも,WUSは花の幹細胞の増殖を維持しつづける.WUSおよびAGは約2日間にわたり花原基の中心部において同時に機能する.そして,十分な幹細胞の増殖がなされた約2日のちに,AGがWUS遺伝子の発現を転写レベルで抑制することにより幹細胞の増殖を完全に停止する(図4).
花の幹細胞の活性抑制のタイミングの制御においては,AGの直接のターゲット遺伝子であるKNU遺伝子が重要な役割をはたす19).KNUはジンクフィンガーをもつWUSの転写抑制タンパク質であり,花発生ステージ6に幹細胞の存在する領域において発現が誘導される(図3,図4).KNUの発現のタイミングは増殖と分化のバランスを直接的に制御するものであり,早期の発現により花器官の数は減少し,逆に,発現の遅延は花器官の数の増加につながる(図4).すなわち,KNUはWUSとAGによる時間的に隔てられた負のフィードバック経路の介在タンパク質として作用する.また,KNUの早期の発現はポリコームタンパク質により導入および維持されるヒストン修飾により抑制されている.
6.ホメオティックタンパク質によるヒストン修飾の制御
転写因子であるAGは花発生ステージ3において発現が誘導されたのち,花発生ステージ6においてKNUおよびSPL/NZZの発現を誘導する19,25).花発生ステージ6の花芽においてAGの発現を誘導すると,SPL/NZZの発現は花粉嚢の形成される領域において即座に誘導される.ところが,KNUの発現はAGの発現を誘導してから2日のちに誘導される.さらに,幹細胞の増殖の抑制には花発生ステージ3からはじまるAGの活性が必要であり,AGの発現を1日おくらせて花発生ステージ4において誘導した場合には,KNUの発現も通常よりも1日おくれて観察された19).すなわち,同じ時期に異なった領域において発現の誘導されるAGの2つの直接のターゲット遺伝子であるKNU遺伝子とSPL遺伝子は,それぞれ異なった機構により制御されていると推定される.AGはKNU遺伝子座に直接に結合するのにもかかわらず,発現の誘導に2日間のずれがあるのはなぜだろうか.
AGによる転写の誘導はポリコームタンパク質を排除することよりはじまる(図5).遺伝子発現を抑制するようなヒストン修飾の維持に必要なポリコームタンパク質の結合がAGにより阻害されると,細胞周期の進行にともない遺伝子発現の抑制状態の希釈が起こる.これにより,KNU遺伝子の発現が2日間という時間のずれをともない誘導されるというバイオタイマー機構が提唱されている28)(図5).これは,転写因子がポリコームタンパク質を排除し時間的な制御を細胞周期と連動させるはじめての例となった.
ポリコームタンパク質は発生の過程において,時間的および空間的に特異的な遺伝子発現の制御に重要な役割をはたす10).ポリコームタンパク質はほぼすべての細胞において発現しているが,特異的なゲノム領域への導入とそこからの排除により,時間的および空間的に特異的な遺伝子発現の制御がもたらされていると考えられる.ポリコームタンパク質をリクルートするのに必要なポリコーム応答配列はショウジョウバエにおいて最初に同定された29).KNU遺伝子の上流にはこのポリコーム応答配列があり,その約150 bpの短い配列はポリコームタンパク質を導入し,遺伝子発現を抑制するようなヒストン修飾を蓄積させることにより別の遺伝子プロモーターの活性を抑制するという機能をもつ28).花の発生の過程においてAGの発現が誘導されたのち,AGがKNU遺伝子の上流のポリコーム応答配列に結合すると,ポリコームタンパク質がKNU遺伝子座から排除され結合できなくなる.動物における実験では,遺伝子発現を抑制するようなヒストン修飾を維持するために,ポリコームタンパク質はS期のDNA複製フォークにもつねに結合していることが示されている30,31).すなわち,ポリコームタンパク質が排除されると,遺伝子発現を抑制するようなヒストン修飾は消去される(図5).また,ヒストン修飾のダイナミックな変化には細胞周期の進行が必要である32).実際に,KNU遺伝子の発現の誘導までのタイミングは細胞周期の進行に依存的である28).実験的には,細胞周期の進行を早めることにより1日から1日半でKNU遺伝子の発現は誘導され,逆に,細胞周期の阻害により2日間の時間のずれはさらに遅れた.さらに,人工的な遺伝子プロモーターを構築することによりポリコームタンパク質の排除による転写制御系を再現することもできた.
ショウジョウバエにおいて,ポリコーム応答配列の欠損によりヒストン修飾によって抑制されていたターゲット遺伝子の発現が,細胞周期の進行に依存して脱抑制されることが示されている33,34).植物の幹細胞においては,実際に,転写因子がポリコーム応答配列の活性を阻害することによりターゲット遺伝子の発現を脱抑制していると考えられた28).AGは花器官の分化および幹細胞の増殖の抑制を統合的に制御している.幹細胞の増殖の抑制が細胞周期にもとづくバイオタイマーにより制御されているのはなぜだろうか.ゲノムワイドな解析により,AGの下流において複数の細胞周期の制御タンパク質の発現が制御されていることが示されている.可能性のひとつとして,AGは花器官の分化の誘導と増殖の抑制のタイミングの最適化をはかるため,細胞周期を介して増殖と分化を微調節していることが考えられる.また,動物においては未分化な幹細胞は概日時計のリズムをもたないという報告がある35).花の幹細胞の発生の過程における時間的な制御において,細胞周期の進行にともなうバイオタイマー機構には生物学的な必然性のあることが予想される.
おわりに
ABCモデルの発表から,花の形態形成の分子レベルでの解析は大きく発展してきた.この背景には,変異体から遺伝子をクローニングし既知の変異体との多重変異体を作製することにより,既知の遺伝子との関連を探していく遺伝学的な方法論が非常にうまく機能してきたことがある.さらに,過去十数年間のゲノム科学にもとづくマイクロアレイ法や次世代シークエンサーなどの技術進歩により,Aクラス,Bクラス,Cクラスのタンパク質の下流においてはたらくターゲット遺伝子の全体像が解明されつつある.同時に,鍵となる遺伝子のイメージングや数理生物学的な解析により,遺伝学的に同定された鍵となる遺伝子から表現型にいたるまでの過程を理解しようとするアプローチも活発に行われている.しかしながら,個々の遺伝子がどのようにして時間的および空間的に特異的に制御されているのかは,いまだ明らかになっていない.さらに,それぞれのホメオティックタンパク質により制御される非常に多くの遺伝子が,どのように協調的に作用して秩序をもった美しいかたちや機能がつくりだされるのかについても,ほとんどが未知である.このレビューにおいてその一例を示したように,これらの制御にはエピジェネティックなクロマチンの制御が重要な役割をはたしている.また,植物ホルモンも発生の調和的な制御において欠かせない役割を担っている.
今後の課題として,ゲノムワイドな解析において時間軸にそった空間的な特異性の解像度を上げていく必要があると考えられる.そのためには,これまでの生化学的な解析に用いてきた試料の均一性を上げていくことが肝要である.それには,特定の細胞が異常に増殖する変異体の活用や,特定の遺伝子レポーターを用いたセルソーティング,発現を指標とする精製系などの広い活用が期待される.また,解析手法それ自体の感度を上げることも必要である.今後のさらなる技術的な進歩により細胞に特異的な解析ができるようになれば,時間軸および空間的な配置にもとづく膨大な情報が得られるだろう.そして,それらを統合的に解析していく手法も必要になってくるであろう.そこには,変わりゆく環境に順応して変化する植物の高い可塑性,あるいは,環境適応力も組み込む必要がある.ホメオティック遺伝子から3次元の形態の構築にいたる遺伝子の動的な発現制御の全貌の解明は,現代の植物科学の研究者にとって使命であるといえる.
文 献
- Kobayashi, Y., Kaya, H., Goto, K. et al.: A pair of related genes with antagonistic roles in mediating flowering signals. Science, 286, 1960-1962 (1999)[PubMed]
- Lee, H., Suh, S. S., Park, E. et al.: The AGAMOUS-LIKE 20 MADS domain protein integrates floral inductive pathways in Arabidopsis. Genes Dev., 14, 2366-2376 (2000)[PubMed]
- Mandel, M. A., Gustafson-Brown, C., Savidge, B. et al.: Molecular characterization of the Arabidopsis floral homeotic gene APETALA1. Nature, 360, 273-277 (1992)[PubMed]
- Weigel, D. & Meyerowitz, E. M.: Activation of floral homeotic genes in Arabidopsis. Science, 261, 1723-1726 (1993)[PubMed]
- He, Y. & Amasino, R. M.: Role of chromatin modification in flowering-time control. Trends Plant Sci., 10, 30-35 (2005)[PubMed]
- Gan, E. S., Xu, Y., Wong, J. Y. et al.: Jumonji demethylases moderate precocious flowering at elevated temperature via regulation of FLC in Arabidopsis. Nat. Commun., 5, 5098 (2014)[PubMed]
- Xu, Y., Wang, Y., Stroud, H. et al.: A matrix protein silences transposons and repeats through interaction with retinoblastoma-associated proteins. Curr. Biol., 23, 345-350 (2013)[PubMed]
- Xu, Y., Gan, E. S., Zhou, J. et al.: Arabidopsis MRG domain proteins bridge two histone modifications to elevate expression of flowering genes. Nucleic Acids Res., 42, 10960-10974 (2015)[PubMed]
- Ito, T.: Coordination of flower development by homeotic master regulators. Curr. Opin. Plant Biol., 14, 53-59 (2011)[PubMed]
- Gan, E. S., Huang, J. & Ito, T.: Functional roles of histone modification, chromatin remodeling and microRNAs in Arabidopsis flower development. Int. Rev. Cell Mol. Biol., 305, 115-161 (2013)[PubMed]
- Coen, E. S. & Meyerowitz, E. M.: The war of the whorls: genetic interactions controlling flower development. Nature, 353, 31-37 (1991)[PubMed]
- Pelaz, S., Ditta, G. S., Baumann, E. et al.: B and C floral organ identity functions require SEPALLATA MADS-box genes. Nature, 405, 200-203 (2000)[PubMed]
- Honma, T. & Goto, K.: Complexes of MADS-box proteins are sufficient to convert leaves into floral organs. Nature, 409, 525-529 (2001)[PubMed]
- Smaczniak, C., Immink, R. G., Muino, J. M. et al.: Characterization of MADS-domain transcription factor complexes in Arabidopsis flower development. Proc. Natl. Acad. Sci. USA, 109, 1560-1565 (2012)[PubMed]
- Krizek, B. A. & Meyerowitz, E. M.: Mapping the protein regions responsible for the functional specificities of the Arabidopsis MADS domain organ-identity proteins. Proc. Natl. Acad. Sci. USA, 93, 4063-4070 (1996)[PubMed]
- Krogan, N. T., Hogan, K. & Long, J. A.: APETALA2 negatively regulates multiple floral organ identity genes in Arabidopsis by recruiting the co-repressor TOPLESS and the histone deacetylase HDA19. Development, 139, 4180-4190 (2012)[PubMed]
- Smyth, D. R., Bowman, J. L. & Meyerowitz, E. M.: Early flower development in Arabidopsis. Plant Cell, 2, 755-767 (1990)[PubMed]
- Ito, T., Ng, K. H., Lim, T. S. et al.: The homeotic protein AGAMOUS controls late stamen development by regulating a jasmonate biosynthetic gene in Arabidopsis. Plant Cell, 19, 3516-3529 (2007)[PubMed]
- Sun, B., Xu, Y., Ng, K. H. et al.: A timing mechanism for stem cell maintenance and differentiation in the Arabidopsis floral meristem. Genes Dev., 23, 1791-1804 (2009)[PubMed]
- O'Maoileidigh, D. S., Wuest, S. E., Rae, L. et al.: Control of reproductive floral organ identity specification in Arabidopsis by the C function regulator AGAMOUS. Plant Cell, 25, 2482-2503 (2013)[PubMed]
- Wuest, S. E., Maoileidigh, D. S. O., Rae, L. et al.: Molecular basis for the specification of floral organs by APETALA3 and PISTILLATA. Proc. Natl. Acad. Sci. USA, 109, 13452-13457 (2012)[PubMed]
- Kaufmann, K., Muino, J. M., Jauregui, R. et al.: Target genes of the MADS transcription factor SEPALLATA3: integration of developmental and hormonal pathways in the Arabidopsis flower. PLoS Biol., 7, e1000090 (2009)[PubMed]
- Kaufmann, K., Wellmer, F., Muino, J. M. et al.: Orchestration of floral initiation by APETALA1. Science, 328, 85-89 (2010)[PubMed]
- Yant, L., Mathieu, J., Dinh, T. T. et al.: Orchestration of the floral transition and floral development in Arabidopsis by the bifunctional transcription factor APETALA2. Plant Cell, 22, 2156-2170 (2010)[PubMed]
- Ito, T., Wellmer, F., Yu, H. et al.: The homeotic protein AGAMOUS controls microsporogenesis by regulation of SPOROCYTELESS. Nature, 430, 356-360 (2004)[PubMed]
- Lenhard, M., Bohnert, A., Jurgens, G. et al.: Termination of stem cell maintenance in Arabidopsis floral meristems by interactions between WUSCHEL and AGAMOUS. Cell, 105, 805-814 (2001)[PubMed]
- Lohmann, J. U., Hong, R. L., Hobe, M. et al.: A molecular link between stem cell regulation and floral patterning in Arabidopsis. Cell, 105, 793-803 (2001)[PubMed]
- Sun, B., Looi, L. S., Guo, S. et al.: Timing mechanism dependent on cell division is invoked by Polycomb eviction in plant stem cells. Science, 343, 1248559 (2014)[PubMed]
- Muller, J. & Kassis, J. A.: Polycomb response elements and targeting of Polycomb group proteins in Drosophila. Curr. Opin. Genet. Dev., 16, 476-484 (2006)[PubMed]
- Hansen, K. H., Bracken, A. P., Pasini, D. et al.: A model for transmission of the H3K27me3 epigenetic mark. Nat. Cell Biol., 10, 1291-1300 (2008)[PubMed]
- Francis, N. J., Follmer, N. E., Simon, M. D. et al.: Polycomb proteins remain bound to chromatin and DNA during DNA replication in vitro. Cell, 137, 110-122 (2009)[PubMed]
- Dodd, I. B., Micheelsen, M. A., Sneppen, K. et al.: Theoretical analysis of epigenetic cell memory by nucleosome modification. Cell, 129, 813-822 (2007)[PubMed]
- Busturia, A., Wightman, C. D. & Sakonju, S.: A silencer is required for maintenance of transcriptional repression throughout Drosophila development. Development, 124, 4343-4350 (1997)[PubMed]
- Sengupta, A. K., Kuhrs, A. & Muller, J.: General transcriptional silencing by a Polycomb response element in Drosophila. Development, 131, 1959-1965 (2004)[PubMed]
- Yagita, K., Horie, K., Koinuma, S. et al.: Development of the circadian oscillator during differentiation of mouse embryonic stem cells in vitro. Proc. Natl. Acad. Sci. USA, 107, 3846-3851 (2010)[PubMed]
著者プロフィール
略歴:1997年 京都大学大学院理学研究科博士課程 修了,同年 米国California Institute of Technology研究員,2003年 同Senior Research Fellow,2005年 シンガポールTemasek Life Sciences LaboratoryにてPrincipal Investigatorを経て,2011年より同Senior Principal Investigator.
抱負:Things do always work out.
研究室URL:http://www.tll.org.sg/group-leaders/toshiro-ito/
© 2014 伊藤 寿朗 Licensed under CC 表示 2.1 日本