植物の窒素固定:植物と窒素固定細菌との共生の進化
2015/07/29
林 誠
(理化学研究所環境資源科学研究センター 植物共生研究チーム)
email:林 誠
領域融合レビュー, 4, e010 (2015) DOI: 10.7875/leading.author.4.e010
Makoto Hayashi: Nitrogen fixation in plants: how plants established symbiosis with nitrogen fixing bacteria during evolution.

作物の栽培においてもっとも多用される肥料は窒素肥料である.戦後の“緑の革命”において,多量の窒素肥料を投入することにより作物の生産は飛躍的に向上し,世界人口の急激的な増加に対応することができた.その反面,窒素肥料の利用は温室効果ガスの排出を促進することから環境に悪影響をおよぼす.ダイズなどのマメ科植物は土壌微生物である根粒菌と共生することにより大気中の窒素分子を固定し利用する.陸上植物の進化の過程においてマメ科植物が根粒菌との共生能を獲得した機構を解明することにより,持続的な循環農業に貢献できると考えられる.
1960年に30億人であった世界人口は,2000年には60億人に達し,2050年には90億人になると推定されている.20世紀後半における人口爆発をささえたのは食料の大幅な増産であり,これを可能にしたのはコムギおよびイネにおける“緑の革命”であった.すなわち,品種改良により草丈を短くし子実の重量の増加にともなう倒伏性を克服したことにより,窒素肥料の過剰な施用に対応したことである.ハーバー法の発明により窒素肥料の生産量は飛躍的に増大したが,これにともない環境への負荷は増大した.アンモニアの合成および尿素などの2次的な生産において温室効果ガスが排出され,さらに,過剰な施肥により作物に吸収されない窒素は河川および近海の富栄養化の原因となり,これは水圏の酸素濃度の低下による生物の大規模な死滅をひき起こす.また,農耕地において余剰な窒素が硝化や脱窒をうけることにより,二酸化炭素のおよそ300倍の地球温暖化係数をもつ一酸化二窒素が発生する.中長期的にみた農業活動の持続と環境保全のためには,収量を担保しつつも窒素肥料の施用を低減することがもとめられる.このためには,品種改良による作物の窒素の利用効率の向上や薬剤による硝化および脱窒の抑制,緩効性の肥料を利用した窒素の施肥技術の改善などがあげられるが,抜本的かつ効率的な解決策として,共生細菌による生物学的な窒素固定の利用が考えられる.生物学的な窒素固定は一部の原核生物のみにみられ,ニトロゲナーゼのはたらきにより大気中の窒素分子をアンモニアに変換する.すなわち,共生的な窒素固定を利用することにより窒素肥料への依存から脱却することが可能になり,したがって,窒素肥料の施用にかかわる温室効果ガスの排出を大幅に削減することにつながる.
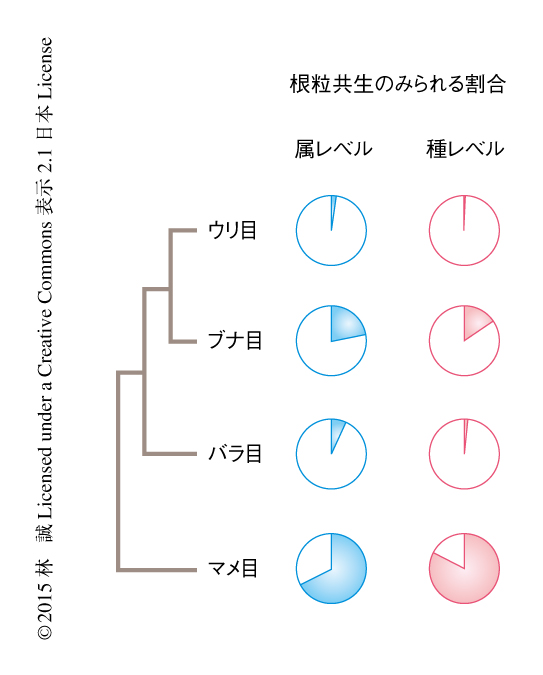
窒素固定細菌との明確な共生の知られている種はかぎられており,根粒における細胞内共生は,窒素固定クレードとよばれる進化的に単一の分類群(すなわち,単系統群)に属する種のみにみられる1).窒素固定クレードはマメ目,ブナ目,ウリ目,バラ目に分類され,それぞれの分類群に根粒共生能が認められる(図1).しかしながら,その系統的な分布は不均一であり,マメ目ではマメ科に属する種に普遍的に根粒共生がみられるのに対し,ウリ目,ブナ目,バラ目には根粒共生のみられる種は少ない.また,マメ科植物はグラム陰性細菌である根粒菌と共生するのに対し,ほかの窒素固定クレードの植物は,根粒菌と共生するバラ目のParasponia属を除き,グラム陽性細菌である放線菌のFrankia属細菌と共生する(Frankia属細菌の宿主をアクチノリザル植物とよぶ).
このように,根粒共生は窒素固定クレードに散見されることなどから,根粒共生は窒素固定クレードにおいて複数回にわたり独立して進化したと考えられる2).定量的な系統解析によるとこの進化は独立しておよそ8回起こったが,進化の起因となる形質の獲得(プレディスポジョション)は窒素固定クレードの基部において1回のみ起こったとされる3).つまり,窒素固定クレードにおけるプレディスポジョションが根粒共生の前提条件になったのである.いい換えると,根粒における窒素固定細菌との細胞内共生は収斂進化の側面があり,同様の機能を異なった遺伝子が担う場合がある.逆に,プレディスポジションにもとづく進化は遺伝的な制約により相同遺伝子を利用している可能性が高い.そこで,プレディスポジションの起因になる遺伝子の進化について明らかにされれば,そののちに起こった根粒共生の進化についての洞察も得られると想定される.さらに,根粒共生における相同性および相似性を遺伝子レベルで解析しその進化を理解することにより,窒素固定クレードとは系統的に離れた主要穀物であるイネ,コムギ,トウモロコシに根粒共生能を付与することが可能になると考えられる.このレビューにおいては,マメ科のモデル植物ミヤコグサ(Lotus japonicus)およびタルウマゴヤシ(Medicago truncatula)において明らかにされた根粒の形成にかかわる遺伝子を軸として,窒素固定クレードにおける根粒の形成の進化について考察する.
コケ植物やシダ植物を含む陸上植物の7割以上の種は,近年になり確立されたグロムス門に属する真菌であるアーバスキュラー菌根菌と共生する4).宿主の系統関係から植物の陸上化にともない共生が確立されたと考えられており,すでに4億年以上まえの化石においてアーバスキュラー菌根菌が感染したと考えられる構造が認められる5).窒素固定細菌との共生とは対照的に,アーバスキュラー菌根菌は根圏に発達させた外生菌糸のはたらきにより宿主に水分やミネラル,とくにリンを供給する.
アーバスキュラー菌根菌との共生の起源が古いこと,および,共生の宿主特異性が低いことから,宿主のもつ共生遺伝子は陸上植物に広く保存されていると考えられる.マメ科のモデル植物の研究において明らかにされたアーバスキュラー菌根菌との共生に必要な主要な遺伝子は,イネとアーバスキュラー菌根菌との共生にも必要であることが遺伝学的に立証されている6-9).逆に,アーバスキュラー菌根菌との共生能を失ったアブラナ科の植物などではこれらの遺伝子が多く失われていることから,これらの遺伝子は共生に特異的な機能を保持していると考えられる10).これらの遺伝子はマメ科植物においてはアーバスキュラー菌根菌との共生のみならず根粒菌との共生にも必要であり,根粒共生を開始するための細胞内シグナル伝達経路である共通共生経路を構成する共通共生遺伝子である4)(ここで“共通”というのは,菌根共生と根粒共生とのあいだにおける遺伝的な共通性のことである).興味深いことに,イネの共通共生遺伝子は,程度の差こそあれ,基本的にマメ科植物における根粒共生においても機能する6,9,11-14).したがって,根粒共生の進化をへていない窒素固定クレード以外の種においても共通共生遺伝子の機能は保存されている.逆の見方をすると,根粒共生の進化において共通共生遺伝子は進化していないことになる.実際に,マメ科植物とは独立して根粒共生能を獲得したと考えられるアクチノリザル植物においても同様に,共通共生遺伝子はアーバスキュラー菌根菌との共生のみならず根粒の形成にも必要であることが示されている12,15,16).以上のことから,窒素固定クレードにおける根粒共生の進化は,すでに確立された菌根共生における機構(すなわち,共通共生経路)をそのまま利用したと考えられる17).
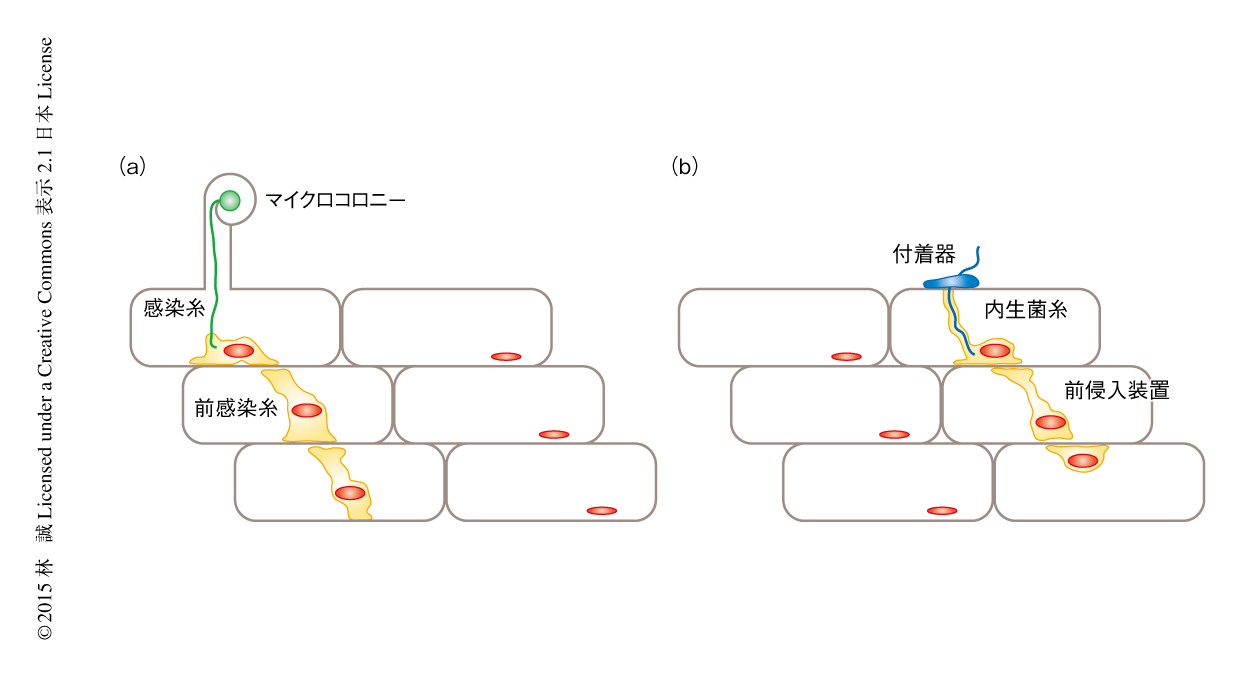
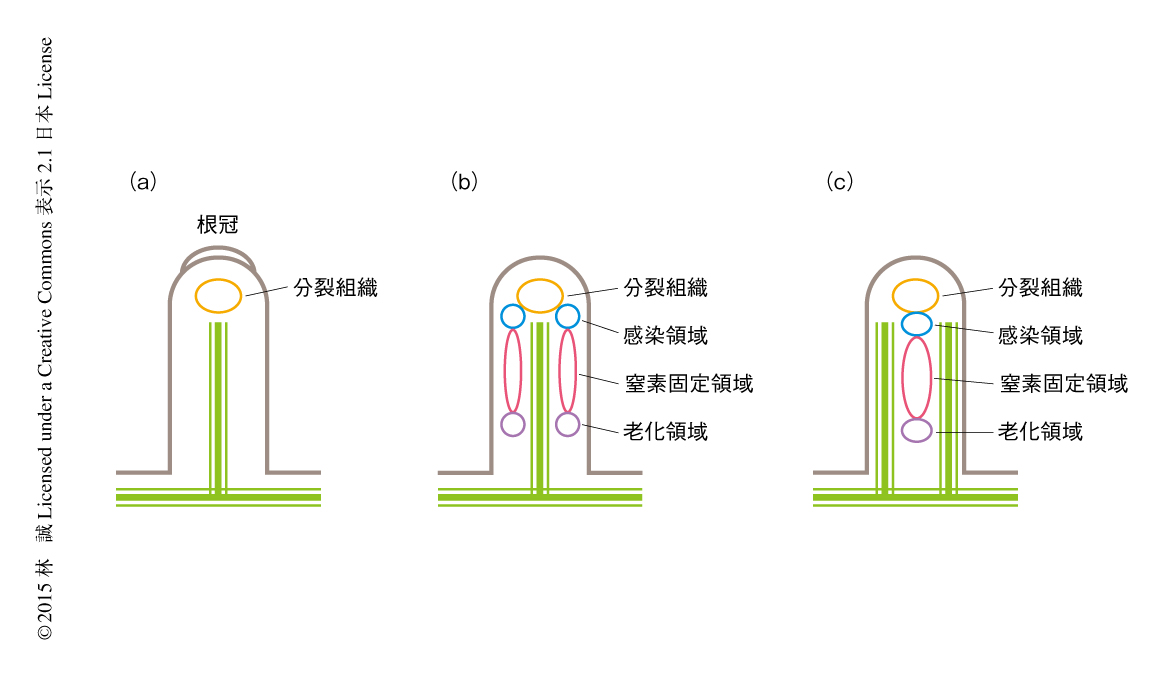
根粒菌との共生において共通共生経路のはたす役割は,根の表皮における根粒菌の感染および皮層における根粒原基の形成の誘導である.これに対して,アーバスキュラー菌根菌との共生において共通共生経路のはたす役割は,表皮における付着器からの内生菌糸の形成および皮層における内生菌糸の侵入の前段階である細胞質の再構成である4,18-21).すなわち,共通共生経路はそれを構成する遺伝子のみならず,その機能においても根粒共生と菌根共生とのあいだで相同であるといえる(図2).したがって,共通共生遺伝子は根粒共生の進化において重要な役割をはたしたものの,進化の直接の起因とは考えにくい.すなわち,根粒共生におけるプレディスポジョションを明らかにするためには,共通共生経路以外の因子が共生においてはたす役割をみていく必要がある.
根粒菌との共生では,共通共生経路の上流において根粒菌の分泌する共生シグナルであるNod因子の受容が機能する.Nod因子はアシル基の付加された短鎖キチンオリゴ糖(LCO)であり,根粒菌の種により,キチンオリゴ糖の重合度(3~5糖),非還元末端に付加されたアシル基の鎖長および不飽和度,還元末端および非還元末端の修飾基に違いがあり,根粒菌の宿主特異性を決定する要因のひとつになっている22).Nod因子は宿主の根の表皮の細胞膜に局在する2種のLysM型受容体キナーゼ(ミヤコグサNFR5/タルウマゴヤシNFP,および,ミヤコグサNFR1/タルウマゴヤシLYK3)により受容されると考えられている23-26).この受容体はヘテロマーを形成しており27-29),NFR5/NFPのLysMドメインの多様性はさきに述べた宿主特異性を決定している30,31).NFR1はNFR5と同様に生理学的な活性濃度のNod因子と結合する32,33).NFR1/LYK3は自己リン酸化活性を示すのに対し,NFR5/NFPには自己リン酸化活性が失われているため,受容体の相互作用が共通共生経路にいたるシグナル伝達において重要であると考えられる25,27).
アーバスキュラー菌根菌は多核で遺伝学的な手法が確立されていないこともあり,その共生シグナルは長いあいだ未知であったが,最近になり,アーバスキュラー菌根菌がNod因子に類似した構造の複数の化合物を分泌することが示された34).興味深いことに,この化合物(Myc-LCO)はタルウマゴヤシにおいてアーバスキュラー菌根菌の感染を促進するのみならず,側根の発達を促進する34).側根の発達はNod因子によっても促進されるが,この促進は共通共生経路に依存するのみならず,Nod因子の特異的な受容体として考えられてきたNFPにも依存する34,35).しかしながら,イネにおけるアーバスキュラー菌根菌あるいはアーバスキュラー菌根菌の胞子の浸出液による側根の発達の促進は共通共生経路に依存しないこと36,37),アーバスキュラー菌根菌の感染にNFPは必要とされないことなどから38,39),Myc-LCOのみがアーバスキュラー菌根菌の共生応答に必要とされるかどうかについては疑問の余地がある.
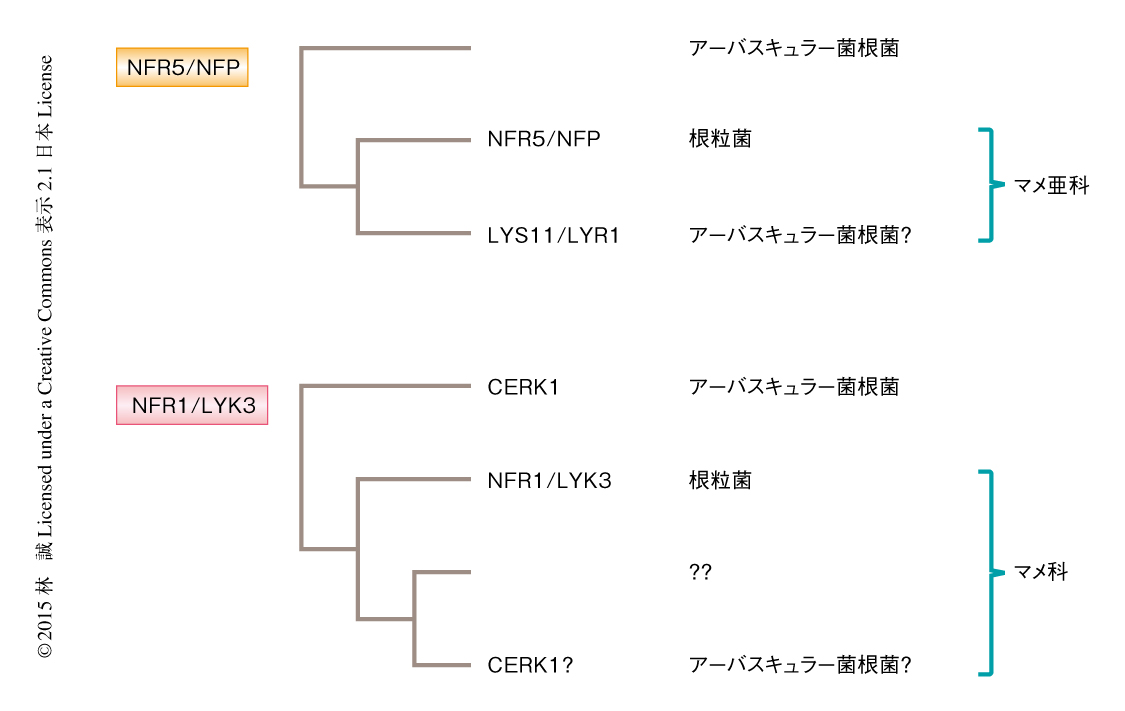
NFR5/NFPはマメ亜科における全ゲノム重複により生じており40,41),そのParasponia属におけるオーソログは根粒共生および菌根共生の両方に関与することから42),タルウマゴヤシやミヤコグサなどのマメ亜科では重複したパラログの一方(すなわち,NFR5/NFP)がNod因子の受容体として進化し,もう一方(アーバスキュラー菌根菌の接種により遺伝子の発現が誘導されるLYS11/LYR1)がMyc-LCOの受容体として機能を分担した可能性がある40,42)(図3).マメ科の基部においてマメ亜科と分岐したジャケツイバラ亜科のヤマウズラエンドウ(Chamaecrista fasciculata)においてはマメ亜科にみられた全ゲノム重複が認められず41),Parasponia属と同様にNFR5/NFPのオーソログは単一の遺伝子にコードされており42),根粒共生がみられることから,この遺伝子が根粒共生および菌根共生の両方に関与しているかどうかは興味深いところである.なぜならば,Parasponia属やヤマウズラエンドウにおいてNFR5/NFPのオーソログが根粒菌およびアーバスキュラー菌根菌に由来する異なる分子種のLCOを受容することにより,マメ亜科(ミヤコグサ,タルウマゴヤシ,エンドウ)においてみられたNod因子の構造に特異的な認識は起こらず43),これはすなわち,窒素固定クレードのみならず,潜在的にはアーバスキュラー菌根菌の宿主の全般において根粒菌(に由来する共生シグナル)を認識できることになるからである.実際に,被子植物には根粒共生とは独立にLCOの受容体が存在すると考えてもよい報告もあり44),LCOの機能とその受容についてはさらなる研究が必要である.
一方,Nod因子とは異なり修飾基の付加されていない短鎖キチンオリゴ糖(4~5糖)がアーバスキュラー菌根菌の共生シグナルとして認識され共通共生経路の活性化につながるという報告もある45,46).この応答にはNFPあるいはNFR1/LYK3は関与しないこと39,45,46),そもそも,NFR5およびNFR1の短鎖キチンオリゴ糖に対する結合能は低いことから32,33),Nod因子の受容体とは別の受容体(複合体)が重要であると考えられる.ごく最近になり,キチンオリゴ糖(6~8糖)の認識に関与するLysM型受容体キナーゼであるCERK1がアーバスキュラー菌根菌の共生にも影響することが明らかにされた39,47).このキチンオリゴ糖は植物病原性の真菌を認識する際のエリシターとして作用するが,CERK1は植物病原細菌に由来するエリシターとしてのペプチドグリカンの受容にも関与していること,キチンオリゴ糖に対する結合能は高くないことなどから,実際の受容体ではなく受容体と相互作用するアダプターとして機能していると考えられている48).現在のところ,CERK1がMyc-LCOあるいは短鎖キチンオリゴ糖のどちらの認識に関与しているかは不明であるが,エリシターの認識と同様にアダプターとしてはたらいている可能性もある.
マメ科植物以外のLysM型受容体キナーゼのうち,マメ科植物のNFR1/LYK3にもっとも相同性の高いものはCERK1であるが,マメ科においてはCERK1の遺伝子重複により3つのクレードが形成されており,そのうちのひとつがNod因子の認識に,もうひとつがキチンオリゴ糖の認識に関与していると考えられている49)(図3).NFR1/LYK3およびCERK1のキナーゼドメインは保存されており,実際に,ドメインスワップによりNFR1のキナーゼドメインをCERK1のキナーゼドメインと代替することができる47,49)(ただし,アーバスキュラー菌根菌との共生のみられる植物に由来するCERK1のみ).このことは,CERK1の下流およびNFR1の下流が共通共生経路として保存されており,相互作用の影響によりキナーゼドメインに対する負の選択圧がみられることを示唆している49).NFR5/NFPと同様に,Parasponia属のCERK1はアーバスキュラー菌根菌との共生のみならず根粒菌との共生にも重要であるのか,今後の解析が待たれる.
以上を総合すると,アーバスキュラー菌根菌の共生に関与する共生シグナルの受容については不明な点が多いものの,受容体としてLysM型受容体キナーゼの関与していることは確からしい.また,それらの少なくとも一部についてはマメ科植物におけるNod因子の受容に進化したと想定される.しかしながら,マメ科植物における根粒共生とは独立に進化した,アクチノリザル植物の根粒共生における共生シグナルについては,主要なFrankia属細菌のゲノム情報においてはLCOを合成する遺伝子がみあたらないことからも50,51),根粒共生におけるLysM型受容体キナーゼの進化は根粒菌に特異的であってプレディスポジョションの要因とは考えにくい.つまり,マメ科植物における共生シグナルの受容とアクチノリザル植物における共生シグナルの受容は収斂進化の結果であると考えてよいだろう.
共通共生経路の下流では根粒の形成に必要な遺伝子の転写が活性化され,多くの遺伝子の発現が誘導されている.これまでに,マメ科のモデル植物において根粒の形成のみに関与する転写因子様のタンパク質がいくつか遺伝学的に同定されてきたが,機能欠失変異により根粒の形成の最初の兆候である皮層細胞の分裂が誘導されない遺伝子はNIN遺伝子のみである.したがって,根粒共生の進化におけるプレディスポジションの実体としてNIN遺伝子は有力な候補になる.NINはDNA結合ドメインであるRWP-RKをもち,NIN遺伝子は根粒原基の形成の開始に必要な遺伝子のなかでは唯一,根粒菌の接種によりすみやかかつ顕著に発現が上昇する52-55).NIN遺伝子を過剰に発現させると根粒菌の存在にかかわらず根粒様の構造が形成されることから,NINは共通共生経路の下流で機能する根粒の形成におけるマスターレギュレーターであると考えられる56).
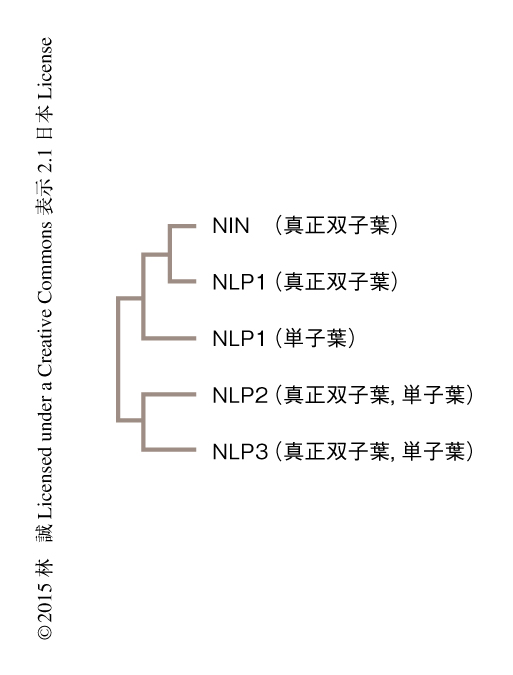
NINは根粒の形成に特異的な転写因子であると考えられているが,そのホモログであるNLPは(少なくとも)被子植物において広く保存されている57-59)(図4).NLPは3つのサブファミリーに分類され,NINはNLP1クレードに属する57,59).興味深いことに,NLP1はバラ類およびキク類を含む真正双子葉類の基部において分岐しており,そのNINサブクレードは窒素固定クレードに属する種のみならず,バラ類およびキク類の全般においてホモログが認められる59).根粒共生は窒素固定クレードのみにみられることから,真正双子葉類の基部において根粒共生のプレディスポジョションが獲得されたとは考えにくく,NLP1からNINが進化した過程においてNINは別の機能を進化させていたと想定される.NLPの機能はその命名からしばらくのあいだ知られていなかったが57),最近になり,硝酸応答のマスターレギュレーターであることが明らかにされた60,61).シロイヌナズナのゲノムには9つのNLPのホモログが認められるが,そのうち,NLP3クレードに属するシロイヌナズナNLP7の遺伝子欠損は窒素欠乏の表現型を示し62),硝酸のシグナル伝達および同化に関係する多くの遺伝子の転写制御領域に結合することが示された60).シロイヌナズナNLP7以外のホモログについても硝酸応答配列に結合することが明らかにされている61).興味深いことに,硝酸応答配列はNINが結合するシス配列と相同性を示し,実際に,NINは硝酸応答配列と結合することから,NINを含むNLPファミリーにおいてDNA結合ドメインであるRWP-RKは機能的に保存されていると考えられる63,64).ところが,根粒の形成は硝酸により抑制され,また,根粒菌が感染しても主要な硝酸応答遺伝子の発現は誘導されないことから,硝酸応答を正に制御するNLPと根粒の形成を正に制御するNINは転写制御領域への結合に対し拮抗的に作用しているのかもしれない64).
根粒の形成(根粒菌の感染)はさらなる根粒の形成を負に制御する65).これは植物の生存戦略からみると合理的であり,必要以上に根粒を形成することはそれだけ光合成産物を消費することになり宿主に負担が生じる.実際に,このフィードバックが破綻した変異体においては根粒菌の感染に依存して,とくに地上部の生育がいちじるしく阻害される66).同様に,十分な硝酸が得られる条件では根粒における窒素固定も必要ないことから,硝酸による根粒の形成の抑制も合理的である.根粒菌による根粒の形成の抑制にもまたNINが関与している.根粒菌が感染するとNINのもつ転写活性によりCLEペプチドをコードする遺伝子の発現が誘導される67).ここには2種類のCLEペプチドが関与するが,そのうちのひとつの発現は硝酸によっても誘導される68).生成したCLEペプチドは地上部へと輸送されLRR型受容体キナーゼHAR1のリガンドとしてはたらく69).HAR1の下流において地上部におけるサイトカイニンの合成が促進され,最終的に根における根粒の形成を抑制する70).
ミヤコグサHAR1はシロイヌナズナCLV1のオーソログであり,CLV1はCLEペプチドであるCLV3を認識することにより茎頂分裂組織の大きさを負に制御する.側根の形成における硝酸応答にはCLV1が関与しており71),HAR1による根粒の形成の制御も硝酸応答から進化したのかもしれない.実際に,さきに述べたようにHAR1は硝酸に依存して根粒の数を負に制御するほか,側根の数も負に制御する66).そもそも,硝酸応答は側根の形成の制御に重要な役割をはたしており72),たとえば,シロイヌナズナNLP7は側根の形成にも影響する62).また,シロイヌナズナにおいて,硝酸の欠乏により発現が誘導されるCEP1ペプチドに結合するLRR型受容体キナーゼCEPRは,側根の形成を負に制御する73).同様の関係はタルウマゴヤシにも存在するが,根粒の形成は正に制御されることから側根の形成と根粒の形成とが拮抗する関係になっている74,75).
以上に述べたように,根粒の形成のマスターレギュレーターであるNINは硝酸応答のマスターレギュレーターであるNLPから進化し,硝酸応答は側根の形成と密接な関係のあることから,根粒の形成の進化においてNLPが制御する側根の形成プログラムを部分的に流用した可能性がある59).したがって,NINが転写を制御する下流の遺伝子のなかに側根の形成に関与する遺伝子があってもおかしくない.
NINはNF-YのサブユニットをコードするNF-YA1遺伝子およびNF-YB1遺伝子の発現を正に制御する56).NF-YはCCAATボックスに結合する転写因子であり,これらの遺伝子を過剰に発現させると皮層の細胞分裂が促進されるとともに異常な側根の形成が誘導される56).逆に,NF-YA1遺伝子の発現を抑制すると根粒の形成が阻害される56,76).マメ科においてはNF-YA1の属するクレードが重複しており77),NF-YA1のパラログは根粒においては発現が誘導されない78).マメ科植物のNF-YA1のシロイヌナズナにおけるオーソログは側根の形成に関与することから79),マメ科植物においては側根の形成に関与していたNF-Yの遺伝子重複により生じたパラログが,新規にNINの制御をうける転写制御領域を獲得し根粒の形成に関与するようになった可能性がある.実際に,アクチノリザル植物であるグラウカモクマオウ(Casuarina glauca)においては,NINの発現を抑制することにより根粒の形成が阻害されるものの,マメ科植物とは異なり根粒において発現するNF-YA1遺伝子およびNF-YB1遺伝子のホモログの発現には影響しない55).このことは,NINによるNF-Yの発現制御機構はマメ科植物の根粒に特徴的であることを示唆している.根粒の形態を比較すると,アクチノリザル植物の根粒は側根に類似しており(図5),根粒の形成の過程からみても側根の変形したものだと考えられている80).一方,マメ科植物の根粒は,内部の構成はアクチノリザル植物の根粒に類似しているものの,維管束の構成および形成の過程は大きく異なる51).したがって,アクチノリザル植物の根粒はマメ科植物の根粒とは相似であり,つまり,収斂進化の結果であると考えられる2).
以上のことから,マメ科植物における根粒の形成は,進化の過程において複数の制御機構をモジュールとして獲得したことにより成立したと考えられる.その中心と考えられるのが転写因子NINであり,動物における形態形成の進化と同様に81),遺伝子重複などによる転写因子の新規の機能の獲得とともに,その上流および下流における転写制御領域の新規の獲得がモジュールの再結線をうながした結果,新規の器官である根粒の進化につながったと考えられる59).
筆者らは,結論として,根粒共生のプレディスポジョションにおいて重要なはたらきをした実体はNINだと想定している.すでに議論したとおり,Nod因子の受容に関与するLysM型受容体キナーゼは,その由来が菌根共生だとしても主要なFrankia属細菌がNod因子を合成しないことから除外される.また,アーバスキュラー菌根菌との共生にも必要な共通共生遺伝子は,陸上植物に広く保存されていることから対象外である.したがって,現時点で考えられるのはNINのみであり,マメ科植物あるいはアクチノリザル植物を問わずNINは根粒の形成において重要であることから,その下流において誘導される根粒の形成プログラムはマメ科植物とアクチノリザル植物とでは一部が異なると考えられるものの,共通共生経路がNINを含む転写制御機構を獲得したことが鍵であると結論される.NINの祖先型であるNLPは硝酸応答のマスターレギュレーターであり,さきに述べたように,側根の形成は硝酸によっても制御され,根におけるNINの下流の形態形成プログラムはNLPの形態形成プログラムと類似している可能性がある.マメ科植物においては側根の形態形成プログラムをNF-Yの獲得をつうじ新たに転用したことにより,新規の器官である根粒の形成につながったのではないだろうか.
窒素固定クレードの進化はおよそ1億年まえに起こったと考えられているが,根粒共生の進化は6千万年まえより以前にはさかのぼらない2).したがって,少なくとも4千万年のあいだ,窒素固定クレードの祖先種は根粒共生を進化させるポテンシャル(すなわち,プレディスポジョション)は獲得していたものの,実際に根粒共生を進化させることはなかったことになる.すなわち,プレディスポジションの役割は根粒共生ではない未知の形質において重要な役割をはたしていたことになる.しかしながら,さきにも述べた定量的な系統解析によると,根粒共生のプレディスポジションは窒素固定クレードにおいて17回失われていると予測されている3).このことは,窒素固定クレードの放散においてプレディスポジションは重要であったものの,そののち,なんらかの(環境)要因の変化により多くの窒素固定クレードではプレディスポジションに対する選択圧が低下し,結果として,根粒の形成の進化能を失ったのだろう.プレディスポジションの喪失と現生種の生理環境とを比較することにより,プレディスポジョションの本質にせまることが可能かもしれない82).
根粒共生は,ここで述べた根粒の形成のほか,根粒菌の宿主への感染と細胞への取り込み(細胞内共生),共生的な窒素固定,というおおまかには3つの段階にその過程を分けることができ,それぞれの段階において,遺伝子導入などによる検証をふまえた合成生物学的なアプローチにより主要穀物における再構成が可能であると考えている.根粒の形成については近年の分子遺伝学の成果から概略がつかめているが,根粒菌の感染および細胞内共生と共生的な窒素固定についてはさらなる知見が必要である.今後,イネ科植物における根粒形成の再構成と並行して,マメ科のモデル植物において根粒菌との共生の全貌を分子遺伝学的に明らかにすることが重要である.
略歴:1996年 東京大学大学院理学系研究科博士課程 修了,1997年 大阪大学大学院工学研究科 助手,2006年 ドイツMunich大学 教授,2008年 農業生物資源研究所植物科学研究領域 ユニット長を経て,2014年より理化学研究所環境資源科学研究センター チームリーダー.
研究テーマ:植物の形態形成.
関心事:生物の多様性.
研究室URL:http://www.riken.jp/research/labs/csrs/plant_symb/
© 2015 林 誠 Licensed under CC 表示 2.1 日本
(理化学研究所環境資源科学研究センター 植物共生研究チーム)
email:林 誠
領域融合レビュー, 4, e010 (2015) DOI: 10.7875/leading.author.4.e010
Makoto Hayashi: Nitrogen fixation in plants: how plants established symbiosis with nitrogen fixing bacteria during evolution.
要 約
作物の栽培においてもっとも多用される肥料は窒素肥料である.戦後の“緑の革命”において,多量の窒素肥料を投入することにより作物の生産は飛躍的に向上し,世界人口の急激的な増加に対応することができた.その反面,窒素肥料の利用は温室効果ガスの排出を促進することから環境に悪影響をおよぼす.ダイズなどのマメ科植物は土壌微生物である根粒菌と共生することにより大気中の窒素分子を固定し利用する.陸上植物の進化の過程においてマメ科植物が根粒菌との共生能を獲得した機構を解明することにより,持続的な循環農業に貢献できると考えられる.
はじめに
1960年に30億人であった世界人口は,2000年には60億人に達し,2050年には90億人になると推定されている.20世紀後半における人口爆発をささえたのは食料の大幅な増産であり,これを可能にしたのはコムギおよびイネにおける“緑の革命”であった.すなわち,品種改良により草丈を短くし子実の重量の増加にともなう倒伏性を克服したことにより,窒素肥料の過剰な施用に対応したことである.ハーバー法の発明により窒素肥料の生産量は飛躍的に増大したが,これにともない環境への負荷は増大した.アンモニアの合成および尿素などの2次的な生産において温室効果ガスが排出され,さらに,過剰な施肥により作物に吸収されない窒素は河川および近海の富栄養化の原因となり,これは水圏の酸素濃度の低下による生物の大規模な死滅をひき起こす.また,農耕地において余剰な窒素が硝化や脱窒をうけることにより,二酸化炭素のおよそ300倍の地球温暖化係数をもつ一酸化二窒素が発生する.中長期的にみた農業活動の持続と環境保全のためには,収量を担保しつつも窒素肥料の施用を低減することがもとめられる.このためには,品種改良による作物の窒素の利用効率の向上や薬剤による硝化および脱窒の抑制,緩効性の肥料を利用した窒素の施肥技術の改善などがあげられるが,抜本的かつ効率的な解決策として,共生細菌による生物学的な窒素固定の利用が考えられる.生物学的な窒素固定は一部の原核生物のみにみられ,ニトロゲナーゼのはたらきにより大気中の窒素分子をアンモニアに変換する.すなわち,共生的な窒素固定を利用することにより窒素肥料への依存から脱却することが可能になり,したがって,窒素肥料の施用にかかわる温室効果ガスの排出を大幅に削減することにつながる.
窒素固定細菌との明確な共生の知られている種はかぎられており,根粒における細胞内共生は,窒素固定クレードとよばれる進化的に単一の分類群(すなわち,単系統群)に属する種のみにみられる1).窒素固定クレードはマメ目,ブナ目,ウリ目,バラ目に分類され,それぞれの分類群に根粒共生能が認められる(図1).しかしながら,その系統的な分布は不均一であり,マメ目ではマメ科に属する種に普遍的に根粒共生がみられるのに対し,ウリ目,ブナ目,バラ目には根粒共生のみられる種は少ない.また,マメ科植物はグラム陰性細菌である根粒菌と共生するのに対し,ほかの窒素固定クレードの植物は,根粒菌と共生するバラ目のParasponia属を除き,グラム陽性細菌である放線菌のFrankia属細菌と共生する(Frankia属細菌の宿主をアクチノリザル植物とよぶ).
このように,根粒共生は窒素固定クレードに散見されることなどから,根粒共生は窒素固定クレードにおいて複数回にわたり独立して進化したと考えられる2).定量的な系統解析によるとこの進化は独立しておよそ8回起こったが,進化の起因となる形質の獲得(プレディスポジョション)は窒素固定クレードの基部において1回のみ起こったとされる3).つまり,窒素固定クレードにおけるプレディスポジョションが根粒共生の前提条件になったのである.いい換えると,根粒における窒素固定細菌との細胞内共生は収斂進化の側面があり,同様の機能を異なった遺伝子が担う場合がある.逆に,プレディスポジションにもとづく進化は遺伝的な制約により相同遺伝子を利用している可能性が高い.そこで,プレディスポジションの起因になる遺伝子の進化について明らかにされれば,そののちに起こった根粒共生の進化についての洞察も得られると想定される.さらに,根粒共生における相同性および相似性を遺伝子レベルで解析しその進化を理解することにより,窒素固定クレードとは系統的に離れた主要穀物であるイネ,コムギ,トウモロコシに根粒共生能を付与することが可能になると考えられる.このレビューにおいては,マメ科のモデル植物ミヤコグサ(Lotus japonicus)およびタルウマゴヤシ(Medicago truncatula)において明らかにされた根粒の形成にかかわる遺伝子を軸として,窒素固定クレードにおける根粒の形成の進化について考察する.
1.菌根共生と根粒共生
コケ植物やシダ植物を含む陸上植物の7割以上の種は,近年になり確立されたグロムス門に属する真菌であるアーバスキュラー菌根菌と共生する4).宿主の系統関係から植物の陸上化にともない共生が確立されたと考えられており,すでに4億年以上まえの化石においてアーバスキュラー菌根菌が感染したと考えられる構造が認められる5).窒素固定細菌との共生とは対照的に,アーバスキュラー菌根菌は根圏に発達させた外生菌糸のはたらきにより宿主に水分やミネラル,とくにリンを供給する.
アーバスキュラー菌根菌との共生の起源が古いこと,および,共生の宿主特異性が低いことから,宿主のもつ共生遺伝子は陸上植物に広く保存されていると考えられる.マメ科のモデル植物の研究において明らかにされたアーバスキュラー菌根菌との共生に必要な主要な遺伝子は,イネとアーバスキュラー菌根菌との共生にも必要であることが遺伝学的に立証されている6-9).逆に,アーバスキュラー菌根菌との共生能を失ったアブラナ科の植物などではこれらの遺伝子が多く失われていることから,これらの遺伝子は共生に特異的な機能を保持していると考えられる10).これらの遺伝子はマメ科植物においてはアーバスキュラー菌根菌との共生のみならず根粒菌との共生にも必要であり,根粒共生を開始するための細胞内シグナル伝達経路である共通共生経路を構成する共通共生遺伝子である4)(ここで“共通”というのは,菌根共生と根粒共生とのあいだにおける遺伝的な共通性のことである).興味深いことに,イネの共通共生遺伝子は,程度の差こそあれ,基本的にマメ科植物における根粒共生においても機能する6,9,11-14).したがって,根粒共生の進化をへていない窒素固定クレード以外の種においても共通共生遺伝子の機能は保存されている.逆の見方をすると,根粒共生の進化において共通共生遺伝子は進化していないことになる.実際に,マメ科植物とは独立して根粒共生能を獲得したと考えられるアクチノリザル植物においても同様に,共通共生遺伝子はアーバスキュラー菌根菌との共生のみならず根粒の形成にも必要であることが示されている12,15,16).以上のことから,窒素固定クレードにおける根粒共生の進化は,すでに確立された菌根共生における機構(すなわち,共通共生経路)をそのまま利用したと考えられる17).
根粒菌との共生において共通共生経路のはたす役割は,根の表皮における根粒菌の感染および皮層における根粒原基の形成の誘導である.これに対して,アーバスキュラー菌根菌との共生において共通共生経路のはたす役割は,表皮における付着器からの内生菌糸の形成および皮層における内生菌糸の侵入の前段階である細胞質の再構成である4,18-21).すなわち,共通共生経路はそれを構成する遺伝子のみならず,その機能においても根粒共生と菌根共生とのあいだで相同であるといえる(図2).したがって,共通共生遺伝子は根粒共生の進化において重要な役割をはたしたものの,進化の直接の起因とは考えにくい.すなわち,根粒共生におけるプレディスポジョションを明らかにするためには,共通共生経路以外の因子が共生においてはたす役割をみていく必要がある.
2.宿主による共生シグナルの受容
根粒菌との共生では,共通共生経路の上流において根粒菌の分泌する共生シグナルであるNod因子の受容が機能する.Nod因子はアシル基の付加された短鎖キチンオリゴ糖(LCO)であり,根粒菌の種により,キチンオリゴ糖の重合度(3~5糖),非還元末端に付加されたアシル基の鎖長および不飽和度,還元末端および非還元末端の修飾基に違いがあり,根粒菌の宿主特異性を決定する要因のひとつになっている22).Nod因子は宿主の根の表皮の細胞膜に局在する2種のLysM型受容体キナーゼ(ミヤコグサNFR5/タルウマゴヤシNFP,および,ミヤコグサNFR1/タルウマゴヤシLYK3)により受容されると考えられている23-26).この受容体はヘテロマーを形成しており27-29),NFR5/NFPのLysMドメインの多様性はさきに述べた宿主特異性を決定している30,31).NFR1はNFR5と同様に生理学的な活性濃度のNod因子と結合する32,33).NFR1/LYK3は自己リン酸化活性を示すのに対し,NFR5/NFPには自己リン酸化活性が失われているため,受容体の相互作用が共通共生経路にいたるシグナル伝達において重要であると考えられる25,27).
アーバスキュラー菌根菌は多核で遺伝学的な手法が確立されていないこともあり,その共生シグナルは長いあいだ未知であったが,最近になり,アーバスキュラー菌根菌がNod因子に類似した構造の複数の化合物を分泌することが示された34).興味深いことに,この化合物(Myc-LCO)はタルウマゴヤシにおいてアーバスキュラー菌根菌の感染を促進するのみならず,側根の発達を促進する34).側根の発達はNod因子によっても促進されるが,この促進は共通共生経路に依存するのみならず,Nod因子の特異的な受容体として考えられてきたNFPにも依存する34,35).しかしながら,イネにおけるアーバスキュラー菌根菌あるいはアーバスキュラー菌根菌の胞子の浸出液による側根の発達の促進は共通共生経路に依存しないこと36,37),アーバスキュラー菌根菌の感染にNFPは必要とされないことなどから38,39),Myc-LCOのみがアーバスキュラー菌根菌の共生応答に必要とされるかどうかについては疑問の余地がある.
NFR5/NFPはマメ亜科における全ゲノム重複により生じており40,41),そのParasponia属におけるオーソログは根粒共生および菌根共生の両方に関与することから42),タルウマゴヤシやミヤコグサなどのマメ亜科では重複したパラログの一方(すなわち,NFR5/NFP)がNod因子の受容体として進化し,もう一方(アーバスキュラー菌根菌の接種により遺伝子の発現が誘導されるLYS11/LYR1)がMyc-LCOの受容体として機能を分担した可能性がある40,42)(図3).マメ科の基部においてマメ亜科と分岐したジャケツイバラ亜科のヤマウズラエンドウ(Chamaecrista fasciculata)においてはマメ亜科にみられた全ゲノム重複が認められず41),Parasponia属と同様にNFR5/NFPのオーソログは単一の遺伝子にコードされており42),根粒共生がみられることから,この遺伝子が根粒共生および菌根共生の両方に関与しているかどうかは興味深いところである.なぜならば,Parasponia属やヤマウズラエンドウにおいてNFR5/NFPのオーソログが根粒菌およびアーバスキュラー菌根菌に由来する異なる分子種のLCOを受容することにより,マメ亜科(ミヤコグサ,タルウマゴヤシ,エンドウ)においてみられたNod因子の構造に特異的な認識は起こらず43),これはすなわち,窒素固定クレードのみならず,潜在的にはアーバスキュラー菌根菌の宿主の全般において根粒菌(に由来する共生シグナル)を認識できることになるからである.実際に,被子植物には根粒共生とは独立にLCOの受容体が存在すると考えてもよい報告もあり44),LCOの機能とその受容についてはさらなる研究が必要である.
一方,Nod因子とは異なり修飾基の付加されていない短鎖キチンオリゴ糖(4~5糖)がアーバスキュラー菌根菌の共生シグナルとして認識され共通共生経路の活性化につながるという報告もある45,46).この応答にはNFPあるいはNFR1/LYK3は関与しないこと39,45,46),そもそも,NFR5およびNFR1の短鎖キチンオリゴ糖に対する結合能は低いことから32,33),Nod因子の受容体とは別の受容体(複合体)が重要であると考えられる.ごく最近になり,キチンオリゴ糖(6~8糖)の認識に関与するLysM型受容体キナーゼであるCERK1がアーバスキュラー菌根菌の共生にも影響することが明らかにされた39,47).このキチンオリゴ糖は植物病原性の真菌を認識する際のエリシターとして作用するが,CERK1は植物病原細菌に由来するエリシターとしてのペプチドグリカンの受容にも関与していること,キチンオリゴ糖に対する結合能は高くないことなどから,実際の受容体ではなく受容体と相互作用するアダプターとして機能していると考えられている48).現在のところ,CERK1がMyc-LCOあるいは短鎖キチンオリゴ糖のどちらの認識に関与しているかは不明であるが,エリシターの認識と同様にアダプターとしてはたらいている可能性もある.
マメ科植物以外のLysM型受容体キナーゼのうち,マメ科植物のNFR1/LYK3にもっとも相同性の高いものはCERK1であるが,マメ科においてはCERK1の遺伝子重複により3つのクレードが形成されており,そのうちのひとつがNod因子の認識に,もうひとつがキチンオリゴ糖の認識に関与していると考えられている49)(図3).NFR1/LYK3およびCERK1のキナーゼドメインは保存されており,実際に,ドメインスワップによりNFR1のキナーゼドメインをCERK1のキナーゼドメインと代替することができる47,49)(ただし,アーバスキュラー菌根菌との共生のみられる植物に由来するCERK1のみ).このことは,CERK1の下流およびNFR1の下流が共通共生経路として保存されており,相互作用の影響によりキナーゼドメインに対する負の選択圧がみられることを示唆している49).NFR5/NFPと同様に,Parasponia属のCERK1はアーバスキュラー菌根菌との共生のみならず根粒菌との共生にも重要であるのか,今後の解析が待たれる.
以上を総合すると,アーバスキュラー菌根菌の共生に関与する共生シグナルの受容については不明な点が多いものの,受容体としてLysM型受容体キナーゼの関与していることは確からしい.また,それらの少なくとも一部についてはマメ科植物におけるNod因子の受容に進化したと想定される.しかしながら,マメ科植物における根粒共生とは独立に進化した,アクチノリザル植物の根粒共生における共生シグナルについては,主要なFrankia属細菌のゲノム情報においてはLCOを合成する遺伝子がみあたらないことからも50,51),根粒共生におけるLysM型受容体キナーゼの進化は根粒菌に特異的であってプレディスポジョションの要因とは考えにくい.つまり,マメ科植物における共生シグナルの受容とアクチノリザル植物における共生シグナルの受容は収斂進化の結果であると考えてよいだろう.
3.硝酸応答と根粒の形成
共通共生経路の下流では根粒の形成に必要な遺伝子の転写が活性化され,多くの遺伝子の発現が誘導されている.これまでに,マメ科のモデル植物において根粒の形成のみに関与する転写因子様のタンパク質がいくつか遺伝学的に同定されてきたが,機能欠失変異により根粒の形成の最初の兆候である皮層細胞の分裂が誘導されない遺伝子はNIN遺伝子のみである.したがって,根粒共生の進化におけるプレディスポジションの実体としてNIN遺伝子は有力な候補になる.NINはDNA結合ドメインであるRWP-RKをもち,NIN遺伝子は根粒原基の形成の開始に必要な遺伝子のなかでは唯一,根粒菌の接種によりすみやかかつ顕著に発現が上昇する52-55).NIN遺伝子を過剰に発現させると根粒菌の存在にかかわらず根粒様の構造が形成されることから,NINは共通共生経路の下流で機能する根粒の形成におけるマスターレギュレーターであると考えられる56).
NINは根粒の形成に特異的な転写因子であると考えられているが,そのホモログであるNLPは(少なくとも)被子植物において広く保存されている57-59)(図4).NLPは3つのサブファミリーに分類され,NINはNLP1クレードに属する57,59).興味深いことに,NLP1はバラ類およびキク類を含む真正双子葉類の基部において分岐しており,そのNINサブクレードは窒素固定クレードに属する種のみならず,バラ類およびキク類の全般においてホモログが認められる59).根粒共生は窒素固定クレードのみにみられることから,真正双子葉類の基部において根粒共生のプレディスポジョションが獲得されたとは考えにくく,NLP1からNINが進化した過程においてNINは別の機能を進化させていたと想定される.NLPの機能はその命名からしばらくのあいだ知られていなかったが57),最近になり,硝酸応答のマスターレギュレーターであることが明らかにされた60,61).シロイヌナズナのゲノムには9つのNLPのホモログが認められるが,そのうち,NLP3クレードに属するシロイヌナズナNLP7の遺伝子欠損は窒素欠乏の表現型を示し62),硝酸のシグナル伝達および同化に関係する多くの遺伝子の転写制御領域に結合することが示された60).シロイヌナズナNLP7以外のホモログについても硝酸応答配列に結合することが明らかにされている61).興味深いことに,硝酸応答配列はNINが結合するシス配列と相同性を示し,実際に,NINは硝酸応答配列と結合することから,NINを含むNLPファミリーにおいてDNA結合ドメインであるRWP-RKは機能的に保存されていると考えられる63,64).ところが,根粒の形成は硝酸により抑制され,また,根粒菌が感染しても主要な硝酸応答遺伝子の発現は誘導されないことから,硝酸応答を正に制御するNLPと根粒の形成を正に制御するNINは転写制御領域への結合に対し拮抗的に作用しているのかもしれない64).
根粒の形成(根粒菌の感染)はさらなる根粒の形成を負に制御する65).これは植物の生存戦略からみると合理的であり,必要以上に根粒を形成することはそれだけ光合成産物を消費することになり宿主に負担が生じる.実際に,このフィードバックが破綻した変異体においては根粒菌の感染に依存して,とくに地上部の生育がいちじるしく阻害される66).同様に,十分な硝酸が得られる条件では根粒における窒素固定も必要ないことから,硝酸による根粒の形成の抑制も合理的である.根粒菌による根粒の形成の抑制にもまたNINが関与している.根粒菌が感染するとNINのもつ転写活性によりCLEペプチドをコードする遺伝子の発現が誘導される67).ここには2種類のCLEペプチドが関与するが,そのうちのひとつの発現は硝酸によっても誘導される68).生成したCLEペプチドは地上部へと輸送されLRR型受容体キナーゼHAR1のリガンドとしてはたらく69).HAR1の下流において地上部におけるサイトカイニンの合成が促進され,最終的に根における根粒の形成を抑制する70).
ミヤコグサHAR1はシロイヌナズナCLV1のオーソログであり,CLV1はCLEペプチドであるCLV3を認識することにより茎頂分裂組織の大きさを負に制御する.側根の形成における硝酸応答にはCLV1が関与しており71),HAR1による根粒の形成の制御も硝酸応答から進化したのかもしれない.実際に,さきに述べたようにHAR1は硝酸に依存して根粒の数を負に制御するほか,側根の数も負に制御する66).そもそも,硝酸応答は側根の形成の制御に重要な役割をはたしており72),たとえば,シロイヌナズナNLP7は側根の形成にも影響する62).また,シロイヌナズナにおいて,硝酸の欠乏により発現が誘導されるCEP1ペプチドに結合するLRR型受容体キナーゼCEPRは,側根の形成を負に制御する73).同様の関係はタルウマゴヤシにも存在するが,根粒の形成は正に制御されることから側根の形成と根粒の形成とが拮抗する関係になっている74,75).
4.側根の形成と根粒の形成
以上に述べたように,根粒の形成のマスターレギュレーターであるNINは硝酸応答のマスターレギュレーターであるNLPから進化し,硝酸応答は側根の形成と密接な関係のあることから,根粒の形成の進化においてNLPが制御する側根の形成プログラムを部分的に流用した可能性がある59).したがって,NINが転写を制御する下流の遺伝子のなかに側根の形成に関与する遺伝子があってもおかしくない.
NINはNF-YのサブユニットをコードするNF-YA1遺伝子およびNF-YB1遺伝子の発現を正に制御する56).NF-YはCCAATボックスに結合する転写因子であり,これらの遺伝子を過剰に発現させると皮層の細胞分裂が促進されるとともに異常な側根の形成が誘導される56).逆に,NF-YA1遺伝子の発現を抑制すると根粒の形成が阻害される56,76).マメ科においてはNF-YA1の属するクレードが重複しており77),NF-YA1のパラログは根粒においては発現が誘導されない78).マメ科植物のNF-YA1のシロイヌナズナにおけるオーソログは側根の形成に関与することから79),マメ科植物においては側根の形成に関与していたNF-Yの遺伝子重複により生じたパラログが,新規にNINの制御をうける転写制御領域を獲得し根粒の形成に関与するようになった可能性がある.実際に,アクチノリザル植物であるグラウカモクマオウ(Casuarina glauca)においては,NINの発現を抑制することにより根粒の形成が阻害されるものの,マメ科植物とは異なり根粒において発現するNF-YA1遺伝子およびNF-YB1遺伝子のホモログの発現には影響しない55).このことは,NINによるNF-Yの発現制御機構はマメ科植物の根粒に特徴的であることを示唆している.根粒の形態を比較すると,アクチノリザル植物の根粒は側根に類似しており(図5),根粒の形成の過程からみても側根の変形したものだと考えられている80).一方,マメ科植物の根粒は,内部の構成はアクチノリザル植物の根粒に類似しているものの,維管束の構成および形成の過程は大きく異なる51).したがって,アクチノリザル植物の根粒はマメ科植物の根粒とは相似であり,つまり,収斂進化の結果であると考えられる2).
おわりに
以上のことから,マメ科植物における根粒の形成は,進化の過程において複数の制御機構をモジュールとして獲得したことにより成立したと考えられる.その中心と考えられるのが転写因子NINであり,動物における形態形成の進化と同様に81),遺伝子重複などによる転写因子の新規の機能の獲得とともに,その上流および下流における転写制御領域の新規の獲得がモジュールの再結線をうながした結果,新規の器官である根粒の進化につながったと考えられる59).
筆者らは,結論として,根粒共生のプレディスポジョションにおいて重要なはたらきをした実体はNINだと想定している.すでに議論したとおり,Nod因子の受容に関与するLysM型受容体キナーゼは,その由来が菌根共生だとしても主要なFrankia属細菌がNod因子を合成しないことから除外される.また,アーバスキュラー菌根菌との共生にも必要な共通共生遺伝子は,陸上植物に広く保存されていることから対象外である.したがって,現時点で考えられるのはNINのみであり,マメ科植物あるいはアクチノリザル植物を問わずNINは根粒の形成において重要であることから,その下流において誘導される根粒の形成プログラムはマメ科植物とアクチノリザル植物とでは一部が異なると考えられるものの,共通共生経路がNINを含む転写制御機構を獲得したことが鍵であると結論される.NINの祖先型であるNLPは硝酸応答のマスターレギュレーターであり,さきに述べたように,側根の形成は硝酸によっても制御され,根におけるNINの下流の形態形成プログラムはNLPの形態形成プログラムと類似している可能性がある.マメ科植物においては側根の形態形成プログラムをNF-Yの獲得をつうじ新たに転用したことにより,新規の器官である根粒の形成につながったのではないだろうか.
窒素固定クレードの進化はおよそ1億年まえに起こったと考えられているが,根粒共生の進化は6千万年まえより以前にはさかのぼらない2).したがって,少なくとも4千万年のあいだ,窒素固定クレードの祖先種は根粒共生を進化させるポテンシャル(すなわち,プレディスポジョション)は獲得していたものの,実際に根粒共生を進化させることはなかったことになる.すなわち,プレディスポジションの役割は根粒共生ではない未知の形質において重要な役割をはたしていたことになる.しかしながら,さきにも述べた定量的な系統解析によると,根粒共生のプレディスポジションは窒素固定クレードにおいて17回失われていると予測されている3).このことは,窒素固定クレードの放散においてプレディスポジションは重要であったものの,そののち,なんらかの(環境)要因の変化により多くの窒素固定クレードではプレディスポジションに対する選択圧が低下し,結果として,根粒の形成の進化能を失ったのだろう.プレディスポジションの喪失と現生種の生理環境とを比較することにより,プレディスポジョションの本質にせまることが可能かもしれない82).
根粒共生は,ここで述べた根粒の形成のほか,根粒菌の宿主への感染と細胞への取り込み(細胞内共生),共生的な窒素固定,というおおまかには3つの段階にその過程を分けることができ,それぞれの段階において,遺伝子導入などによる検証をふまえた合成生物学的なアプローチにより主要穀物における再構成が可能であると考えている.根粒の形成については近年の分子遺伝学の成果から概略がつかめているが,根粒菌の感染および細胞内共生と共生的な窒素固定についてはさらなる知見が必要である.今後,イネ科植物における根粒形成の再構成と並行して,マメ科のモデル植物において根粒菌との共生の全貌を分子遺伝学的に明らかにすることが重要である.
文 献
- Soltis, D. E., Soltis, P. S., Morgan, D. R. et al.: Chloroplast gene sequence data suggest a single origin of the predisposition for symbiotic nitrogen fixation in angiosperms. Proc. Natl. Acad. Sci. USA, 92, 2647-2651 (1995)[PubMed]
- Doyle, J. J.: Phylogenetic perspectives on the origins of nodulation. Mol. Plant Microbe Interact., 24, 1289-1295 (2011)[PubMed]
- Werner, G. D. A., Cornwell, W. K., Sprent, J. I. et al.: A single evolutionary innovation drives the deep evolution of symbiotic N2-fixation in angiosperms. Nat. Commun., 5, 4087 (2014)[PubMed]
- Gutjahr, C. & Parniske, M.: Cell and developmental biology of arbuscular mycorrhiza symbiosis. Annu. Rev. Cell Dev. Biol., 29, 593-617 (2013)[PubMed]
- Remy, W., Taylor, T. N., Hass, H. et al.: Four hundred-million-year-old vesicular arbuscular mycorrhizae. Proc. Natl. Acad. Sci. USA, 91, 11841-11843 (1994)[PubMed]
- Chen, C., Gao, M., Liu, J. et al.: Fungal symbiosis in rice requires an ortholog of a legume common symbiosis gene encoding a Ca2+/calmodulin-dependent protein kinase. Plant Physiol., 145, 1619-1628 (2007)[PubMed]
- Gutjahr, C., Banba, M., Croset, V. et al.: Arbuscular mycorrhiza: specific signaling in rice transcends the common symbiosis signaling pathway. Plant Cell, 20, 2989-3005 (2008)[PubMed]
- Chen, C., Ane, J. M. & Zhu, H.: OsIPD3, an ortholog of the Medicago truncatula DMI3 interacting protein IPD3, is required for mycorrhizal symbiosis in rice. New Phytol., 180, 311-315 (2008)[PubMed]
- Chen, C., Fan, C., Gao, M. et al.: Antiquity and function of CASTOR and POLLUX, the twin ion channel-encoding genes key to the evolution of root symbioses in plants. Plant Physiol., 149, 306-317 (2009)[PubMed]
- Delaux, P. M., Varala, K., Edger, P. P. et al.: Comparative phylogenomics uncovers the impact of symbiotic associations on host genome evolution. PLoS Genet., 10, e1004487 (2014)[PubMed]
- Godfroy, O., Debelle, F., Timmers, T. et al.: A rice calcium- and calmodulin-dependent protein kinase restores nodulation to a legume mutant. Mol. Plant Microbe Interact., 19, 495-501 (2006)[PubMed]
- Markmann, K., Giczey, G. & Parniske, M.: Functional adaptation of a plant receptor-kinase paved the way for the evolution of intracellular root symbioses with bacteria. PLoS Biol., 6, e68 (2008)[PubMed]
- Banba, M., Gutjahr, C., Miyao, A. et al.: Divergence of evolutionary ways among common sym genes: CASTOR and CCaMK show functional conservation between two symbiosis systems and constitute the root of a common symbiosis pathway. Plant Cell Physiol., 49, 1659-1671 (2008)[PubMed]
- Yano, K., Yoshida, S., Muller, J. et al.: CYCLOPS, a mediator of symbiotic intracellular accommodation. Proc. Natl. Acad. Sci. USA, 105, 20540-20545 (2008)[PubMed]
- Gherbi, H., Markmann, K., Svistoonoff, S. et al.: SymRK defines a common genetic basis for plant root endosymbioses with arbuscular mycorrhiza fungi, rhizobia, and Frankia bacteria. Proc. Natl. Acad. Sci. USA, 105, 4928-4932 (2008)[PubMed]
- Svistoonoff, S., Benabdoun, F. M., Nambiar-Veetil, M. et al.: The independent acquisition of plant root nitrogen-fixing symbiosis in Fabids recruited the same genetic pathway for nodule organogenesis. PLoS One, 8, e64515 (2013)[PubMed]
- Kistner, C. & Parniske, M.: Evolution of signal transduction in intracellular symbiosis. Trends Plant Sci., 7, 511-518 (2002)[PubMed]
- Kistner, C., Winzer, T., Pitzschke, A. et al.: Seven Lotus japonicus genes required for transcriptional reprogramming of the root during fungal and bacterial symbiosis. Plant Cell, 17, 2217-2229 (2005)[PubMed]
- Genre, A., Chabaud, M., Timmers, T. et al.: Arbuscular mycorrhizal fungi elicit a novel intracellular apparatus in Medicago truncatula root epidermal cells before infection. Plant Cell, 17, 3489-3499 (2005)[PubMed]
- Genre, A., Chabaud, M., Faccio, A. et al.: Prepenetration apparatus assembly precedes and predicts the colonization patterns of arbuscular mycorrhizal fungi within the root cortex of both Medicago truncatula and Daucus carota. Plant Cell, 20, 1407-1420 (2008)[PubMed]
- Takeda, N., Maekawa, T. & Hayashi, M.: Nuclear-localized and deregulated calcium- and calmodulin-dependent protein kinase activates rhizobial and mycorrhizal responses in Lotus japonicus. Plant Cell, 24, 810-822 (2012)[PubMed]
- D'Haeze, W. & Holsters, M.: Nod factor structures, responses, and perception during initiation of nodule development. Glycobiology, 12, 79R-105R (2002)[PubMed]
- Radutoiu, S., Madsen, L. H., Madsen, E. B. et al.: Plant recognition of symbiotic bacteria requires two LysM receptor-like kinases. Nature, 425, 585-592 (2003)[PubMed]
- Madsen, E. B., Madsen, L. H., Radutoiu, S. et al.: A receptor kinase gene of the LysM type is involved in legume perception of rhizobial signals. Nature, 425, 637-640 (2003)[PubMed]
- Arrighi, J. F., Barre, A., Ben Amor, B. et al.: The Medicago truncatula lysin motif-receptor-like kinase gene family includes NFP and new nodule-expressed genes. Plant Physiol., 142, 265-279 (2006)[PubMed]
- Smit, P., Limpens, E., Geurts, R. et al.: Medicago LYK3, an entry receptor in rhizobial nodulation factor signaling. Plant Physiol., 145, 183-191 (2007)[PubMed]
- Madsen, E. B., Antolin-Llovera, M., Grossmann, C. et al.: Autophosphorylation is essential for the in vivo function of the Lotus japonicus Nod factor receptor 1 and receptor-mediated signalling in cooperation with Nod factor receptor 5. Plant J., 65, 404-417 (2011)[PubMed]
- Pietraszewska-Bogiel, A., Lefebvre, B., Koini, M. A. et al.: Interaction of Medicago truncatula lysin motif receptor-like kinases, NFP and LYK3, produced in Nicotiana benthamiana induces defence-like responses. PLoS One, 8, e65055 (2013)[PubMed]
- Moling, S., Pietraszewska-Bogiel, A., Postma, M. et al.: Nod factor receptors form heteromeric complexes and are essential for intracellular infection in Medicago nodules. Plant Cell, 26, 4188-4199 (2014)[PubMed]
- Radutoiu, S., Madsen, L. H., Madsen, E. B. et al.: LysM domains mediate lipochitin-oligosaccharide recognition and Nfr genes extend the symbiotic host range. EMBO J., 26, 3923-3935 (2007)[PubMed]
- Bensmihen, S., de Billy, F. & Gough, C.: Contribution of NFP LysM domains to the recognition of Nod factors during the Medicago truncatula/Sinorhizobium meliloti symbiosis. PLoS One, 6, e26114 (2011)[PubMed]
- Broghammer, A., Krusell, L., Blaise, M. et al.: Legume receptors perceive the rhizobial lipochitin oligosaccharide signal molecules by direct binding. Proc. Natl. Acad. Sci. USA, 109, 13859-13864 (2012)[PubMed]
- Sorensen, K. K., Simonsen, J. B., Maolanon, N. N. et al.: Chemically synthesized 58-mer LysM domain binds lipochitin oligosaccharide. Chembiochem, 15, 2097-2105 (2014)[PubMed]
- Maillet, F., Poinsot, V., Andre, O. et al.: Fungal lipochitooligosaccharide symbiotic signals in arbuscular mycorrhiza. Nature, 469, 58-63 (2011)[PubMed]
- Olah, B., Briere, C., Becard, G. et al.: Nod factors and a diffusible factor from arbuscular mycorrhizal fungi stimulate lateral root formation in Medicago truncatula via the DMI1/DMI2 signalling pathway. Plant J., 44, 195-207 (2005)[PubMed]
- Gutjahr, C., Casieri, L. & Paszkowski, U.: Glomus intraradices induces changes in root system architecture of rice independently of common symbiosis signaling. New Phytol., 182, 829-837 (2009)[PubMed]
- Mukherjee, A. & Ane, J. M.: Germinating spore exudates from arbuscular mycorrhizal fungi: molecular and developmental responses in plants and their regulation by ethylene. Mol. Plant Microbe Interact., 24, 260-270 (2011)[PubMed]
- Amor, B. B., Shaw, S. L., Oldroyd, G. E. et al.: The NFP locus of Medicago truncatula controls an early step of Nod factor signal transduction upstream of a rapid calcium flux and root hair deformation. Plant J., 34, 495-506 (2003)[PubMed]
- Zhang, X., Dong, W., Sun, J. et al.: The receptor kinase CERK1 has dual functions in symbiosis and immunity signalling. Plant J., 81, 258-267 (2015)[PubMed]
- Young, N. D., Debelle, F., Oldroyd, G. E. et al.: The Medicago genome provides insight into the evolution of rhizobial symbioses. Nature, 480, 520-524 (2011)[PubMed]
- Cannon, S. B., Ilut, D., Farmer, A. D. et al.: Polyploidy did not predate the evolution of nodulation in all legumes. PLoS One, 5, e11630 (2010)[PubMed]
- Op den Camp, R., Streng, A., De Mita, S. et al.: LysM-type mycorrhizal receptor recruited for rhizobium symbiosis in nonlegume Parasponia. Science, 331, 909-912 (2011)[PubMed]
- Op den Camp, R. H., Polone, E., Fedorova, E. et al.: Nonlegume Parasponia andersonii deploys a broad rhizobium host range strategy resulting in largely variable symbiotic effectiveness. Mol. Plant Microbe Interact., 25, 954-963 (2012)[PubMed]
- Liang, Y., Cao, Y., Tanaka, K. et al.: Nonlegumes respond to rhizobial Nod factors by suppressing the innate immune response. Science, 341, 1384-1387 (2013)[PubMed]
- Genre, A., Chabaud, M., Balzergue, C. et al.: Short-chain chitin oligomers from arbuscular mycorrhizal fungi trigger nuclear Ca2+ spiking in Medicago truncatula roots and their production is enhanced by strigolactone. New Phytol., 198, 190-202 (2013)[PubMed]
- Sun, J., Miller, J. B., Granqvist, E. et al.: Activation of symbiosis signaling by arbuscular mycorrhizal fungi in legumes and rice. Plant Cell, 27, 823-838 (2015)[PubMed]
- Miyata, K., Kozaki, T. & Kouzai, Y. et al.: The bifunctional plant receptor, OsCERK1, regulates both chitin-triggered immunity and arbuscular mycorrhizal symbiosis in rice. Plant Cell Physiol., 55, 1864-1872 (2014)[PubMed]
- Sanchez-Vallet, A., Mesters, J. R. & Thomma, B. P.: The battle for chitin recognition in plant-microbe interactions. FEMS Microbiol. Rev., 39, 171-183 (2014)[PubMed]
- De Mita, S., Streng, A., Bisseling, T. et al.: Evolution of a symbiotic receptor through gene duplications in the legume-rhizobium mutualism. New Phytol., 201, 961-972 (2014)[PubMed]
- Normand, P., Lapierre, P., Tisa, L. S. et al.: Genome characteristics of facultatively symbiotic Frankia sp. strains reflect host range and host plant biogeography. Genome Res., 17, 7-15 (2007)[PubMed]
- Svistoonoff, S., Hocher, V. & Gherbi, H.: Actinorhizal root nodule symbioses: what is signalling telling on the origins of nodulation? Curr. Opin. Plant Biol., 20, 11-18 (2014)[PubMed]
- Schauser, L., Roussis, A., Stiller, J. et al.: A plant regulator controlling development of symbiotic root nodules. Nature, 402, 191-195 (1999)[PubMed]
- Borisov, A. Y., Madsen, L. H., Tsyganov, V. E. et al.: The Sym35 gene required for root nodule development in pea is an ortholog of Nin from Lotus japonicus. Plant Physiol., 131, 1009-1017 (2003)[PubMed]
- Marsh, J. F., Rakocevic, A., Mitra, R. M. et al.: Medicago truncatula NIN is essential for rhizobial-independent nodule organogenesis induced by autoactive calcium/calmodulin-dependent protein kinase. Plant Physiol., 144, 324-335 (2007)[PubMed]
- Clavijo, F., Diedhiou, I., Vaissayre, V. et al.: The Casuarina NIN gene is transcriptionally activated throughout Frankia root infection as well as in response to bacterial diffusible signals. New Phytol., 208, 887-903 (2015)[PubMed]
- Soyano, T., Kouchi, H., Hirota, A. et al.: Nodule inception directly targets NF-Y subunit genes to regulate essential processes of root nodule development in Lotus japonicus. PLoS Genet, 9, e1003352 (2013)[PubMed]
- Schauser, L., Wieloch, W. & Stougaard, J.: Evolution of NIN-like proteins in Arabidopsis, rice, and Lotus japonicus. J. Mol. Evol., 60, 229-237 (2005)[PubMed]
- Chardin, C., Girin, T., Roudier, F. et al.: The plant RWP-RK transcription factors: key regulators of nitrogen responses and of gametophyte development. J. Exp. Bot., 65, 5577-5587 (2014)[PubMed]
- Soyano, T. & Hayashi, M.: Transcriptional networks leading to symbiotic nodule organogenesis. Curr. Opin. Plant Biol., 20, 146-154 (2014)[PubMed]
- Marchive, C., Roudier, F., Castaings, L. et al.: Nuclear retention of the transcription factor NLP7 orchestrates the early response to nitrate in plants. Nat. Commun., 4, 1713 (2013)[PubMed]
- Konishi, M. & Yanagisawa, S.: Arabidopsis NIN-like transcription factors have a central role in nitrate signalling. Nat. Commun., 4, 1617 (2013)[PubMed]
- Castaings, L., Camargo, A., Pocholle, D. et al.: The nodule inception-like protein 7 modulates nitrate sensing and metabolism in Arabidopsis. Plant J., 57, 426-435 (2009)[PubMed]
- Suzuki, W., Konishi, M. & Yanagisawa, S.: The evolutionary events necessary for the emergence of symbiotic nitrogen fixation in legumes may involve a loss of nitrate responsiveness of the NIN transcription factor. Plant Signal. Behav., 8, e25975 (2013)[PubMed]
- Soyano, T., Shimoda, Y. & Hayashi, M.: NODULE INCEPTION antagonistically regulates gene expression with nitrate in Lotus japonicus. Plant Cell Physiol. 56: 368-376 (2015)[PubMed]
- Oka-Kira, E. & Kawaguchi, M.: Long-distance signaling to control root nodule number. Curr. Opin. Plant Biol., 9, 496-502 (2006)[PubMed]
- Wopereis, J., Pajuelo, E., Dazzo, F. B. et al.: Short root mutant of Lotus japonicus with a dramatically altered symbiotic phenotype. Plant J., 23, 97-114 (2000)[PubMed]
- Soyano, T., Hirakawa, H., Sato, S. et al.: NODULE INCEPTION creates a long-distance negative feedback loop involved in homeostatic regulation of nodule organ production. Proc. Natl. Acad. Sci. USA, 111, 14607-14612 (2014)[PubMed]
- Okamoto, S., Ohnishi, E., Sato, S. et al.: Nod factor/nitrate-induced CLE genes that drive HAR1-mediated systemic regulation of nodulation. Plant Cell Physiol., 50, 67-77 (2009)[PubMed]
- Okamoto, S., Shinohara, H., Mori, T. et al.: Root-derived CLE glycopeptides control nodulation by direct binding to HAR1 receptor kinase. Nat. Commun., 4, 2191 (2013)[PubMed]
- Sasaki, T., Suzaki, T., Soyano, T. et al.: Shoot-derived cytokinins systemically regulate root nodulation. Nat. Commun., 5, 4983 (2014)[PubMed]
- Araya, T., Miyamoto, M., Wibowo, J. et al.: CLE-CLAVATA1 peptide-receptor signaling module regulates the expansion of plant root systems in a nitrogen-dependent manner. Proc. Natl. Acad. Sci. USA, 111, 2029-2034 (2014)[PubMed]
- Forde, B. G.: Nitrogen signalling pathways shaping root system architecture: an update. Curr. Opin. Plant Biol., 21, 30-36 (2014)[PubMed]
- Tabata, R., Sumida, K., Yoshii, T. et al.: Perception of root-derived peptides by shoot LRR-RKs mediates systemic N-demand signaling. Science, 346, 343-346 (2014)[PubMed]
- Imin, N., Mohd-Radzman, N. A., Ogilvie, H. A. et al.: The peptide-encoding CEP1 gene modulates lateral root and nodule numbers in Medicago truncatula. J. Exp. Bot., 64, 5395-5409 (2013)[PubMed]
- Huault, E., Laffont, C., Wen, J. et al.: Local and systemic regulation of plant root system architecture and symbiotic nodulation by a receptor-like kinase. PLoS Genet., 10, e1004891 (2014)[PubMed]
- Laporte, P., Lepage, A., Fournier, J. et al.: The CCAAT box-binding transcription factor NF-YA1 controls rhizobial infection. J. Exp. Bot., 65, 481-494 (2014)[PubMed]
- Laloum, T., De Mita, S., Gamas, P. et al.: CCAAT-box binding transcription factors in plants: Y so many? Trends Plant Sci., 18, 157-166 (2013)[PubMed]
- Asamizu, E., Shimoda, Y., Kouchi, H. et al.: A positive regulatory role for LjERF1 in the nodulation process is revealed by systematic analysis of nodule-associated transcription factors of Lotus japonicus. Plant Physiol., 147, 2030-2040 (2008)[PubMed]
- Sorin, C., Declerck, M., Christ, A. et al.: A miR169 isoform regulates specific NF-YA targets and root architecture in Arabidopsis. New Phytol., 202, 1197-1211 (2014)[PubMed]
- Pawlowski, K. & Demchenko, K. N.: The diversity of actinorhizal symbiosis. Protoplasma, 249, 967-979 (2012)[PubMed]
- Monteiro, A. & Podlaha, O.: Wings, horns, and butterfly eyespots: how do complex traits evolve? PLoS Biol., 7, e37 (2009)[PubMed]
- Werner, G. D., Cornwell, W. K., Cornelissen, J. H. et al.: Evolutionary signals of symbiotic persistence in the legume-rhizobia mutualism. Proc. Natl. Acad. Sci. USA, 112, 10262-10269 (2015)[PubMed]
著者プロフィール
略歴:1996年 東京大学大学院理学系研究科博士課程 修了,1997年 大阪大学大学院工学研究科 助手,2006年 ドイツMunich大学 教授,2008年 農業生物資源研究所植物科学研究領域 ユニット長を経て,2014年より理化学研究所環境資源科学研究センター チームリーダー.
研究テーマ:植物の形態形成.
関心事:生物の多様性.
研究室URL:http://www.riken.jp/research/labs/csrs/plant_symb/
© 2015 林 誠 Licensed under CC 表示 2.1 日本