真核生物における染色体DNAの複製開始の機構
2016/07/05
荒木 弘之
(国立遺伝学研究所 微生物遺伝研究部門)
email:荒木弘之
領域融合レビュー, 5, e006 (2016) DOI: 10.7875/leading.author.5.e006
Hiroyuki Araki: Molecular mechanism of the initiation of chromosomal DNA replication in eukaryotes.

真核生物における染色体DNAの複製は特定の領域から開始する.この複製開始点にはOrcが結合し,DNAヘリカーゼの触媒コアであるMcm2-7へテロ六量体が主としてG1期にロードされる.複製開始の過程が細胞周期により制御されることによりDNA複製と細胞分裂は共役するが,これには細胞周期の制御の鍵となるサイクリン依存性キナーゼおよびDbf4依存性キナーゼによる複製タンパク質のリン酸化が重要な役割を担う.すなわち,これらリン酸化によりCdc45とGINS複合体とが結合したCMG複合体が複製開始点に形成され,複製において活性をもつDNAヘリカーゼとしてはたらくようになることが複製開始の制御において要となる.そして,DNAポリメラーゼがリクルートされDNA複製が開始する.現時点において詳細は不明だが,Mcm2-7へテロ六量体およびCMG複合体の構造,そして,in vitroにおける再構築により,CMG複合体の形成の機構を類推できるようになってきており,複製開始の機構について全容の解明が近づいている.
生命現象を分子レベルの機構として知りたいとき,どのように研究を進めるだろうか? クラッシックには,1)反応に関与するタンパク質をみつけ,2)そのタンパク質を除いたり変異をくわえたりすることによる変化から反応機構を推察しモデルを構築する.さらに,3)単離したタンパク質を組み合わせることにより反応をin vitroにおいて再構築することができれば,モデルの証明と同時にさらに詳細な機構を知ることができる.真核生物におけるDNA複製の機構の研究は,ウイルスを用いたin vitro DNA複製系が構築され,3)の段階にまで到達した.しかし,ウイルスは宿主細胞の複製系そのものではなくウイルス独自の機構をもつため,細胞のなかで起こる複製反応を知るには不十分であった.また,アフリカツメガエルの卵抽出液を用いた複製系も構築され解析も進んだが,2)の段階でとまっている.一方,シンプルな単細胞の真核生物である酵母をモデルとした研究においては,遺伝学的な手法による1)や2)の段階の研究が進み,現在,3)の段階に進みつつある.
一方で,タンパク質の構造解析の技術の進展がめざましい.X線による結晶解析やNMRによる溶液における構造解析にくわえ,近年,電子顕微鏡を用いた単粒子解析の技術が急速に進み巨大なタンパク質の構造が原子レベルで決定されるようになってきた.これらの構造と,さきに述べた解析を組み合わせることにより,真核生物における分子レベルでの複製機構の理解は,今世紀にはいって急激に進んでいる.ここでは,最近の研究を中心に,真核生物における染色体DNAの複製開始の機構を解説する.
染色体DNAの複製は染色体に散在する特定の部位である複製開始点あるいは複製開始領域から開始する.複製開始点の数は酵母で約1000,ヒトでは数万になる.その部位を規定するのは6つのサブユニットからなるOrcである.OrcはATPと結合し加水分解するATPaseであり,2本鎖DNAと結合する.酵母からヒトまでよく保存されており,酵母やショウジョウバエのOrcは電子顕微鏡による単粒子解析やX線結晶構造解析からその構造が明らかにされている1-3).このOrcの性質が複製開始点を決めるうえで重要な役割を担う.出芽酵母(Saccharomyces cerevisiae)においては特定の11 bpの配列であるACSと結合するが,分裂酵母(Schizosaccharomyces pombe)においてはOrc4がATフックとよばれる構造をもつためATを多く含む領域と結合する4,5).しかし,ヒトやショウジョウバエのOrcは特定の塩基配列を認識しない.したがって,酵母ではDNA配列により複製開始点が規定されるが,多細胞生物ではDNA配列よりむしろクロマチンの状態などによりOrcの結合部位が決まると考えられている6).実際,Orcのもっとも大きなサブユニットであるOrc1はヒストンとの結合部位をもち,その部位を除くと染色体におけるOrcの結合様式が変化する7).また,Orcと染色体DNAとの結合は,出芽酵母においては細胞周期をつうじて起こるが,分裂酵母においては細胞周期により結合の状態が変わるとの報告もある.さらに,哺乳類においてOrc1はG1期から結合しM期には分解される5).
DNA鎖を合成するDNAポリメラーゼは2本鎖DNAに対してDNA鎖を合成することはできない.DNA鎖の合成をはじめるには,鋳型となる1本鎖DNAおよびプライマーが必要である.そのため,複製開始点において2本鎖DNAが1本鎖DNAにほどかれないかぎりDNAの合成は開始されない.大腸菌においては複製開始点に複数のDnaAが結合するとその近傍の2本鎖DNAがほどかれるが,真核生物においてはOrcの結合により複製開始点において2本鎖DNAがほどかれるという報告はない.
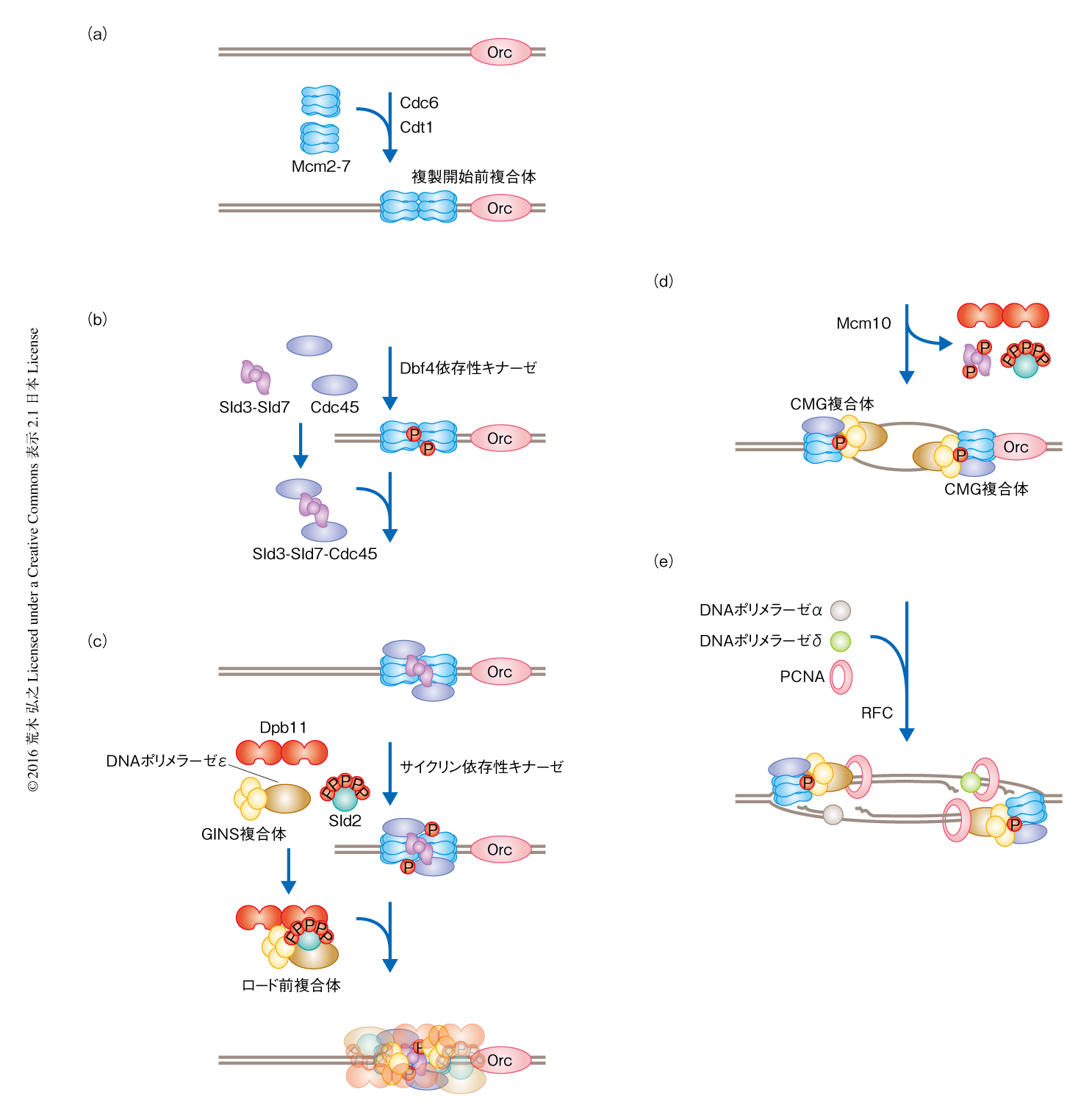
真核生物においてはサイクリン依存性キナーゼの活性の低いM期の後期からG1期にかけて,2本鎖DNAを1本鎖DNAにほどくDNAヘリカーゼの触媒コアとなるMcm2-7へテロ六量体がOrcの結合した複製開始点にロードされる(図1a).この染色体DNAにおけるOrcおよびMcm2-7へテロ六量体からなる構造を複製開始前複合体(pre-replicative complexあるいはpre-replication complex:pre-RC)とよぶ.複製開始前複合体の形成には,OrcおよびMcm2-7へテロ六量体にくわえCdc6およびCdt1が必要である.出芽酵母においてCdt1はMcm2-7へテロ六量体と複合体を形成しており,Cdc6はOrcとともにはたらく.Orc,Mcm2-7へテロ六量体,Cdc6はATPase活性をもち,ATP加水分解のエネルギーを用いてMcm2-7へテロ六量体を複製開始点にロードする.
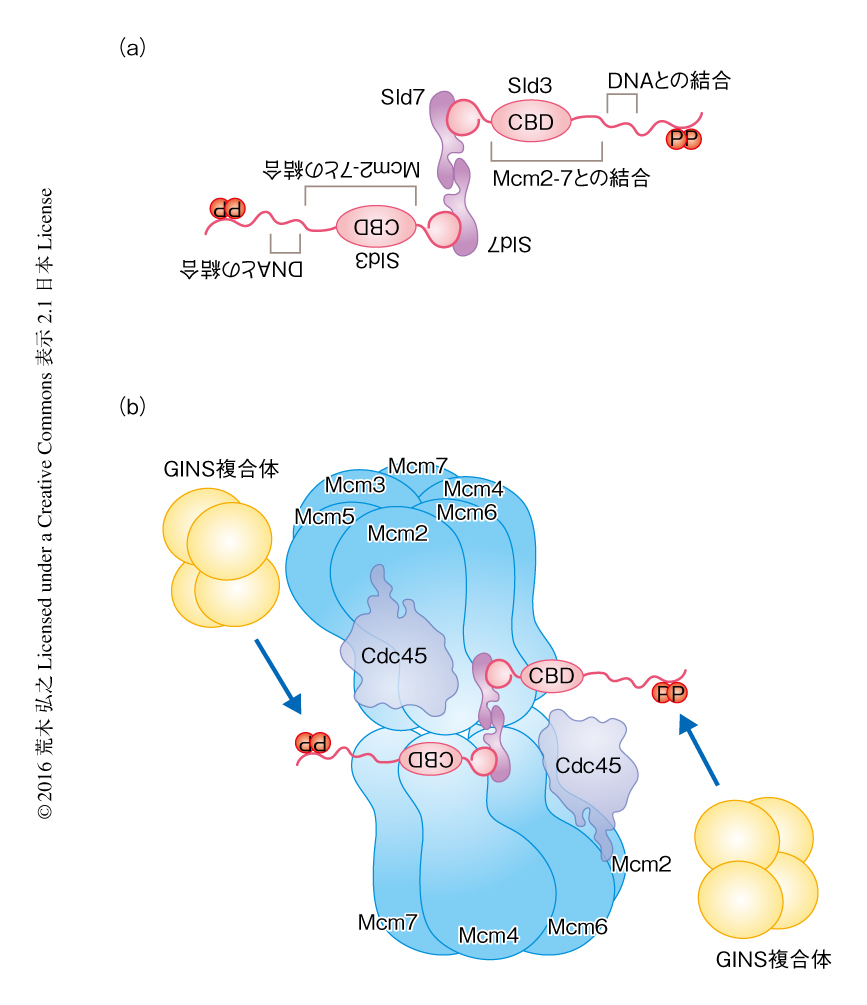
Mcm2-7へテロ六量体のそれぞれのサブユニットは共通する特徴をもつ.すなわち,N末端側にOBフォールドおよびジンクフィンガードメイン,C末端側にAAA+ファミリーに属するATPaseドメインが位置する.複製開始点には2分子のMcm2-7ヘテロ6量体のN末端側がむかい合うようにロードされるが,電子顕微鏡による単粒子解析などからおのおののサブユニットの配置が報告されている2)(図2).そして,Mcm2とMcm5とのあいだに隙間が生じ,この部分を用いて2本鎖DNAにロードされると考えられている.また,Mcm2-Mcm6の側部にも隙間が観察される.さらに,相対するMcm5とMcm7のN末端およびジンクフィンガードメインの相互作用により結合する8).
Mcm2-7へテロ六量体が複製開始点へロードされる機構は,出芽酵母において精製タンパク質を用いた再構成系により解析されている.残念ながら,一対のMcm2-7へテロ六量体がどのように複製開始点へロードされるかはまだ完全にはわかっていない.ロードされたMcm2-7へテロ六量体は必ず対になっているため,一対のMcm2-7へテロ六量体がいちどにロードされるのではないかとの説もあったが,1分子観察などによると,まず1分子のMcm2-7へテロ六量体がロードされ,すぐにつぎの1分子のMcm2-7へテロ六量体がロードされており9),Mcm2-7へテロ六量体のロードが正常に起こっているかどうかの検知機構もあるようである10,11).いずれにせよ,厳密な機構,とくに,2分子目のMcm2-7へテロ六量体のロードについては不明な点が多く,今後の研究を待たなければならない.
Mcm2-7へテロ六量体はDNAヘリカーゼ活性を担うが,Mcm2-7へテロ六量体だけでは活性を示さない(生物種によっては,いくつかのサブユニットからなる複合体が弱い活性を示すものもある).DNAヘリカーゼの活性化にはG1期の後期からS期において活性化されるサイクリン依存性キナーゼおよびDbf4依存性キナーゼによるリン酸化,および,それにともなう複数のタンパク質の複製開始点への集合が必要であることが出芽酵母を用いた解析からわかってきた.ここでは,Mcm2-7へテロ六量体のもつDNAヘリカーゼ活性の活性化について,出芽酵母を中心に説明する.
Dbf4依存性キナーゼはCdc7を触媒サブユニットとし,制御サブユニットであるDbf4と複合体を形成する.G1期の後期にDBF4遺伝子の転写が活性化され,Dbf4のタンパク質量が増加することによりDbf4依存性キナーゼは活性化される.活性化されたDbf4依存性キナーゼはMcm2,Mcm4,Mcm6のN末端側の構造をとらない領域にある複数のSerおよびThrをリン酸化する.とくに,Mcm6のDbf4依存性キナーゼによるリン酸化はSld3-Sld7複合体とCdc45との複合体の複製開始点との結合を促進する12)(図1b).
Sld3-Sld7複合体は2分子のSld3がそのN末端領域においてSld7二量体により結合された強固な構造をとる13)(図2a).Sld3の中央にはCdc45との結合領域があり,1分子のCdc45が1分子のSld3と結合するため14),Sld3-Sld7複合体は2分子のCdc45と結合する.細胞においてSld3-Sld7複合体とCdc45は互いに依存して複製開始点と結合するが15,16),in vitroにおいて,Sld3-Sld7複合体はCdc45がなくても複製開始点と結合できるが,Cdc45の複製開始点との結合にはSld3が必要である12).このように,Dbf4依存性キナーゼによるMcm6のリン酸化によりSld3-Sld7複合体とCdc45との複合体がMcm2-7へテロ六量体と結合する.筆者らは,Sld3-Sld7複合体の構造からその配置を推測しており(図2b),この配置は,のちに述べる活性化型のDNAヘリカーゼの形成にも都合がよいものとなっている.
サイクリン依存性キナーゼは出芽酵母においてはSld2およびSld3をリン酸化することにより複製開始を促進する17).Sld2にはサイクリン依存性キナーゼによりリン酸化されうる11箇所の部位が存在する.このうち,Thr84がサイクリン依存性キナーゼによりリン酸化されるとDpb11と結合する.Dpb11はC末端側およびN末端側にタンデムにならんだBRCT領域をもち,このタンデムにならんだBRCT領域はリン酸化されたペプチドと結合することが知られている.リン酸化されたSld2はDpb11のC末端側にあるタンデムにならんだBRCT領域と結合する.一方,Dpb11のN末端側にあるタンデムにならんだBRCT領域はリン酸化されたSld3と結合する.Sld3もサイクリン依存性キナーゼによりリン酸化されうる12箇所の部位をもつが,C末端側にあるThr600およびSer622のリン酸化がDpb11との結合に関与する.どちらか一方がリン酸化されれば結合は可能であるが,両方ともリン酸化されると強く結合する.
サイクリン依存性キナーゼによりリン酸化されたSld2およびSld3がDpb11と結合することにより,どのようして複製開始が促進されるのであろうか.その説明のまえに,活性型のDNAヘリカーゼについて説明したほうが理解しやすいだろう.Mcm2-7へテロ六量体はDNAヘリカーゼとしての活性をもつが,さきに述べたように,Mcm2-7へテロ六量体だけでは活性を示さない.細胞においては,Mcm2-7へテロ六量体はCdc45およびGINS複合体とCMG複合体とよばれる強固な複合体を形成し,このCMG複合体が複製において活性をもつDNAヘリカーゼとしてはたらく18).さきに述べたように,Cdc45はSld3-Sld7複合体と結合し,Mcm2-7へテロ六量体に相互依存的にリクルートされる.GINS複合体はSld5,Psf1,Psf2,Psf3の4つのサブユニットからなる複合体で,筆者らにより,末尾の数字の日本語読みでgo,ichi,nii,sanと名づけられたものである.Sld3はCdc45のリクルートおよびGINS複合体の複製開始点へのリクルートに必要である.そして,筆者らにより,Sld2のサイクリン依存性キナーゼによるリン酸化もGINS複合体のリクルートに関与していることが明らかにされ,この機構についても解析した.その結果,リン酸化されたSld2がDpb11と結合すると,DNAポリメラーゼεおよびGINS複合体がこの複合体にさらにくわわることが示された.この複合体をロード前複合体(pre-loading complex:pre-LC)と名づけ,ロード前複合体に含まれるDpb11が複製開始点においてリン酸化されたSld3と結合することにより,GINS複合体は複製開始点にリクルートされるものと考えている19)(図1c).DNAポリメラーゼεは細胞周期をつうじGINS複合体と安定な複合体を形成しており,DNAポリメラーゼεおよびGINS複合体はDpb11と弱く相互作用する.これらの結合がSld2により安定化されているのであろう.
哺乳類においてはDpb11と対応するBRCT領域を9つもつTopBP1が知られていたが,TopBP1の結合タンパク質としてSld3に対応するTreslin/Ticrrがみつかった20,21).アフリカツメガエルの卵抽出液を用いた解析により,TopBP1およびTreslinは独立にクロマチンと結合するが,TopBP1とTreslinとの結合はサイクリン依存性キナーゼによるTreslinのリン酸化により起こり,複製開始に必須であることがわかった.さらに,TreslinにはMTBPが結合し,このMTBPも複製開始に必須であることが示されている22).これがSld3と複合体を形成するSld7と同じはたらきをしているかどうかはわからない.一方,哺乳類においてSld2と弱い相同性を示すタンパク質としてRecQL4が知られている.RecQL4はC末端側にヘリカーゼドメインをもち,このドメインの欠損は早老症のひとつであるRothmund-Thomson症候群の発症の原因となる.ヘリカーゼドメインは細胞の増殖には必須ではないが,Sld2と相同性を示すN末端側の領域は必須である17).アフリカツメガエルの卵抽出液を用いた解析から,RecQL4は複製開始の後半においてはたらいているようで,DNAポリメラーゼαのリクルートに必要との報告もあり,今後の解析が必要である.
複製開始点に結合したMcm2-7へテロ六量体,Cdc45,GINS複合体は強固なCMG複合体を形成する18)(図1c).しかし,CMG複合体の形成はすぐには複製開始にはつながらない.それは,Mcm10を欠くとCMG複合体様の強固な複合体は形成されるが,複製開始は起こらないからである.Mcm10は一対のCMG複合体が両方向にむけ2本鎖DNAをほどくようにするはたらきがあると考えられているが(図1d),分子機能はわかっていない23).Mcm10の機能は複製開始における残されたミステリーである.
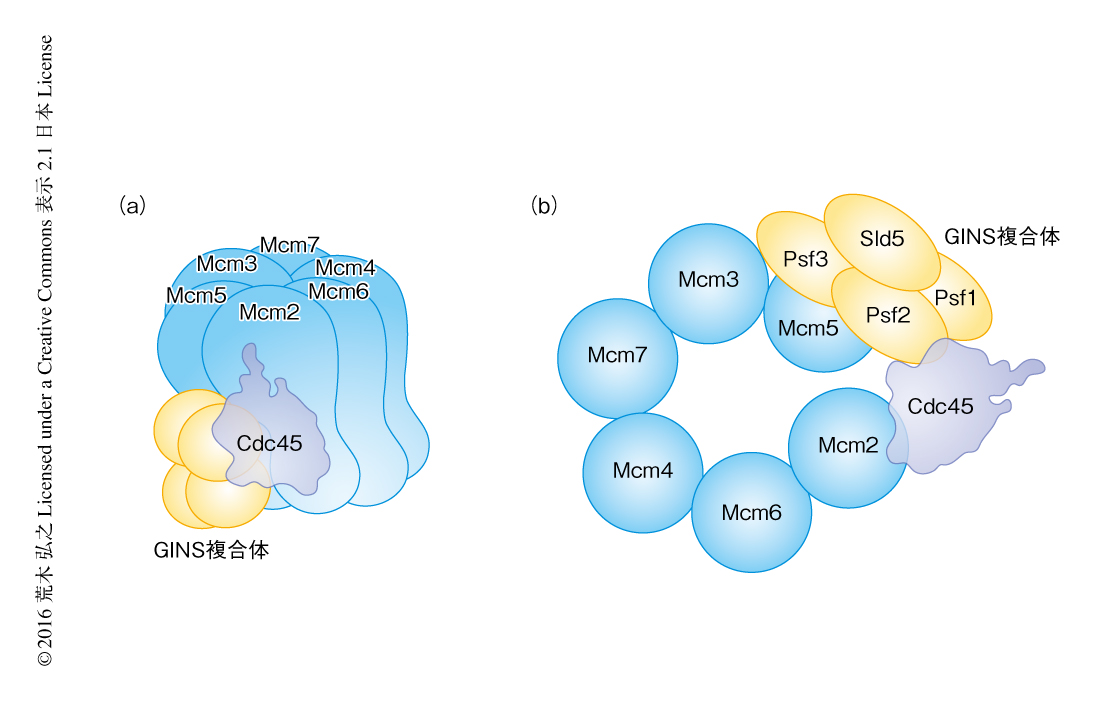
強固なCMG複合体ができる過程はまだわからないが,Mcm2-7へテロ六量体およびCMG複合体の構造は電子顕微鏡による単粒子解析から明らかにされている8,24,25).GINS複合体およびCdc45はMcm2-7へテロ六量体のMcm2とMcm5とのあいだの隙間をうめるよう位置する(図3).さらに,N末端側がむかい合うよう形成された一対のMcm2-7へテロ六量体の位置関係からは,Mcm2とMcm5とのあいだの隙間は反対の方向をむいている(図2b).Mcm2-7へテロ六量体をDNAにロードさせ電子顕微鏡により観察すると,2本鎖DNAをとおすかたちにみえる.一方で,CMG複合体におけるDNAヘリカーゼ活性の解析から,CMG複合体は1本鎖DNAのうえを3’側から5’側の方向に動いていることがわかった(図4).すなわち,CMG複合体はリーディング鎖の鋳型DNAのうえを複製の方向に動いていることになる.これらのことから,Mcm2-7へテロ六量体はまず2本鎖DNAをリングにとおし,活性化された時点あるいはCMG複合体が形成された時点で1本鎖DNAをリングにとおす.このとき,Mcm2とMcm5とのあいだの隙間の位置が同一の方向をむかないのは,複製開始点にロードされた一対のMcm2-7へテロ六量体あるいはCMG複合体が2本鎖DNAから1本鎖DNAへと乗り換えるのには都合がよいからかもしれない.
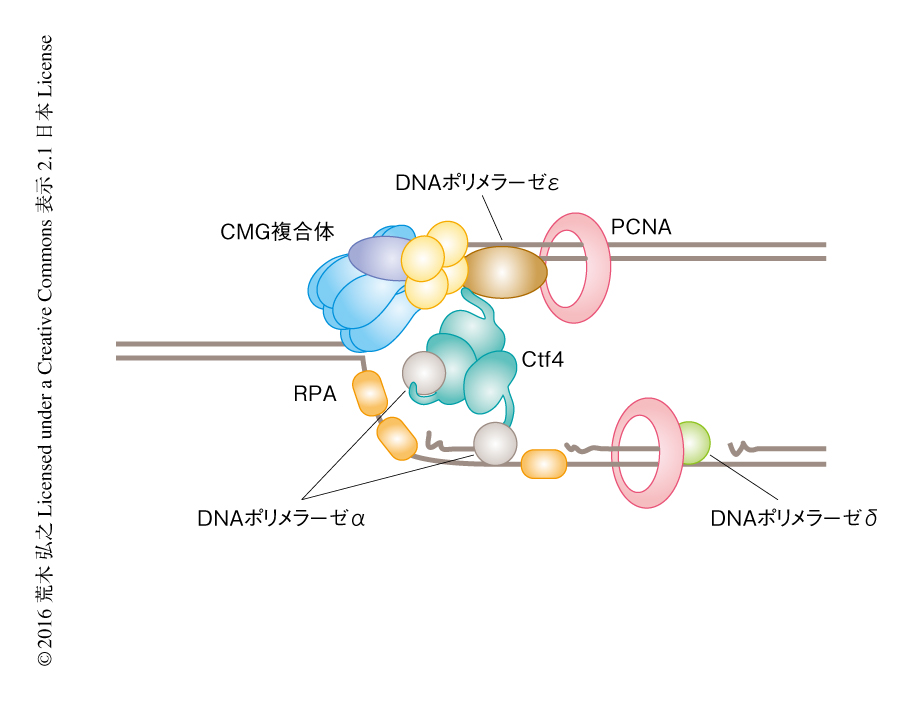
複製開始点において2本鎖DNAが1本鎖DNAにほどかれると,こんどはDNAポリメラーゼの出番である.真核生物における染色体DNAの複製にはDNAポリメラーゼα,DNAポリメラーゼδ,DNAポリメラーゼεが必要であり,これらのDNAポリメラーゼがどのように複製開始点にリクルートされるのか詳細は不明であるが,DNAヘリカーゼ活性をもつCMG複合体やそれに結合するCtf4とDNAポリメラーゼとの結合が重要なはたらきをすることが推測される26).
複製開始点においてまずはたらくのはDNAポリメラーゼαである.DNAポリメラーゼαはプライマーRNAを合成するプライマーゼと強固な複合体を形成しており,このプライマーゼの合成したRNAにつづいて新たなDNA鎖を合成する.DNAポリメラーゼαはCtf4(哺乳類においては,And1)と結合する.Ctf4はDNAポリメラーゼαと結合する領域においてCMG複合体に含まれるGINS複合体とも結合する.さらに,Ctf4はホモ三量体を形成するため,2分子のDNAポリメラーゼαをCMG複合体を介して複製フォークにとどめ,DNAポリメラーゼαが効率よく使われるようにすると考えられている27)(図4).
DNAポリメラーゼδおよびDNAポリメラーゼεはともにDNAポリメラーゼαの合成したDNAをプライマーとしてDNA鎖を伸長するが,DNAポリメラーゼεもGINS複合体のサブユニットであるPsf1と強く結合するため,GINS複合体を介してCMG複合体と結合する28).また,DNAポリメラーゼεはロード前複合体の構成タンパク質として複製開始にはたらき,同時に,複製開始点にリクルートされると考えられる.そして,DNAポリメラーゼαおよびDNAポリメラーゼεがDNAヘリカーゼ活性をもつCMG複合体と同時に結合し,リーディング鎖およびラギング鎖の合成に関与するものと思われる.
DNAポリメラーゼδおよびDNAポリメラーゼεがDNA鎖を伸長する際には,5つのサブユニットからなるクランプローダーであるRFCがプライマーの3’末端に結合し,DNAポリメラーゼの鋳型DNAからの脱落を防止するリング状のスライディングクランプであるPCNAをロードする29)(図1e).これらDNAポリメラーゼがどのようにDNA合成を進めているかは長く議論のわかれるところであったが,DNAポリメラーゼδおよびDNAポリメラーゼεにおいてrNTPを取り込みやすい変異体が分離され,それらを用いた解析から,DNAポリメラーゼδがラギング鎖,DNAポリメラーゼεがリーディング鎖を主として合成すると結論された.この結論は,まちがったヌクレオチドを取り込みやすいDNAポリメラーゼの変異体を用いたこれまでの解析と一致した.しかし,rNTPの取り込みの解析にてDNAポリメラーゼの役割論は一件落着かと思いきや,rNTPを取り込んだDNA鎖の修復においてリーディング鎖とラギング鎖とのあいだには差があり,実際には,リーディング鎖およびラギング鎖ともDNAポリメラーゼδがおもに合成しているとする報告がなされて,この問題はまた混沌としたなかに放り込まれている30).DNAポリメラーゼεがリーディング鎖を合成し,DNAヘリカーゼ活性をもつCMG複合体と効率よく共役しているとの報告もあるが,DNAポリメラーゼδとDNAポリメラーゼεの役割分担がはっきりしないため評価のわかれるところである.現在のところ,DNAポリメラーゼδがどのように複製開始点にリクルートされるのかはわからない.
リーディング鎖は複製フォークの進行方向と同じ方向に進み連続したDNA合成が可能であるが,ラギング鎖は複製フォークの進行方向とは反対の方向に進むため不連続なDNA合成が起こる.真核生物においてはラギング鎖の長さがヌクレオソームにより規定されており,これはヌクレオソームほぼ1つ分に相当する165 bpとなる31).DNAポリメラーゼαはRNAプライマーを用いて30~50ヌクレオチドのDNA鎖を合成するが,ラギング鎖ではDNAポリメラーゼαがDNA鎖の合成をはじめるので,ラギング鎖の長さを考慮するとラギング鎖の1/4~1/3を合成していることになる.一方,DNAポリメラーゼδおよびDNAポリメラーゼεはあやまって取り込んだヌクレオチドを除く校正用のエキソヌクレアーゼ活性をもつが,DNAポリメラーゼαはこの活性をもたない.したがって,DNAポリメラーゼαの合成したDNA鎖の多くはDNAポリメラーゼδやDNAポリメラーゼεにより除かれているのではないかという指摘もある.
ラギング鎖はプライマーRNAが除かれたのちDNAリガーゼにより連結される.RNAの除去には複数のステップが考えられている32).RNase HはRNA-DNAハイブリッド鎖のRNAを分解する酵素で,この酵素がプライマーRNAを除去すると考えられがちであるが,RNase Hはリボヌクレオチドからデオキシヌクレオチドの結合部分のリボヌクレオチドを除去することができない.実際には,DNAポリメラーゼδがDNA合成の過程においてプライマーRNAにぶつかると,RNAを鋳型DNAからはがし,5’→3’のエキソヌクレアーゼ活性をもつフラップエンドヌクレアーゼであるFEN1がRNAの部分を含む1本鎖を除く.DNAポリメラーゼδによりはがされた1本鎖が長いとヘリカーゼ活性およびヌクレアーゼ活性をもつDna2が付け根の部分を切断する.これらの反応により隙間ができたならDNAポリメラーゼδがうめたのち,DNAリガーゼにより先行のDNA鎖と連結される.
最近になり,真核生物における複製開始の機構の研究において大きな進展があった.それは,個々の複製タンパク質の分子機構とそれらの集合による複製の機構の解明である.今後,in vitro DNA複製系の構築により個々の複製タンパク質の分子機能は急速に解明されていくものと思われる.現在はまだ酵母を主体としたものであるが,基本的な機構はヒトを含む多細胞生物でも似ており,多細胞生物における分子機構の解明も近いことを期待している.一方で,染色体DNAの複製にともない起こる反応,たとえば,複製の際のクロマチン構造の維持,染色体分配に必要なコヒーシンのロードなどと複製との関係は今後の課題である.
略歴:1982年 大阪大学大学院理学研究科 修了,同年 大阪大学工学部 助手,1988年~1990年 米国NIH National Institute of Environmental Health SciencesにてVisiting Associate,1992年 大阪大学微生物病研究所 助教授を経て,1998年より国立遺伝学研究所 教授.
研究テーマ:染色体DNAの複製の機構に興味をもち,酵母を用いて研究を進めてきた.現在は,複製のみならず,複製とともに染色体において起こる反応にも興味を広げ,複製との関係を基礎に解析を進めている.
研究室URL:https://www.nig.ac.jp/labs/MicGen/
© 2016 荒木 弘之 Licensed under CC 表示 2.1 日本
(国立遺伝学研究所 微生物遺伝研究部門)
email:荒木弘之
領域融合レビュー, 5, e006 (2016) DOI: 10.7875/leading.author.5.e006
Hiroyuki Araki: Molecular mechanism of the initiation of chromosomal DNA replication in eukaryotes.
要 約
真核生物における染色体DNAの複製は特定の領域から開始する.この複製開始点にはOrcが結合し,DNAヘリカーゼの触媒コアであるMcm2-7へテロ六量体が主としてG1期にロードされる.複製開始の過程が細胞周期により制御されることによりDNA複製と細胞分裂は共役するが,これには細胞周期の制御の鍵となるサイクリン依存性キナーゼおよびDbf4依存性キナーゼによる複製タンパク質のリン酸化が重要な役割を担う.すなわち,これらリン酸化によりCdc45とGINS複合体とが結合したCMG複合体が複製開始点に形成され,複製において活性をもつDNAヘリカーゼとしてはたらくようになることが複製開始の制御において要となる.そして,DNAポリメラーゼがリクルートされDNA複製が開始する.現時点において詳細は不明だが,Mcm2-7へテロ六量体およびCMG複合体の構造,そして,in vitroにおける再構築により,CMG複合体の形成の機構を類推できるようになってきており,複製開始の機構について全容の解明が近づいている.
はじめに
生命現象を分子レベルの機構として知りたいとき,どのように研究を進めるだろうか? クラッシックには,1)反応に関与するタンパク質をみつけ,2)そのタンパク質を除いたり変異をくわえたりすることによる変化から反応機構を推察しモデルを構築する.さらに,3)単離したタンパク質を組み合わせることにより反応をin vitroにおいて再構築することができれば,モデルの証明と同時にさらに詳細な機構を知ることができる.真核生物におけるDNA複製の機構の研究は,ウイルスを用いたin vitro DNA複製系が構築され,3)の段階にまで到達した.しかし,ウイルスは宿主細胞の複製系そのものではなくウイルス独自の機構をもつため,細胞のなかで起こる複製反応を知るには不十分であった.また,アフリカツメガエルの卵抽出液を用いた複製系も構築され解析も進んだが,2)の段階でとまっている.一方,シンプルな単細胞の真核生物である酵母をモデルとした研究においては,遺伝学的な手法による1)や2)の段階の研究が進み,現在,3)の段階に進みつつある.
一方で,タンパク質の構造解析の技術の進展がめざましい.X線による結晶解析やNMRによる溶液における構造解析にくわえ,近年,電子顕微鏡を用いた単粒子解析の技術が急速に進み巨大なタンパク質の構造が原子レベルで決定されるようになってきた.これらの構造と,さきに述べた解析を組み合わせることにより,真核生物における分子レベルでの複製機構の理解は,今世紀にはいって急激に進んでいる.ここでは,最近の研究を中心に,真核生物における染色体DNAの複製開始の機構を解説する.
1.複製開始点
染色体DNAの複製は染色体に散在する特定の部位である複製開始点あるいは複製開始領域から開始する.複製開始点の数は酵母で約1000,ヒトでは数万になる.その部位を規定するのは6つのサブユニットからなるOrcである.OrcはATPと結合し加水分解するATPaseであり,2本鎖DNAと結合する.酵母からヒトまでよく保存されており,酵母やショウジョウバエのOrcは電子顕微鏡による単粒子解析やX線結晶構造解析からその構造が明らかにされている1-3).このOrcの性質が複製開始点を決めるうえで重要な役割を担う.出芽酵母(Saccharomyces cerevisiae)においては特定の11 bpの配列であるACSと結合するが,分裂酵母(Schizosaccharomyces pombe)においてはOrc4がATフックとよばれる構造をもつためATを多く含む領域と結合する4,5).しかし,ヒトやショウジョウバエのOrcは特定の塩基配列を認識しない.したがって,酵母ではDNA配列により複製開始点が規定されるが,多細胞生物ではDNA配列よりむしろクロマチンの状態などによりOrcの結合部位が決まると考えられている6).実際,Orcのもっとも大きなサブユニットであるOrc1はヒストンとの結合部位をもち,その部位を除くと染色体におけるOrcの結合様式が変化する7).また,Orcと染色体DNAとの結合は,出芽酵母においては細胞周期をつうじて起こるが,分裂酵母においては細胞周期により結合の状態が変わるとの報告もある.さらに,哺乳類においてOrc1はG1期から結合しM期には分解される5).
2.Mcm2-7へテロ六量体の複製開始点へのロード
DNA鎖を合成するDNAポリメラーゼは2本鎖DNAに対してDNA鎖を合成することはできない.DNA鎖の合成をはじめるには,鋳型となる1本鎖DNAおよびプライマーが必要である.そのため,複製開始点において2本鎖DNAが1本鎖DNAにほどかれないかぎりDNAの合成は開始されない.大腸菌においては複製開始点に複数のDnaAが結合するとその近傍の2本鎖DNAがほどかれるが,真核生物においてはOrcの結合により複製開始点において2本鎖DNAがほどかれるという報告はない.
真核生物においてはサイクリン依存性キナーゼの活性の低いM期の後期からG1期にかけて,2本鎖DNAを1本鎖DNAにほどくDNAヘリカーゼの触媒コアとなるMcm2-7へテロ六量体がOrcの結合した複製開始点にロードされる(図1a).この染色体DNAにおけるOrcおよびMcm2-7へテロ六量体からなる構造を複製開始前複合体(pre-replicative complexあるいはpre-replication complex:pre-RC)とよぶ.複製開始前複合体の形成には,OrcおよびMcm2-7へテロ六量体にくわえCdc6およびCdt1が必要である.出芽酵母においてCdt1はMcm2-7へテロ六量体と複合体を形成しており,Cdc6はOrcとともにはたらく.Orc,Mcm2-7へテロ六量体,Cdc6はATPase活性をもち,ATP加水分解のエネルギーを用いてMcm2-7へテロ六量体を複製開始点にロードする.
Mcm2-7へテロ六量体のそれぞれのサブユニットは共通する特徴をもつ.すなわち,N末端側にOBフォールドおよびジンクフィンガードメイン,C末端側にAAA+ファミリーに属するATPaseドメインが位置する.複製開始点には2分子のMcm2-7ヘテロ6量体のN末端側がむかい合うようにロードされるが,電子顕微鏡による単粒子解析などからおのおののサブユニットの配置が報告されている2)(図2).そして,Mcm2とMcm5とのあいだに隙間が生じ,この部分を用いて2本鎖DNAにロードされると考えられている.また,Mcm2-Mcm6の側部にも隙間が観察される.さらに,相対するMcm5とMcm7のN末端およびジンクフィンガードメインの相互作用により結合する8).
Mcm2-7へテロ六量体が複製開始点へロードされる機構は,出芽酵母において精製タンパク質を用いた再構成系により解析されている.残念ながら,一対のMcm2-7へテロ六量体がどのように複製開始点へロードされるかはまだ完全にはわかっていない.ロードされたMcm2-7へテロ六量体は必ず対になっているため,一対のMcm2-7へテロ六量体がいちどにロードされるのではないかとの説もあったが,1分子観察などによると,まず1分子のMcm2-7へテロ六量体がロードされ,すぐにつぎの1分子のMcm2-7へテロ六量体がロードされており9),Mcm2-7へテロ六量体のロードが正常に起こっているかどうかの検知機構もあるようである10,11).いずれにせよ,厳密な機構,とくに,2分子目のMcm2-7へテロ六量体のロードについては不明な点が多く,今後の研究を待たなければならない.
3.Mcm2-7へテロ六量体のもつDNAヘリカーゼ活性の活性化
Mcm2-7へテロ六量体はDNAヘリカーゼ活性を担うが,Mcm2-7へテロ六量体だけでは活性を示さない(生物種によっては,いくつかのサブユニットからなる複合体が弱い活性を示すものもある).DNAヘリカーゼの活性化にはG1期の後期からS期において活性化されるサイクリン依存性キナーゼおよびDbf4依存性キナーゼによるリン酸化,および,それにともなう複数のタンパク質の複製開始点への集合が必要であることが出芽酵母を用いた解析からわかってきた.ここでは,Mcm2-7へテロ六量体のもつDNAヘリカーゼ活性の活性化について,出芽酵母を中心に説明する.
Dbf4依存性キナーゼはCdc7を触媒サブユニットとし,制御サブユニットであるDbf4と複合体を形成する.G1期の後期にDBF4遺伝子の転写が活性化され,Dbf4のタンパク質量が増加することによりDbf4依存性キナーゼは活性化される.活性化されたDbf4依存性キナーゼはMcm2,Mcm4,Mcm6のN末端側の構造をとらない領域にある複数のSerおよびThrをリン酸化する.とくに,Mcm6のDbf4依存性キナーゼによるリン酸化はSld3-Sld7複合体とCdc45との複合体の複製開始点との結合を促進する12)(図1b).
Sld3-Sld7複合体は2分子のSld3がそのN末端領域においてSld7二量体により結合された強固な構造をとる13)(図2a).Sld3の中央にはCdc45との結合領域があり,1分子のCdc45が1分子のSld3と結合するため14),Sld3-Sld7複合体は2分子のCdc45と結合する.細胞においてSld3-Sld7複合体とCdc45は互いに依存して複製開始点と結合するが15,16),in vitroにおいて,Sld3-Sld7複合体はCdc45がなくても複製開始点と結合できるが,Cdc45の複製開始点との結合にはSld3が必要である12).このように,Dbf4依存性キナーゼによるMcm6のリン酸化によりSld3-Sld7複合体とCdc45との複合体がMcm2-7へテロ六量体と結合する.筆者らは,Sld3-Sld7複合体の構造からその配置を推測しており(図2b),この配置は,のちに述べる活性化型のDNAヘリカーゼの形成にも都合がよいものとなっている.
サイクリン依存性キナーゼは出芽酵母においてはSld2およびSld3をリン酸化することにより複製開始を促進する17).Sld2にはサイクリン依存性キナーゼによりリン酸化されうる11箇所の部位が存在する.このうち,Thr84がサイクリン依存性キナーゼによりリン酸化されるとDpb11と結合する.Dpb11はC末端側およびN末端側にタンデムにならんだBRCT領域をもち,このタンデムにならんだBRCT領域はリン酸化されたペプチドと結合することが知られている.リン酸化されたSld2はDpb11のC末端側にあるタンデムにならんだBRCT領域と結合する.一方,Dpb11のN末端側にあるタンデムにならんだBRCT領域はリン酸化されたSld3と結合する.Sld3もサイクリン依存性キナーゼによりリン酸化されうる12箇所の部位をもつが,C末端側にあるThr600およびSer622のリン酸化がDpb11との結合に関与する.どちらか一方がリン酸化されれば結合は可能であるが,両方ともリン酸化されると強く結合する.
サイクリン依存性キナーゼによりリン酸化されたSld2およびSld3がDpb11と結合することにより,どのようして複製開始が促進されるのであろうか.その説明のまえに,活性型のDNAヘリカーゼについて説明したほうが理解しやすいだろう.Mcm2-7へテロ六量体はDNAヘリカーゼとしての活性をもつが,さきに述べたように,Mcm2-7へテロ六量体だけでは活性を示さない.細胞においては,Mcm2-7へテロ六量体はCdc45およびGINS複合体とCMG複合体とよばれる強固な複合体を形成し,このCMG複合体が複製において活性をもつDNAヘリカーゼとしてはたらく18).さきに述べたように,Cdc45はSld3-Sld7複合体と結合し,Mcm2-7へテロ六量体に相互依存的にリクルートされる.GINS複合体はSld5,Psf1,Psf2,Psf3の4つのサブユニットからなる複合体で,筆者らにより,末尾の数字の日本語読みでgo,ichi,nii,sanと名づけられたものである.Sld3はCdc45のリクルートおよびGINS複合体の複製開始点へのリクルートに必要である.そして,筆者らにより,Sld2のサイクリン依存性キナーゼによるリン酸化もGINS複合体のリクルートに関与していることが明らかにされ,この機構についても解析した.その結果,リン酸化されたSld2がDpb11と結合すると,DNAポリメラーゼεおよびGINS複合体がこの複合体にさらにくわわることが示された.この複合体をロード前複合体(pre-loading complex:pre-LC)と名づけ,ロード前複合体に含まれるDpb11が複製開始点においてリン酸化されたSld3と結合することにより,GINS複合体は複製開始点にリクルートされるものと考えている19)(図1c).DNAポリメラーゼεは細胞周期をつうじGINS複合体と安定な複合体を形成しており,DNAポリメラーゼεおよびGINS複合体はDpb11と弱く相互作用する.これらの結合がSld2により安定化されているのであろう.
哺乳類においてはDpb11と対応するBRCT領域を9つもつTopBP1が知られていたが,TopBP1の結合タンパク質としてSld3に対応するTreslin/Ticrrがみつかった20,21).アフリカツメガエルの卵抽出液を用いた解析により,TopBP1およびTreslinは独立にクロマチンと結合するが,TopBP1とTreslinとの結合はサイクリン依存性キナーゼによるTreslinのリン酸化により起こり,複製開始に必須であることがわかった.さらに,TreslinにはMTBPが結合し,このMTBPも複製開始に必須であることが示されている22).これがSld3と複合体を形成するSld7と同じはたらきをしているかどうかはわからない.一方,哺乳類においてSld2と弱い相同性を示すタンパク質としてRecQL4が知られている.RecQL4はC末端側にヘリカーゼドメインをもち,このドメインの欠損は早老症のひとつであるRothmund-Thomson症候群の発症の原因となる.ヘリカーゼドメインは細胞の増殖には必須ではないが,Sld2と相同性を示すN末端側の領域は必須である17).アフリカツメガエルの卵抽出液を用いた解析から,RecQL4は複製開始の後半においてはたらいているようで,DNAポリメラーゼαのリクルートに必要との報告もあり,今後の解析が必要である.
複製開始点に結合したMcm2-7へテロ六量体,Cdc45,GINS複合体は強固なCMG複合体を形成する18)(図1c).しかし,CMG複合体の形成はすぐには複製開始にはつながらない.それは,Mcm10を欠くとCMG複合体様の強固な複合体は形成されるが,複製開始は起こらないからである.Mcm10は一対のCMG複合体が両方向にむけ2本鎖DNAをほどくようにするはたらきがあると考えられているが(図1d),分子機能はわかっていない23).Mcm10の機能は複製開始における残されたミステリーである.
強固なCMG複合体ができる過程はまだわからないが,Mcm2-7へテロ六量体およびCMG複合体の構造は電子顕微鏡による単粒子解析から明らかにされている8,24,25).GINS複合体およびCdc45はMcm2-7へテロ六量体のMcm2とMcm5とのあいだの隙間をうめるよう位置する(図3).さらに,N末端側がむかい合うよう形成された一対のMcm2-7へテロ六量体の位置関係からは,Mcm2とMcm5とのあいだの隙間は反対の方向をむいている(図2b).Mcm2-7へテロ六量体をDNAにロードさせ電子顕微鏡により観察すると,2本鎖DNAをとおすかたちにみえる.一方で,CMG複合体におけるDNAヘリカーゼ活性の解析から,CMG複合体は1本鎖DNAのうえを3’側から5’側の方向に動いていることがわかった(図4).すなわち,CMG複合体はリーディング鎖の鋳型DNAのうえを複製の方向に動いていることになる.これらのことから,Mcm2-7へテロ六量体はまず2本鎖DNAをリングにとおし,活性化された時点あるいはCMG複合体が形成された時点で1本鎖DNAをリングにとおす.このとき,Mcm2とMcm5とのあいだの隙間の位置が同一の方向をむかないのは,複製開始点にロードされた一対のMcm2-7へテロ六量体あるいはCMG複合体が2本鎖DNAから1本鎖DNAへと乗り換えるのには都合がよいからかもしれない.
4.複製フォークの形成
複製開始点において2本鎖DNAが1本鎖DNAにほどかれると,こんどはDNAポリメラーゼの出番である.真核生物における染色体DNAの複製にはDNAポリメラーゼα,DNAポリメラーゼδ,DNAポリメラーゼεが必要であり,これらのDNAポリメラーゼがどのように複製開始点にリクルートされるのか詳細は不明であるが,DNAヘリカーゼ活性をもつCMG複合体やそれに結合するCtf4とDNAポリメラーゼとの結合が重要なはたらきをすることが推測される26).
複製開始点においてまずはたらくのはDNAポリメラーゼαである.DNAポリメラーゼαはプライマーRNAを合成するプライマーゼと強固な複合体を形成しており,このプライマーゼの合成したRNAにつづいて新たなDNA鎖を合成する.DNAポリメラーゼαはCtf4(哺乳類においては,And1)と結合する.Ctf4はDNAポリメラーゼαと結合する領域においてCMG複合体に含まれるGINS複合体とも結合する.さらに,Ctf4はホモ三量体を形成するため,2分子のDNAポリメラーゼαをCMG複合体を介して複製フォークにとどめ,DNAポリメラーゼαが効率よく使われるようにすると考えられている27)(図4).
DNAポリメラーゼδおよびDNAポリメラーゼεはともにDNAポリメラーゼαの合成したDNAをプライマーとしてDNA鎖を伸長するが,DNAポリメラーゼεもGINS複合体のサブユニットであるPsf1と強く結合するため,GINS複合体を介してCMG複合体と結合する28).また,DNAポリメラーゼεはロード前複合体の構成タンパク質として複製開始にはたらき,同時に,複製開始点にリクルートされると考えられる.そして,DNAポリメラーゼαおよびDNAポリメラーゼεがDNAヘリカーゼ活性をもつCMG複合体と同時に結合し,リーディング鎖およびラギング鎖の合成に関与するものと思われる.
DNAポリメラーゼδおよびDNAポリメラーゼεがDNA鎖を伸長する際には,5つのサブユニットからなるクランプローダーであるRFCがプライマーの3’末端に結合し,DNAポリメラーゼの鋳型DNAからの脱落を防止するリング状のスライディングクランプであるPCNAをロードする29)(図1e).これらDNAポリメラーゼがどのようにDNA合成を進めているかは長く議論のわかれるところであったが,DNAポリメラーゼδおよびDNAポリメラーゼεにおいてrNTPを取り込みやすい変異体が分離され,それらを用いた解析から,DNAポリメラーゼδがラギング鎖,DNAポリメラーゼεがリーディング鎖を主として合成すると結論された.この結論は,まちがったヌクレオチドを取り込みやすいDNAポリメラーゼの変異体を用いたこれまでの解析と一致した.しかし,rNTPの取り込みの解析にてDNAポリメラーゼの役割論は一件落着かと思いきや,rNTPを取り込んだDNA鎖の修復においてリーディング鎖とラギング鎖とのあいだには差があり,実際には,リーディング鎖およびラギング鎖ともDNAポリメラーゼδがおもに合成しているとする報告がなされて,この問題はまた混沌としたなかに放り込まれている30).DNAポリメラーゼεがリーディング鎖を合成し,DNAヘリカーゼ活性をもつCMG複合体と効率よく共役しているとの報告もあるが,DNAポリメラーゼδとDNAポリメラーゼεの役割分担がはっきりしないため評価のわかれるところである.現在のところ,DNAポリメラーゼδがどのように複製開始点にリクルートされるのかはわからない.
リーディング鎖は複製フォークの進行方向と同じ方向に進み連続したDNA合成が可能であるが,ラギング鎖は複製フォークの進行方向とは反対の方向に進むため不連続なDNA合成が起こる.真核生物においてはラギング鎖の長さがヌクレオソームにより規定されており,これはヌクレオソームほぼ1つ分に相当する165 bpとなる31).DNAポリメラーゼαはRNAプライマーを用いて30~50ヌクレオチドのDNA鎖を合成するが,ラギング鎖ではDNAポリメラーゼαがDNA鎖の合成をはじめるので,ラギング鎖の長さを考慮するとラギング鎖の1/4~1/3を合成していることになる.一方,DNAポリメラーゼδおよびDNAポリメラーゼεはあやまって取り込んだヌクレオチドを除く校正用のエキソヌクレアーゼ活性をもつが,DNAポリメラーゼαはこの活性をもたない.したがって,DNAポリメラーゼαの合成したDNA鎖の多くはDNAポリメラーゼδやDNAポリメラーゼεにより除かれているのではないかという指摘もある.
ラギング鎖はプライマーRNAが除かれたのちDNAリガーゼにより連結される.RNAの除去には複数のステップが考えられている32).RNase HはRNA-DNAハイブリッド鎖のRNAを分解する酵素で,この酵素がプライマーRNAを除去すると考えられがちであるが,RNase Hはリボヌクレオチドからデオキシヌクレオチドの結合部分のリボヌクレオチドを除去することができない.実際には,DNAポリメラーゼδがDNA合成の過程においてプライマーRNAにぶつかると,RNAを鋳型DNAからはがし,5’→3’のエキソヌクレアーゼ活性をもつフラップエンドヌクレアーゼであるFEN1がRNAの部分を含む1本鎖を除く.DNAポリメラーゼδによりはがされた1本鎖が長いとヘリカーゼ活性およびヌクレアーゼ活性をもつDna2が付け根の部分を切断する.これらの反応により隙間ができたならDNAポリメラーゼδがうめたのち,DNAリガーゼにより先行のDNA鎖と連結される.
おわりに
最近になり,真核生物における複製開始の機構の研究において大きな進展があった.それは,個々の複製タンパク質の分子機構とそれらの集合による複製の機構の解明である.今後,in vitro DNA複製系の構築により個々の複製タンパク質の分子機能は急速に解明されていくものと思われる.現在はまだ酵母を主体としたものであるが,基本的な機構はヒトを含む多細胞生物でも似ており,多細胞生物における分子機構の解明も近いことを期待している.一方で,染色体DNAの複製にともない起こる反応,たとえば,複製の際のクロマチン構造の維持,染色体分配に必要なコヒーシンのロードなどと複製との関係は今後の課題である.
文 献
- Li, H. & Stillman, B.: The origin recognition complex: a biochemical and structural view. Subcell. Biochem., 62, 37-58 (2012)[PubMed]
- Sun, J., Yuan, Z., Stillman, B. et al.: Structure and function studies of replication factors. in The Initiation of DNA Replication in Eukaryotes (Kaplan, D. L. ed.), pp.427-441, Springer, Cham (2016)
- Bleichert, F., Botchan, M. R. & Berger, J. M.: Crystal structure of the eukaryotic origin recognition complex. Nature, 519, 321-326 (2015)[PubMed]
- Raghuraman, M. K. & Liachko, I.: Sequence determinants of yeast replication origins. in The Initiation of DNA Replication in Eukaryotes (Kaplan, D. L. ed.), pp.123-141, Springer, Cham (2016)
- Hoggard, T. & Fox, C. A.: The origin recomgnition complex in the initiation of DNA replication. in The Initiation of DNA Replication in Eukaryotes (Kaplan, D. L. ed.), pp.159-188, Springer, Cham (2016)
- Leonard, A. C. & Mechali, M.: DNA replication origins. Cold Spring Harb. Perspect. Biol., 5, a010116 (2013)[PubMed]
- Muller, P., Park, S., Shor, E. et al.: The conserved bromo-adjacent homology domain of yeast Orc1 functions in the selection of DNA replication origins within chromatin. Genes Dev., 24, 1418-1433 (2010)[PubMed]
- Li, N., Zhai, Y., Zhang, Y. et al.: Structure of the eukaryotic MCM complex at 3.8Å. Nature, 524, 186-191 (2015)[PubMed]
- Ticau, S., Friedman, L. J., Ivica, N. A. et al.: Single-molecule studies of origin licensing reveal mechanisms ensuring bidirectional helicase loading. Cell, 161, 513-525 (2015)[PubMed]
- Frigola, J., Remus, D., Mehanna, A. et al.: ATPase-dependent quality control of DNA replication origin licensing. Nature, 495, 339-343 (2013)[PubMed]
- Fernandez-Cid, A., Riera, A., Tognetti, S. et al.: An ORC/Cdc6/MCM2-7 complex is formed in a multistep reaction to serve as a platform for MCM double-hexamer assembly. Mol. Cell, 50, 577-588 (2013)[PubMed]
- Deegan, T. D., Yeeles, J. T. & Diffley, J. F.: Phosphopeptide binding by Sld3 links Dbf4-dependent kinase to MCM replicative helicase activation. EMBO J., 35, 961-973 (2016)[PubMed]
- Itou, H., Shirakihara, Y. & Araki, H.: The quaternary structure of the eukaryotic DNA replication proteins Sld7 and Sld3. Acta Crystallogr. D Biol. Crystallogr., 71, 1649-1656 (2015)[PubMed]
- Itou, H., Muramatsu, S., Shirakihara, Y. et al.: Crystal structure of the homology domain of the eukaryotic DNA replication proteins Sld3/Treslin. Structure, 22, 1341-1347 (2014)[PubMed]
- Kamimura, Y., Tak, Y. S., Sugino, A. et al.: Sld3, which interacts with Cdc45 (Sld4), functions for chromosomal DNA replication in Saccharomyces cerevisiae. EMBO J., 20, 2097-2107 (2001)[PubMed]
- Tanaka, T., Umemori, T., Endo, S. et al.: Sld7, an Sld3-associated protein required for efficient chromosomal DNA replication in budding yeast. EMBO J., 30, 2019-2030 (2011)[PubMed]
- Tanaka, S. & Araki, H.: Role of CDK in replication initiation. in The Initiation of DNA Replication in Eukaryotes (Kaplan, D. L. ed.), pp.263-278, Springer, Cham (2016)
- Moyer, S. E., Lewis, P. W. & Botchan, M. R.: Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc. Natl. Acad. Sci. USA, 103, 10236-10241 (2006)[PubMed]
- Muramatsu, S., Hirai, K., Tak, Y. S. et al.: CDK-dependent complex formation between replication proteins Dpb11, Sld2, Polε, and GINS in budding yeast. Genes Dev., 24, 602-612 (2010)[PubMed]
- Sansam, C. L., Cruz, N. M., Danielian, P. S. et al.: A vertebrate gene, ticrr, is an essential checkpoint and replication regulator. Genes Dev., 24, 183-194 (2010)[PubMed]
- Kumagai, A., Shevchenko, A., Shevchenko, A. et al.: Treslin collaborates with TopBP1 in triggering the initiation of DNA replication. Cell, 140, 349-359 (2010)[PubMed]
- Boos, D., Yekezare, M. & Diffley, J. F.: Identification of a heteromeric complex that promotes DNA replication origin firing in human cells. Science, 340, 981-984 (2013)[PubMed]
- Yeeles, J. T., Deegan, T. D., Janska, A. et al.: Regulated eukaryotic DNA replication origin firing with purified proteins. Nature, 519, 431-435 (2015)[PubMed]
- Abid Ali, F., Renault, L., Gannon, J. et al.: Cryo-EM structures of the eukaryotic replicative helicase bound to a translocation substrate. Nat. Commun., 7, 10708 (2016)[PubMed]
- Yuan, Z., Bai, L., Sun, J. et al.: Structure of the eukaryotic replicative CMG helicase suggests a pumpjack motion for translocation. Nat. Struct. Mol. Biol., 23, 217-224 (2016)[PubMed]
- Araki, H.: Molecular mechanism of DNA replication. in DNA Replication, Recombination, and Repair: Molecular Mechanisms and Pathology (Hanaoka, F. & Sugasawa, K. eds.), pp.3-22, Springer, Tokyo (2016)
- Simon, A. C., Zhou, J. C., Perera, R. L. et al.: A Ctf4 trimer couples the CMG helicase to DNA polymerase α in the eukaryotic replisome. Nature, 1-18 (2014)[PubMed]
- Sengupta, S., van Deursen, F., de Piccoli, G. et al.: Dpb2 integrates the leading-strand DNA polymerase into the eukaryotic replisome. Curr. Biol., 23, 543-552 (2013)[PubMed]
- Hedglin, M., Kumar, R. & Benkovic, S. J.: Replication clamps and clamp loaders. Cold Spring Harb. Perspect. Biol., 5, a010165 (2013)[PubMed]
- Burgers, P. M., Gordenin, D. & Kunkel, T. A.: Who is leading the replication fork, Polε or Polδ? Mol. Cell, 61, 492-493 (2016)[PubMed]
- Smith, D. J. & Whitehouse, I.: Intrinsic coupling of lagging-strand synthesis to chromatin assembly. Nature, 483, 434-438 (2012)[PubMed]
- Balakrishnan, L. & Bambara, R. A.: Okazaki fragment metabolism. Cold Spring Harb. Perspect. Biol., 5, a010173 (2013)[PubMed]
著者プロフィール
略歴:1982年 大阪大学大学院理学研究科 修了,同年 大阪大学工学部 助手,1988年~1990年 米国NIH National Institute of Environmental Health SciencesにてVisiting Associate,1992年 大阪大学微生物病研究所 助教授を経て,1998年より国立遺伝学研究所 教授.
研究テーマ:染色体DNAの複製の機構に興味をもち,酵母を用いて研究を進めてきた.現在は,複製のみならず,複製とともに染色体において起こる反応にも興味を広げ,複製との関係を基礎に解析を進めている.
研究室URL:https://www.nig.ac.jp/labs/MicGen/
© 2016 荒木 弘之 Licensed under CC 表示 2.1 日本