新時代:クライオ電子顕微鏡による近原子分解能での解析
2016/11/04
岩崎 憲治
(大阪大学蛋白質研究所 附属蛋白質解析先端研究センター分子創製学研究室)
email:岩崎憲治
領域融合レビュー, 5, e010 (2016) DOI: 10.7875/leading.author.5.e010
Keiji Iwasaki: A new era for cryo-electron microscopy: near atomic resolution.

ここ数年,クライオ電子顕微鏡を用いた単粒子再構成法が構造生物学者にとり魅力的な手法のひとつとなってきた.結晶をつくることなく原子モデルを構築することのできるほど高分解能の3次元構造が得られるようになってきたからである.データベースへの登録数は加速度的に増加しており,大きな膜タンパク質の構造やスプライソソームのような複雑かつ多数の分子からなる複合体の構造も近原子分解能で解かれた.このようなブレークスルーをもたらしたものとして,コンピューターにより制御され高性能のステージを搭載した専用の電子顕微鏡の開発,電子直接検出器の登場,これまでになかったアルゴリズムを取り入れた新しい解析ソフトウェアの登場,の3つがあげられる.近原子分解能とよばれる高分解能の達成においては電子直接検出器および解析ソフトウェアが重要であり,高い生産性でデータが報告されるようになったのは専用の電子顕微鏡の登場によるところが大きい.透過型電子顕微鏡の誕生から80年以上をへて,ようやく電子顕微鏡を用いた構造生物学への道が開けてきた.この急激な進歩をもたらした技術について解説する.
クライオ電子顕微鏡による生体分子の構造解析は,らせん対称性や2次元結晶を利用した方法により幕を開けた.しかし,クライオ電子顕微鏡に強くもとめられていたのは,結晶化や特殊な対称性を利用しない,より一般的な形状の分子が単分散した状態の画像から3次元構造をもとめる方法であった.歴史的には,そのような試みの最初は電子線トモグラフィーである.しかし,この手法は試料の同一の領域に電子線を照射しつづけるため,電子線による試料の損傷という電子顕微鏡による観察における最大の問題がネックとなり,いったんは姿を消した.つぎに,電子線による試料の損傷を克服する方法として単粒子再構成法が誕生した.そのよび方はさまざまで,日本語では単粒子解析法とよばれることがもっとも多く,英語ではとくに統一されていない.実際には多数の粒子を扱うので,日本語としては多粒子再構成法のほうが実際に近い.この手法により得られる構造は,正二十面体のキャプシドをもつウイルスのように高い対称性をもつ場合を除き,nmオーダーの分解能が一般であった.ところが,2013年の末に,(電子顕微鏡にとっては)小さな膜タンパク質であるTRPV1の原子モデルが報告されてから,近原子分解能での構造解析が爆発的に報告されるようになり,いまや,データベースへの登録数においてはNMR法をこえる勢いになってきている.このレビューにおいては,この劇的な進歩の背景,および,現在の単粒子再構成の実際について解説する1).
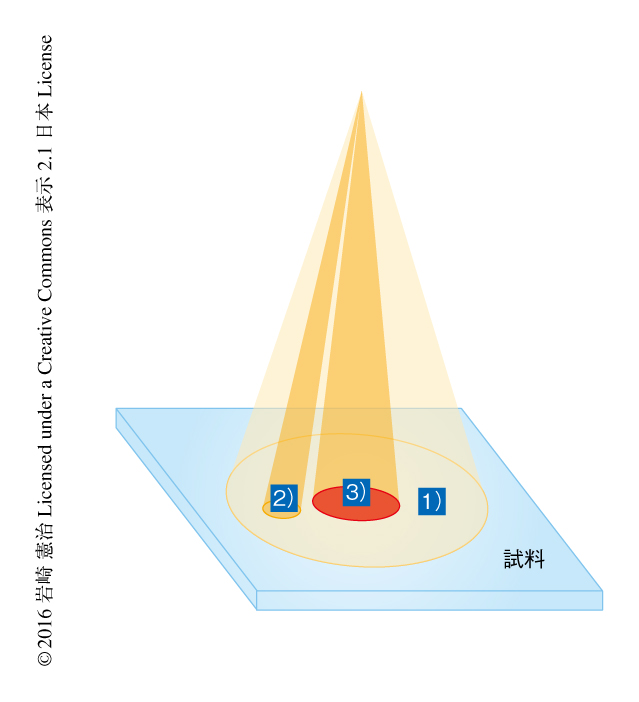
精製した生体分子やその複合体(これを“電顕屋”は粒子とよぶ)が溶液中で単分散している場合,この状態を電子顕微鏡により撮影することができれば,1枚の画像にさまざまな方向をむいた粒子画像が写ることになる.精製して1種類の試料を単離したわけであるから,すべて同じものである.よって,同一の分子をいろいろな方向から撮影したのと同じ粒子画像がいちどに得られるわけである.電子顕微鏡は電子線の照射により試料を破壊するが,とくに軽元素からなる生体試料は損傷をうけやすい.しかし,これならば,たったいちどの照射により画像データを取得できるので,損傷を最小限に抑えることができる.場所を変えて撮影すれば,さらにさまざまな向きの粒子画像が得られる.単粒子再構成法の誕生は1975年とみてもよいだろう2).実際には,たったいちどの照射といえども試料を破壊する.高分解能で解析したければ,高分解能の情報が失われないよう,しかし,解析の可能なシグナルが得られるよう,ギリギリの量の電子線で撮影する(図1).そのため,シグナル/ノイズ比が極端に低い画像しか得られない.このことこそが,高分解能での解析を困難にしてきたおもな要因である.特殊な画像解析が必要になり,一般的な画像解析の技術はあまり適用できない.十分なシグナル/ノイズ比の構造情報を得るには粒子画像の平均化が必要になるが3),このため数千から数万枚の粒子画像,最近では数十万の粒子画像を扱うようになってきている.
クライオ電子顕微鏡においては非晶質層の氷に閉じ込めた試料を観察する.画像を撮影するため電子線を照射すると,そのとたんに試料が動く.その原因について結論は得られていないが,この問題の克服のため詳細な調査が行われている4).さらに,熱の流入による試料ステージの機械的な微動も大いに問題である.試料の破壊を最小限に抑えるため弱い電子線を照射する→露光時間が長くなる→ブレた像が記録される,という悪循環におちいりがちである.こうした撮影対象の微動が高分解能での解析をさまたげている第2の要因である.
また,精製したからといってタンパク質のコンフォメーションがすべて同じでないことはいうまでもない.それでも,Åオーダーの分解能で解析するためには,粒子がÅオーダーのレベルでそろった構造をもつ粒子画像をあつめる必要がある.別の考えとしては,“動いている”部分は分解能が悪いはずであるので,得られた分子構造の分解能を場所に依存して評価すればよいという考え方もある.
分子全体ではないが,一部は側鎖の密度が明瞭に観察されるなど,原子モデルを構築するのに十分な分解能をもつ密度図(電子顕微鏡の場合に得られるのは,“電子”密度図ではない)を近原子分解能とよぶ.結晶をつくらずに,あるいは,らせん対称性という特殊な構造を利用せずに,粒子画像として近原子分解能を達成できるとの理論が報告されたのは1995年のことであり,この報告は単粒子再構成法による原子分解能の実現への取り組みのよりどころとなった3).ここから,正二十面体のキャプシドをもつウイルスを対象にした高分解能での解析への取り組みがつづいた.正二十面体のキャプシドは安定しており,大きな複合体であるので弱い電子線でも十分なシグナル/ノイズ比が得られる.1998年,2つの研究室から,B型肝炎ウイルスのキャプシドにおいてαヘリックスの可視化に成功したという報告がなされた5,6).それから10年をへた2008年,正二十面体のウイルス粒子を使った近原子分解能での解析が報告された7,8).そして2010年,de novoの原子モデリングの報告がなされた9).さらに大きな転機は2013年末にやってきた.小さな対称性の低い膜タンパク質TRPV1の近原子分解能での構造が報告されたのである10,11).これを契機に,近原子分解能での報告は爆発的に増加した.2015年,βガラクトシダーゼの分解能2.2Åでの構造が報告され,ここにおいて,分子に配位した水分子が可視化された12).そして2016年,100 kDaよりも小さい分子について近原子分解能での解析が報告され,クライオ電子顕微鏡による構造解析にブレークスルーが起こったことが確かめられた13).さきに述べた1995年の予測において,原子分解能に達する最小の大きさは100 kDaとされており,この大きさにいどむことができたのである.呼吸鎖複合体Iなど巨大な膜タンパク質についてもあいついで近原子分解能での構造が報告されており,その勢いは加速の一途である.
論文にはおもてだってでてこないが,操作性の向上を実現した専用のクライオ電子顕微鏡の登場が近原子分解能の達成に貢献している事実は疑いようがない.新たなクライオ電子顕微鏡により,習熟したオペレーターのみが腕をかけて寝る間も惜しまず撮影するという生産性の低い状況はさまがわりした.以前からのもっともポピュラーなサイドエントリーという方式のクライオ電子顕微鏡では,2~3時間おきに手動で液体窒素を足す必要がある.よい試料にあたったときには,オペレーターが電子顕微鏡のまえに座り込み,液体窒素を足しながら撮りつづけるのである.さらに悪いことに,いちどに1個の試料しか観察できない.操作に習熟すればいったん観察した試料も回収できるが,その操作により大気中の水分が結晶状になって試料に混入するため必ず汚れてしまう.試料の出し入れに使用するホルダーは試料の交換のときに大気中に取り出すので霜だらけになり,完全に乾燥させるには何時間もかかる.現在のクライオ電子顕微鏡の作業のなかでもっとも重要かつ時間のかかるステップは,粒子が単分散した薄い非晶質層の氷の試料をつくることである.さまざまな条件で作製しつぎつぎに試さねばならない.しかし,この作業はサイドエントリーホルダーを使用していては非常に時間がかかる.このような効率の悪さは,米国FEI社(現 米国Thermo Fisher Scientific社)のTitan Kriosの登場により一変した(図2).2015年から2016年9月まで,データベースへ登録されたデータの取得に使われた電子顕微鏡としては圧倒的なシェアであった.電子顕微鏡はコンピューターにより制御されており,いちどに12個の試料を装填できる.試料の交換はマウスのクリックひとつででき,撮影したのちの凍結試料の回収も可能である.これができるとできないとでは,データ取得の速度に雲泥の差がある.液体窒素の自動供給装置がついているため,好きなときに撮影を中断することもできる.さらに,なんといっても自動撮影機構を備えていて,夕方にセットすれば翌朝には500枚近くの画像が取得されている.試料ステージが非常に安定しているので,機械的な微動はきわめて少ない.このハイエンドな電子顕微鏡には,高分解能での画像が多く必要な単粒子再構成法を強力に推進する機構が満載されている.
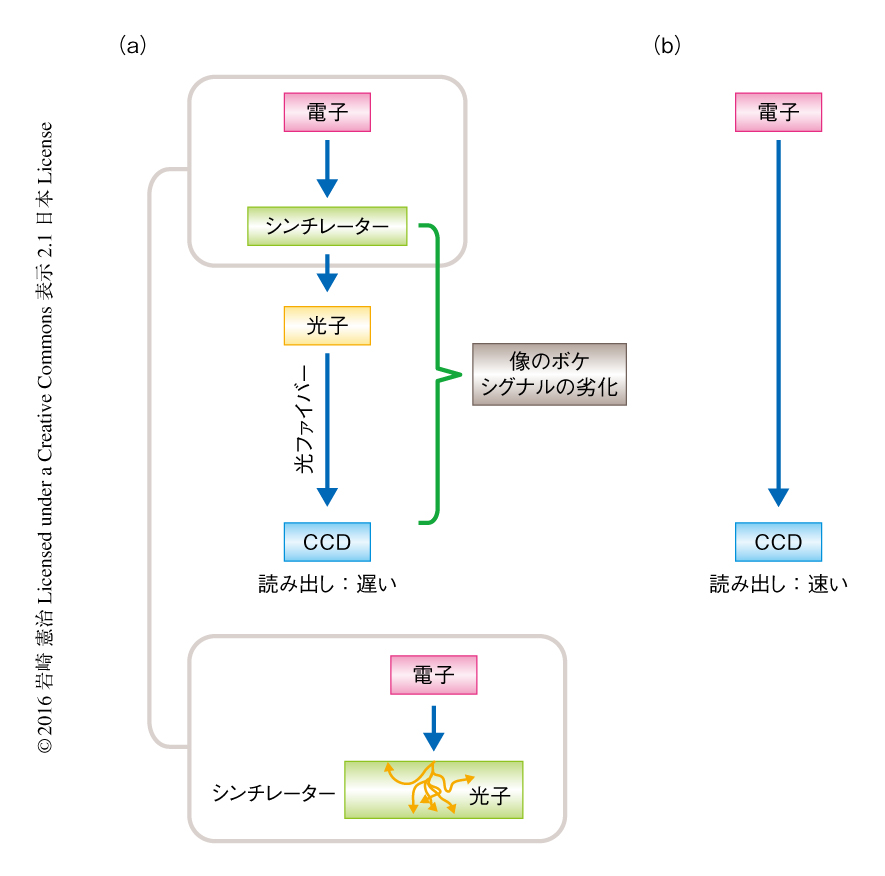
近原子分解能での解析の主役は,電子直接検出器といっても過言ではない.これまで,ファイバーカップルCCDカメラが記録装置として多く使われてきており,現在も,大部分の研究室はこれを使用している.高分解能での情報の記録媒体としてはフィルムのほうがすぐれているものの,フィルムより効率がよくコンピューターにより制御できるため主役の座を奪ってしまった.しかし,そのしくみから,画像がどうしてもボケてしまう.電子線をシンチレーターにより光子に変換し,これが光ファイバーにより伝達されCCD素子により検出される14)(図3).このプロセスにおいて像はボケてしまうのである.さらに,CCDにおける電荷転送のしくみから,どうしても読み出しの速度が遅くなる.そのため,動いている試料を1枚の写真として記録するので,おおげさにいえば,夜に車のヘッドライトを長い露光時間で撮影するようなものである.このような事情から,電子を直接に検出する装置が切望されていた.そこで,CMOS(complementary metal oxide semiconductor,相補型金属酸化膜半導体)を使った電子直接検出器が誕生したのである15).もちろん,光-電子変換がないのでボケが少ない.さらに,読み取りの速度が格段に速い.これにより,第2世代の電子直接検出器とよばれる,入射する電子の計数を可能にした電子直接検出器が開発された.記録装置が像にくわえるノイズはこれで一挙に減少した.
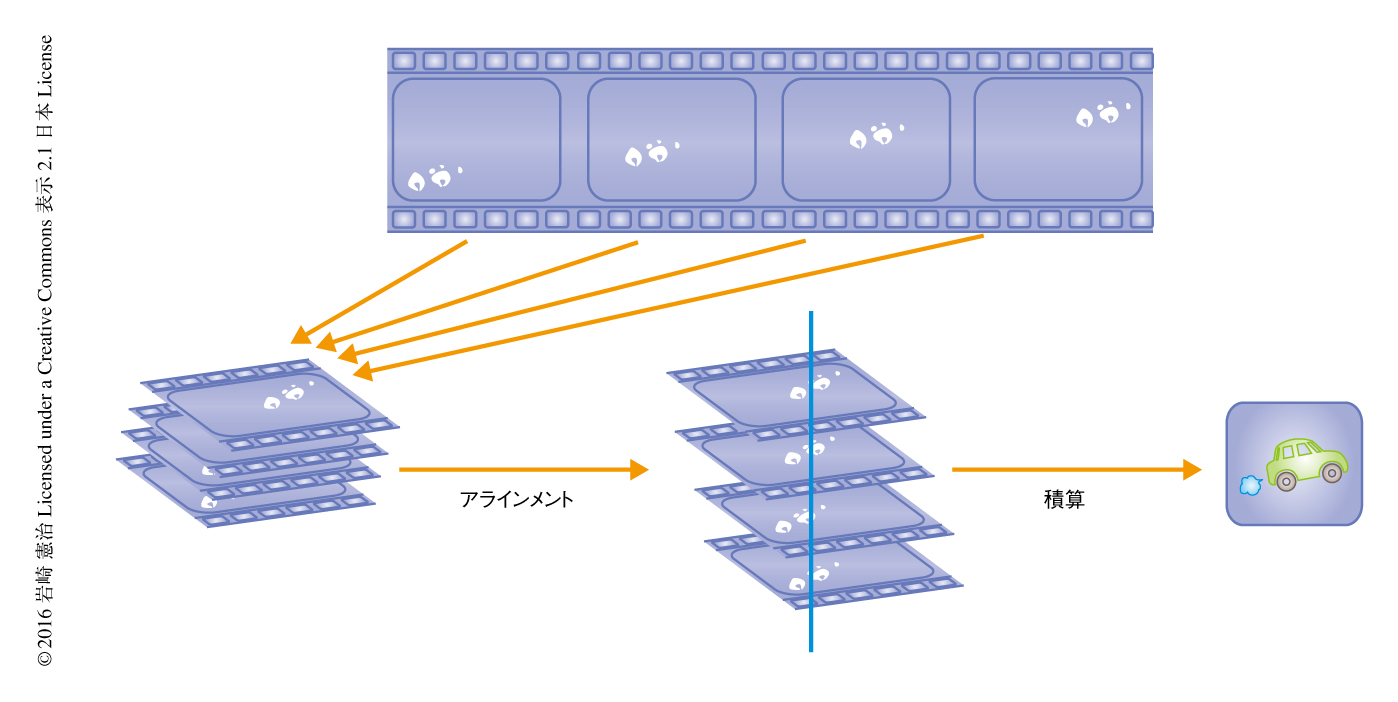
高速度での撮影が可能になったことにより新しい撮影法が生まれた.動画撮影法である4,16)(図4).試料の微動がさけられないなら,動画として撮影し,1コマ1コマ(サブフレームとよぶ)をアラインメントして足し合わせて1枚の画像にすればよい16).とくに,電子を試料に照射したとたんに起こる微動については,最初の1,2枚のサブフレームを使わなければよい.また,多くのサブフレームを撮影し,解析においては十分なシグナルをかせぐためすべてのサブフレームを足し合わせ,最後に電子線の損傷の大きい後半のサブフレームを削ることもできる.静止画の撮影から動画撮影へと変わったことは,近原子分解能での解析へのキーステップであった.
こうしたハードウェアの進歩はわかりやすのだが,ほぼ時期を同じくして,非常にすぐれた解析ソフトウェアが開発されたことは同じく重要である.近原子分解能での解析の速度が飛躍的に改善されて報告が増加しているのも,このRelionというソフトウェアのおかげといってよい17).ベイズ統計をアルゴリズムに組み込み,粒子の構造が均一でないことを考慮に入れて画像を解析する.マニュアルも充実し,操作が非常に簡易で洗練されている.ここにきて,自動撮影によりたくさんの粒子画像を取得し,解析ソフトウェアとしてRelionを使うことにより均一な構造をした粒子のグループから高分解能で解析する,という新しいストラテジーができた.
2013年末のブレークスルーが,TRPV1という膜タンパク質の単粒子再構成だったことは意義深い.試料の調製において,界面活性剤としてamphipolという両親媒性のポリマーが使われ,これにより膜貫通領域も明瞭に可視化された.amphipolと膜タンパク質に関するだけのシンポジウムが開かれるほどこのようなくふうへの興味は深く,最近では,単粒子再構成法において溶液中の界面活性剤はじゃまなコントラストを形成するだけなので,これを取り除くGraDeR法という手法も報告された18).いずれにしても,試料の個性にあわせてくふうが必要なのは結晶化と変わらないだろう.
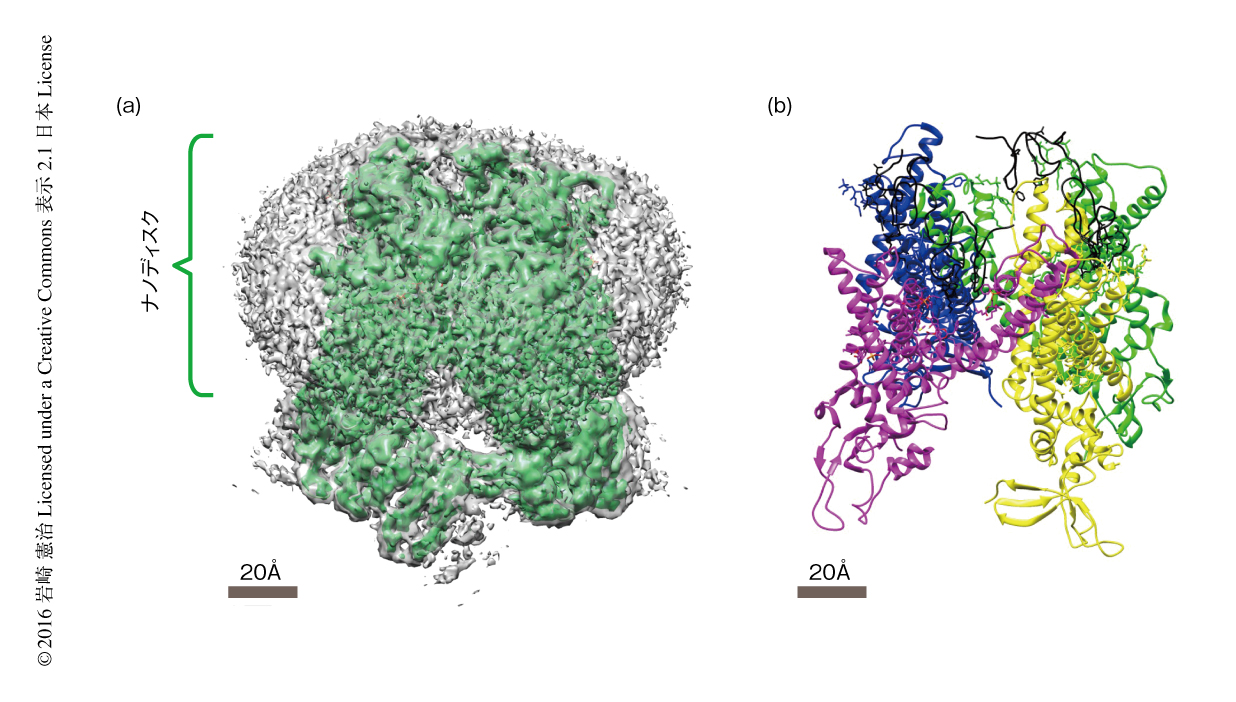
現在,成果をあげつつあるのはナノディスクである.これを使えば,膜という天然の状態にかぎりなく近い状態に再構成された膜タンパク質の構造をみることができる.しかし,膜の強いコントラストにより膜貫通部位が可視化できないのではないかという見方も多かった.2016年に入り,この懸念も払拭された19).TRPV1をナノディスクに再構成した試料における近原子分解能での解析である.再構成された密度図(EMDB ID:8117)から,原子モデル(PDB ID:5IRX)が構築された(図5).Tcトキシンをナノディスクに再構成した試料の解析における膜貫通部位の議論も報告された20).これらの応用は,膜タンパク質の構造研究に新たな展開をもたらすことはいうまでもないだろう.
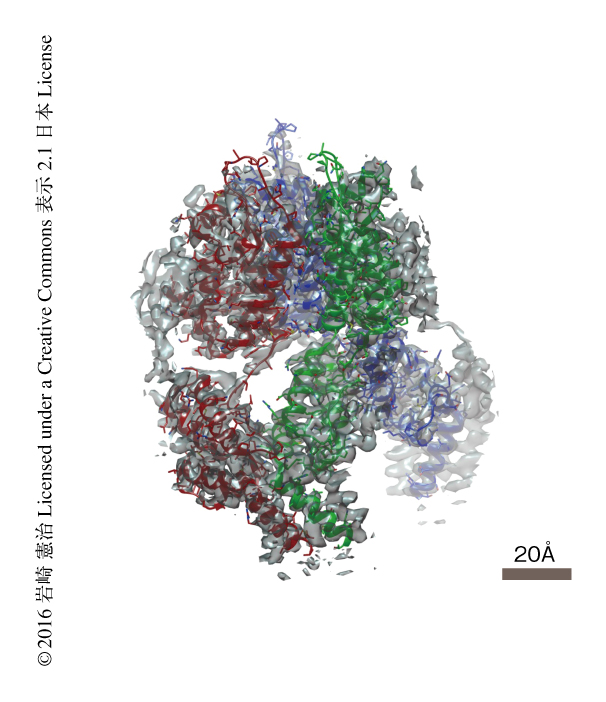
解析ソフトウェアRelionの登場により,ヘテロな構造(形状)をした粒子の構造解析の道が開けつつある.一方,単粒子再構成法の適応の困難な試料に対しても,クライオ電子線トモグラフィー法21) およびサブトモグラム平均法22) を用いた高分解能での解析への取り組みがつづけられており,2016年には,未成熟なHIVにおけるGagポリタンパク質のキャプシドドメインとスペーサーペプチド1について,ついに分解能3.9Åの構造が報告された23).サブトモグラム平均画像(EMDB ID:4015)から原子モデル(PDB ID:5L93)が得られている(図6).さきに述べたように,電子線トモグラフィー法は原理的には高分解能の情報を失う手法である.試料を傾斜させながら同一の領域の画像を取得し,そこから3次元の再構成像を逆投影などの手法によりコンピューターにおいて構築する.病院のX線CTと同じ原理である.試料を電子顕微鏡の内部で傾斜できる角度には限界があるので,データを欠損した領域が生じるが,このような原理的な問題を克服する手法として,同一の構造をしたものを同一の領域の画像から切り出し,それらの向きをあわせて平均化するサブトモグラム平均法が誕生した.しかし,いくらサブトモグラム平均化を用いたとしてもドメインの配置がわかる程度であり,原子モデルの構築が可能なレベルの密度図が得られるとは誰しも予想してはいなかっただろう.細胞まるごとは電子顕微鏡には分厚すぎて高分解能での解析はむずかしいが,再構成系など細胞に近い,しかし,シンプルで小さな系に応用することにより,ナノディスクに再構成した複合体などより生体に近い分子の状態を高分解能で可視化することが可能になるだろう.
クライオ電子顕微鏡による生体分子の近原子分解能での構造解析があいついでトップジャーナルに報告されており,その対象はより大きく複雑な複合体にまで広がり,さらなる可能性が示されている.解析ソフトウェアの扱いも容易になり,電子顕微鏡による画像さえあれば誰しも解析ができる時代になってきた.しかし,それだけに“あやまった構造”を報告しないようにすることは非常に重要である.筆者も,現在の近原子分解能での解析の時代よりまえ,明らかにまちがっていると思われる構造の報告例を何度か目にした.nmオーダーの分解能の構造だとその真偽の判定は非常にむずかしいが,現代,原子モデルが得られると,以前の報告があやまりだったことはすぐにバレてしまう.ランダムコニカルティルト法24) により初期の構造を時間をかけてもとめたり,あやしい構造についてはハイブリッドアプローチを試みたりするなど,多方面からの実験により確信が得られるまで報告を控えるような研究者自身の真摯な心がけが重要である.一方で,客観的なデータの検証システムをより習熟させていく必要がある.とくに,このことについてはデータベースの研究者を含め世界的な取り組みがなされている25).作業に没頭するだけでなく,原理をきちんと理解するこことは電子顕微鏡においても重要である.
略歴:1996年 大阪大学大学院基礎工学研究科博士課程 修了,松下電器中央研究所 研究員,1998年 米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases博士研究員,2001年 理化学研究所播磨研究所 連携研究員,2003年 大阪大学超高圧電子顕微鏡センター 産学官連携研究員を経て,2005年より大阪大学蛋白質研究所 准教授.
研究テーマ:電子顕微鏡を用いた構造生物学.
関心事:タンパク質のグローバルな構造の変化を,計算機シミュレーションとのハイブリッドアプローチによりどこまで解明できるか.
© 2016 岩崎 憲治 Licensed under CC 表示 2.1 日本
(大阪大学蛋白質研究所 附属蛋白質解析先端研究センター分子創製学研究室)
email:岩崎憲治
領域融合レビュー, 5, e010 (2016) DOI: 10.7875/leading.author.5.e010
Keiji Iwasaki: A new era for cryo-electron microscopy: near atomic resolution.
要 約
ここ数年,クライオ電子顕微鏡を用いた単粒子再構成法が構造生物学者にとり魅力的な手法のひとつとなってきた.結晶をつくることなく原子モデルを構築することのできるほど高分解能の3次元構造が得られるようになってきたからである.データベースへの登録数は加速度的に増加しており,大きな膜タンパク質の構造やスプライソソームのような複雑かつ多数の分子からなる複合体の構造も近原子分解能で解かれた.このようなブレークスルーをもたらしたものとして,コンピューターにより制御され高性能のステージを搭載した専用の電子顕微鏡の開発,電子直接検出器の登場,これまでになかったアルゴリズムを取り入れた新しい解析ソフトウェアの登場,の3つがあげられる.近原子分解能とよばれる高分解能の達成においては電子直接検出器および解析ソフトウェアが重要であり,高い生産性でデータが報告されるようになったのは専用の電子顕微鏡の登場によるところが大きい.透過型電子顕微鏡の誕生から80年以上をへて,ようやく電子顕微鏡を用いた構造生物学への道が開けてきた.この急激な進歩をもたらした技術について解説する.
はじめに
クライオ電子顕微鏡による生体分子の構造解析は,らせん対称性や2次元結晶を利用した方法により幕を開けた.しかし,クライオ電子顕微鏡に強くもとめられていたのは,結晶化や特殊な対称性を利用しない,より一般的な形状の分子が単分散した状態の画像から3次元構造をもとめる方法であった.歴史的には,そのような試みの最初は電子線トモグラフィーである.しかし,この手法は試料の同一の領域に電子線を照射しつづけるため,電子線による試料の損傷という電子顕微鏡による観察における最大の問題がネックとなり,いったんは姿を消した.つぎに,電子線による試料の損傷を克服する方法として単粒子再構成法が誕生した.そのよび方はさまざまで,日本語では単粒子解析法とよばれることがもっとも多く,英語ではとくに統一されていない.実際には多数の粒子を扱うので,日本語としては多粒子再構成法のほうが実際に近い.この手法により得られる構造は,正二十面体のキャプシドをもつウイルスのように高い対称性をもつ場合を除き,nmオーダーの分解能が一般であった.ところが,2013年の末に,(電子顕微鏡にとっては)小さな膜タンパク質であるTRPV1の原子モデルが報告されてから,近原子分解能での構造解析が爆発的に報告されるようになり,いまや,データベースへの登録数においてはNMR法をこえる勢いになってきている.このレビューにおいては,この劇的な進歩の背景,および,現在の単粒子再構成の実際について解説する1).
1.単粒子再構成法の歴史
精製した生体分子やその複合体(これを“電顕屋”は粒子とよぶ)が溶液中で単分散している場合,この状態を電子顕微鏡により撮影することができれば,1枚の画像にさまざまな方向をむいた粒子画像が写ることになる.精製して1種類の試料を単離したわけであるから,すべて同じものである.よって,同一の分子をいろいろな方向から撮影したのと同じ粒子画像がいちどに得られるわけである.電子顕微鏡は電子線の照射により試料を破壊するが,とくに軽元素からなる生体試料は損傷をうけやすい.しかし,これならば,たったいちどの照射により画像データを取得できるので,損傷を最小限に抑えることができる.場所を変えて撮影すれば,さらにさまざまな向きの粒子画像が得られる.単粒子再構成法の誕生は1975年とみてもよいだろう2).実際には,たったいちどの照射といえども試料を破壊する.高分解能で解析したければ,高分解能の情報が失われないよう,しかし,解析の可能なシグナルが得られるよう,ギリギリの量の電子線で撮影する(図1).そのため,シグナル/ノイズ比が極端に低い画像しか得られない.このことこそが,高分解能での解析を困難にしてきたおもな要因である.特殊な画像解析が必要になり,一般的な画像解析の技術はあまり適用できない.十分なシグナル/ノイズ比の構造情報を得るには粒子画像の平均化が必要になるが3),このため数千から数万枚の粒子画像,最近では数十万の粒子画像を扱うようになってきている.
クライオ電子顕微鏡においては非晶質層の氷に閉じ込めた試料を観察する.画像を撮影するため電子線を照射すると,そのとたんに試料が動く.その原因について結論は得られていないが,この問題の克服のため詳細な調査が行われている4).さらに,熱の流入による試料ステージの機械的な微動も大いに問題である.試料の破壊を最小限に抑えるため弱い電子線を照射する→露光時間が長くなる→ブレた像が記録される,という悪循環におちいりがちである.こうした撮影対象の微動が高分解能での解析をさまたげている第2の要因である.
また,精製したからといってタンパク質のコンフォメーションがすべて同じでないことはいうまでもない.それでも,Åオーダーの分解能で解析するためには,粒子がÅオーダーのレベルでそろった構造をもつ粒子画像をあつめる必要がある.別の考えとしては,“動いている”部分は分解能が悪いはずであるので,得られた分子構造の分解能を場所に依存して評価すればよいという考え方もある.
分子全体ではないが,一部は側鎖の密度が明瞭に観察されるなど,原子モデルを構築するのに十分な分解能をもつ密度図(電子顕微鏡の場合に得られるのは,“電子”密度図ではない)を近原子分解能とよぶ.結晶をつくらずに,あるいは,らせん対称性という特殊な構造を利用せずに,粒子画像として近原子分解能を達成できるとの理論が報告されたのは1995年のことであり,この報告は単粒子再構成法による原子分解能の実現への取り組みのよりどころとなった3).ここから,正二十面体のキャプシドをもつウイルスを対象にした高分解能での解析への取り組みがつづいた.正二十面体のキャプシドは安定しており,大きな複合体であるので弱い電子線でも十分なシグナル/ノイズ比が得られる.1998年,2つの研究室から,B型肝炎ウイルスのキャプシドにおいてαヘリックスの可視化に成功したという報告がなされた5,6).それから10年をへた2008年,正二十面体のウイルス粒子を使った近原子分解能での解析が報告された7,8).そして2010年,de novoの原子モデリングの報告がなされた9).さらに大きな転機は2013年末にやってきた.小さな対称性の低い膜タンパク質TRPV1の近原子分解能での構造が報告されたのである10,11).これを契機に,近原子分解能での報告は爆発的に増加した.2015年,βガラクトシダーゼの分解能2.2Åでの構造が報告され,ここにおいて,分子に配位した水分子が可視化された12).そして2016年,100 kDaよりも小さい分子について近原子分解能での解析が報告され,クライオ電子顕微鏡による構造解析にブレークスルーが起こったことが確かめられた13).さきに述べた1995年の予測において,原子分解能に達する最小の大きさは100 kDaとされており,この大きさにいどむことができたのである.呼吸鎖複合体Iなど巨大な膜タンパク質についてもあいついで近原子分解能での構造が報告されており,その勢いは加速の一途である.
2.ブレークスルーをもたらしたもの
論文にはおもてだってでてこないが,操作性の向上を実現した専用のクライオ電子顕微鏡の登場が近原子分解能の達成に貢献している事実は疑いようがない.新たなクライオ電子顕微鏡により,習熟したオペレーターのみが腕をかけて寝る間も惜しまず撮影するという生産性の低い状況はさまがわりした.以前からのもっともポピュラーなサイドエントリーという方式のクライオ電子顕微鏡では,2~3時間おきに手動で液体窒素を足す必要がある.よい試料にあたったときには,オペレーターが電子顕微鏡のまえに座り込み,液体窒素を足しながら撮りつづけるのである.さらに悪いことに,いちどに1個の試料しか観察できない.操作に習熟すればいったん観察した試料も回収できるが,その操作により大気中の水分が結晶状になって試料に混入するため必ず汚れてしまう.試料の出し入れに使用するホルダーは試料の交換のときに大気中に取り出すので霜だらけになり,完全に乾燥させるには何時間もかかる.現在のクライオ電子顕微鏡の作業のなかでもっとも重要かつ時間のかかるステップは,粒子が単分散した薄い非晶質層の氷の試料をつくることである.さまざまな条件で作製しつぎつぎに試さねばならない.しかし,この作業はサイドエントリーホルダーを使用していては非常に時間がかかる.このような効率の悪さは,米国FEI社(現 米国Thermo Fisher Scientific社)のTitan Kriosの登場により一変した(図2).2015年から2016年9月まで,データベースへ登録されたデータの取得に使われた電子顕微鏡としては圧倒的なシェアであった.電子顕微鏡はコンピューターにより制御されており,いちどに12個の試料を装填できる.試料の交換はマウスのクリックひとつででき,撮影したのちの凍結試料の回収も可能である.これができるとできないとでは,データ取得の速度に雲泥の差がある.液体窒素の自動供給装置がついているため,好きなときに撮影を中断することもできる.さらに,なんといっても自動撮影機構を備えていて,夕方にセットすれば翌朝には500枚近くの画像が取得されている.試料ステージが非常に安定しているので,機械的な微動はきわめて少ない.このハイエンドな電子顕微鏡には,高分解能での画像が多く必要な単粒子再構成法を強力に推進する機構が満載されている.
近原子分解能での解析の主役は,電子直接検出器といっても過言ではない.これまで,ファイバーカップルCCDカメラが記録装置として多く使われてきており,現在も,大部分の研究室はこれを使用している.高分解能での情報の記録媒体としてはフィルムのほうがすぐれているものの,フィルムより効率がよくコンピューターにより制御できるため主役の座を奪ってしまった.しかし,そのしくみから,画像がどうしてもボケてしまう.電子線をシンチレーターにより光子に変換し,これが光ファイバーにより伝達されCCD素子により検出される14)(図3).このプロセスにおいて像はボケてしまうのである.さらに,CCDにおける電荷転送のしくみから,どうしても読み出しの速度が遅くなる.そのため,動いている試料を1枚の写真として記録するので,おおげさにいえば,夜に車のヘッドライトを長い露光時間で撮影するようなものである.このような事情から,電子を直接に検出する装置が切望されていた.そこで,CMOS(complementary metal oxide semiconductor,相補型金属酸化膜半導体)を使った電子直接検出器が誕生したのである15).もちろん,光-電子変換がないのでボケが少ない.さらに,読み取りの速度が格段に速い.これにより,第2世代の電子直接検出器とよばれる,入射する電子の計数を可能にした電子直接検出器が開発された.記録装置が像にくわえるノイズはこれで一挙に減少した.
高速度での撮影が可能になったことにより新しい撮影法が生まれた.動画撮影法である4,16)(図4).試料の微動がさけられないなら,動画として撮影し,1コマ1コマ(サブフレームとよぶ)をアラインメントして足し合わせて1枚の画像にすればよい16).とくに,電子を試料に照射したとたんに起こる微動については,最初の1,2枚のサブフレームを使わなければよい.また,多くのサブフレームを撮影し,解析においては十分なシグナルをかせぐためすべてのサブフレームを足し合わせ,最後に電子線の損傷の大きい後半のサブフレームを削ることもできる.静止画の撮影から動画撮影へと変わったことは,近原子分解能での解析へのキーステップであった.
こうしたハードウェアの進歩はわかりやすのだが,ほぼ時期を同じくして,非常にすぐれた解析ソフトウェアが開発されたことは同じく重要である.近原子分解能での解析の速度が飛躍的に改善されて報告が増加しているのも,このRelionというソフトウェアのおかげといってよい17).ベイズ統計をアルゴリズムに組み込み,粒子の構造が均一でないことを考慮に入れて画像を解析する.マニュアルも充実し,操作が非常に簡易で洗練されている.ここにきて,自動撮影によりたくさんの粒子画像を取得し,解析ソフトウェアとしてRelionを使うことにより均一な構造をした粒子のグループから高分解能で解析する,という新しいストラテジーができた.
3.これからのクライオ電子顕微鏡による解析
2013年末のブレークスルーが,TRPV1という膜タンパク質の単粒子再構成だったことは意義深い.試料の調製において,界面活性剤としてamphipolという両親媒性のポリマーが使われ,これにより膜貫通領域も明瞭に可視化された.amphipolと膜タンパク質に関するだけのシンポジウムが開かれるほどこのようなくふうへの興味は深く,最近では,単粒子再構成法において溶液中の界面活性剤はじゃまなコントラストを形成するだけなので,これを取り除くGraDeR法という手法も報告された18).いずれにしても,試料の個性にあわせてくふうが必要なのは結晶化と変わらないだろう.
現在,成果をあげつつあるのはナノディスクである.これを使えば,膜という天然の状態にかぎりなく近い状態に再構成された膜タンパク質の構造をみることができる.しかし,膜の強いコントラストにより膜貫通部位が可視化できないのではないかという見方も多かった.2016年に入り,この懸念も払拭された19).TRPV1をナノディスクに再構成した試料における近原子分解能での解析である.再構成された密度図(EMDB ID:8117)から,原子モデル(PDB ID:5IRX)が構築された(図5).Tcトキシンをナノディスクに再構成した試料の解析における膜貫通部位の議論も報告された20).これらの応用は,膜タンパク質の構造研究に新たな展開をもたらすことはいうまでもないだろう.
解析ソフトウェアRelionの登場により,ヘテロな構造(形状)をした粒子の構造解析の道が開けつつある.一方,単粒子再構成法の適応の困難な試料に対しても,クライオ電子線トモグラフィー法21) およびサブトモグラム平均法22) を用いた高分解能での解析への取り組みがつづけられており,2016年には,未成熟なHIVにおけるGagポリタンパク質のキャプシドドメインとスペーサーペプチド1について,ついに分解能3.9Åの構造が報告された23).サブトモグラム平均画像(EMDB ID:4015)から原子モデル(PDB ID:5L93)が得られている(図6).さきに述べたように,電子線トモグラフィー法は原理的には高分解能の情報を失う手法である.試料を傾斜させながら同一の領域の画像を取得し,そこから3次元の再構成像を逆投影などの手法によりコンピューターにおいて構築する.病院のX線CTと同じ原理である.試料を電子顕微鏡の内部で傾斜できる角度には限界があるので,データを欠損した領域が生じるが,このような原理的な問題を克服する手法として,同一の構造をしたものを同一の領域の画像から切り出し,それらの向きをあわせて平均化するサブトモグラム平均法が誕生した.しかし,いくらサブトモグラム平均化を用いたとしてもドメインの配置がわかる程度であり,原子モデルの構築が可能なレベルの密度図が得られるとは誰しも予想してはいなかっただろう.細胞まるごとは電子顕微鏡には分厚すぎて高分解能での解析はむずかしいが,再構成系など細胞に近い,しかし,シンプルで小さな系に応用することにより,ナノディスクに再構成した複合体などより生体に近い分子の状態を高分解能で可視化することが可能になるだろう.
おわりに
クライオ電子顕微鏡による生体分子の近原子分解能での構造解析があいついでトップジャーナルに報告されており,その対象はより大きく複雑な複合体にまで広がり,さらなる可能性が示されている.解析ソフトウェアの扱いも容易になり,電子顕微鏡による画像さえあれば誰しも解析ができる時代になってきた.しかし,それだけに“あやまった構造”を報告しないようにすることは非常に重要である.筆者も,現在の近原子分解能での解析の時代よりまえ,明らかにまちがっていると思われる構造の報告例を何度か目にした.nmオーダーの分解能の構造だとその真偽の判定は非常にむずかしいが,現代,原子モデルが得られると,以前の報告があやまりだったことはすぐにバレてしまう.ランダムコニカルティルト法24) により初期の構造を時間をかけてもとめたり,あやしい構造についてはハイブリッドアプローチを試みたりするなど,多方面からの実験により確信が得られるまで報告を控えるような研究者自身の真摯な心がけが重要である.一方で,客観的なデータの検証システムをより習熟させていく必要がある.とくに,このことについてはデータベースの研究者を含め世界的な取り組みがなされている25).作業に没頭するだけでなく,原理をきちんと理解するこことは電子顕微鏡においても重要である.
文 献
- Subramaniam, S., Earl, L. A., Falconieri, V. et al.: Resolution advances in cryo-EM enable application to drug discovery. Curr. Opin. Struct. Biol., 41, 194-202 (2016)[PubMed]
- Frank, J.: Averaging of low exposure electron micrographs of non-periodic objects. Ultramicroscopy, 1, 159-162 (1975)[PubMed]
- Henderson, R.: The potential and limitations of neutrons, electrons and X-rays for atomic resolution microscopy of unstained biological molecules. Q. Rev. Biophys., 28, 171-193 (1995)[PubMed]
- Brilot, A. F., Chen, J. Z., Cheng, A. et al.: Beam-induced motion of vitrified specimen on holey carbon film. J. Struct. Biol., 177, 630-637 (2012)[PubMed]
- Conway, J. F., Cheng, N., Zlotnick, A. et al.: Visualization of a 4-helix bundle in the hepatitis B virus capsid by cryo-electron microscopy. Nature, 386, 91-94 (1997)[PubMed]
- Bottcher, B., Wynne, S. A. & Crowther, R. A.: Determination of the fold of the core protein of hepatitis B virus by electron cryomicroscopy. Nature, 386, 88-91 (1997)[PubMed]
- Zhang, X., Settembre, E., Xu, C. et al.: Near-atomic resolution using electron cryomicroscopy and single-particle reconstruction. Proc. Natl. Acad. Sci. USA, 105, 1867-1872 (2008)[PubMed]
- Yu, X., Jin, L. & Zhou, Z. H.: 3.88Å structure of cytoplasmic polyhedrosis virus by cryo-electron microscopy. Nature, 453, 415-419 (2008)[PubMed]
- Zhang, X., Jin, L., Fang, Q. et al.: 3.3Å cryo-EM structure of a nonenveloped virus reveals a priming mechanism for cell entry. Cell, 141, 472-482 (2010)[PubMed]
- Liao, M., Cao, E., Julius, D. et al.: Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature, 504, 107-112 (2013)[PubMed]
- Cao, E., Liao, M., Cheng, Y. et al.: TRPV1 structures in distinct conformations reveal activation mechanisms. Nature, 504, 113-118 (2013)[PubMed]
- Bartesaghi, A., Merk, A., Banerjee, S. et al.: 2.2Å resolution cryo-EM structure of β-galactosidase in complex with a cell-permeant inhibitor. Science, 348, 1147-1151 (2015)[PubMed]
- Merk, A., Bartesaghi, A., Banerjee, S. et al.: Breaking cryo-EM resolution barriers to facilitate drug discovery. Cell, 165, 1698-1707 (2016)[PubMed]
- 木村吉秀: CCDの原理と透過型電子顕微鏡. 顕微鏡, 45, 257-264 (2010)
- Mcmullan, G., Faruqi, A. R. & Henderson, R.: Direct electron detectors. Methods Enzymol., 579, 1-17 (2016)[PubMed]
- Li, X., Mooney, P., Zheng, S. et al.: Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM. Nat. Methods, 10, 584-590 (2013)[PubMed]
- Scheres, S. H.: RELION: implementation of a Bayesian approach to cryo-EM structure determination. J. Struct. Biol., 180, 519-530 (2012)[PubMed]
- Hauer, F., Gerle, C., Fischer, N. et al.: GraDeR: membrane protein complex preparation for single-particle cryo-EM. Structure, 23, 1769-1775 (2015)[PubMed]
- Gao, Y., Cao, E., Julius, D. et al.: TRPV1 structures in nanodiscs reveal mechanisms of ligand and lipid action. Nature, 534, 347-351 (2016)[PubMed]
- Gatsogiannis, C., Merino, F., Prumbaum, D. et al.: Membrane insertion of a Tc toxin in near-atomic detail. Nat. Struct. Mol. Biol., 23, 884-890 (2016)[PubMed]
- Frank, J. (ed.): Methods for Three-Dimensional Visualization of Structures in the Cell. Springer-Verlag, New York (2006)
- Iwasaki, K., Mitsuoka, K., Fujiyoshi, Y. et al.: Electron tomography reveals diverse conformations of integrin αIIbβ3 in the active state. J. Struct. Biol., 150, 259-267 (2005)[PubMed]
- Schur, F. K., Obr, M., Hagen, W. J. et al.: An atomic model of HIV-1 capsid-SP1 reveals structures regulating assembly and maturation. Science, 353, 506-508 (2016)[PubMed]
- Radermacher, M.: Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J. Electron Microsc. Tech., 9, 359-394 (1988)[PubMed]
- Sali, A., Berman, H. M., Schwede, T. et al.: Outcome of the first wwPDB Hybrid/Integrative Methods Task Force Workshop. Structure, 23, 1156-1167 (2015)[PubMed]
著者プロフィール
略歴:1996年 大阪大学大学院基礎工学研究科博士課程 修了,松下電器中央研究所 研究員,1998年 米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases博士研究員,2001年 理化学研究所播磨研究所 連携研究員,2003年 大阪大学超高圧電子顕微鏡センター 産学官連携研究員を経て,2005年より大阪大学蛋白質研究所 准教授.
研究テーマ:電子顕微鏡を用いた構造生物学.
関心事:タンパク質のグローバルな構造の変化を,計算機シミュレーションとのハイブリッドアプローチによりどこまで解明できるか.
© 2016 岩崎 憲治 Licensed under CC 表示 2.1 日本