piRNAの生成機構および作用機序
2018/05/07
泉 奈津子・泊 幸秀
(東京大学定量生命科学研究所 RNA機能研究分野)
email:泊 幸秀
領域融合レビュー, 7, e003 (2018) DOI: 10.7875/leading.author.7.e003
Natsuko Izumi & Yukihide Tomari: The biogenesis and function of PIWI-interacting RNAs.

RNAサイレンシング機構のひとつとして知られるpiRNA経路は,動物の生殖巣において,おもにトランスポゾンの発現の抑制に機能する.piRNAは約30塩基の小分子RNAで,配列に相補性のあるトランスポゾンRNAへとPIWIタンパク質をガイドすることにより,転写レベルおよび転写後レベルにおいてその発現を抑制する.piRNA経路にかかわる因子の欠損はトランスポゾンの活性化や配偶子の形成不全をひき起こすことから,piRNA経路は生殖細胞のゲノムの完全性を維持するうえできわめて重要な役割を担うと考えられている.これまでの精力的な研究により,多数の因子が関与する複雑なpiRNA経路の全体像がしだいに明らかにされてきた.自らのゲノムに保持されるトランスポゾンの情報をもとに,piRNAはどのように生成され機能するのか,その分子機構について最近の知見をまじえて解説する.
ヒトのゲノムの約45%は,トランスポゾンとよばれる転移因子やその残骸からなるくり返し配列によりしめられることが明らかにされている.トランスポゾンは,自らの配列を切り出し別の部位に転移するDNA型トランスポゾンと,自らの配列からの転写産物をDNAへと逆転写し,新たな部位へと挿入することにより増幅するレトロトランスポゾンとに分類される.これらトランスポゾンの活性は,遺伝子の多様性の獲得やゲノムにダイナミックな変化をもたらすことにより生物の進化に寄与してきたと考えられている.その一方で,トランスポゾンの転移は遺伝子の発現を撹乱したり遺伝子の変異をひき起こしたりするため,正確な遺伝情報の維持のため,その活性は適切に制御されなければならない.通常,トランスポゾンの領域はDNAメチル化や抑制性のヒストン修飾により転写が抑制されているが,動物の生殖細胞には,さらにRNAサイレンシング機構によりトランスポゾンを抑制するしくみとしてpiRNA経路が存在する.その中心となるのが,piRNA(PIWI-interacting RNA)およびそのパートナーであるPIWIタンパク質(PIWI)である.piRNAとPIWIは複合体を形成し,主として標的となるトランスポゾンの発現を転写レベルおよび転写後レベルで抑制する.これによりトランスポゾンの脅威から生殖細胞のゲノムを保護し,次世代への正確な遺伝情報の継承を保障する.また,piRNA経路により抑制されるべきトランスポゾンなどの非自己に由来する配列の一部は,生殖細胞を介して次世代へとうけつがれることも明らかにされている.
このレビューにおいては,これまでの研究により明らかにされてきたショウジョウバエおよびマウスにおけるpiRNAの生成機構および作用機序を,最近の知見をまじえて解説する.また,ショウジョウバエやマウスとは異なる方法で非自己に由来する配列を識別し抑制する機構を獲得してきた線虫のpiRNA経路についても言及する.
なお,RNAサイレンシングの構造基盤については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001 (2012) も参照されたい.
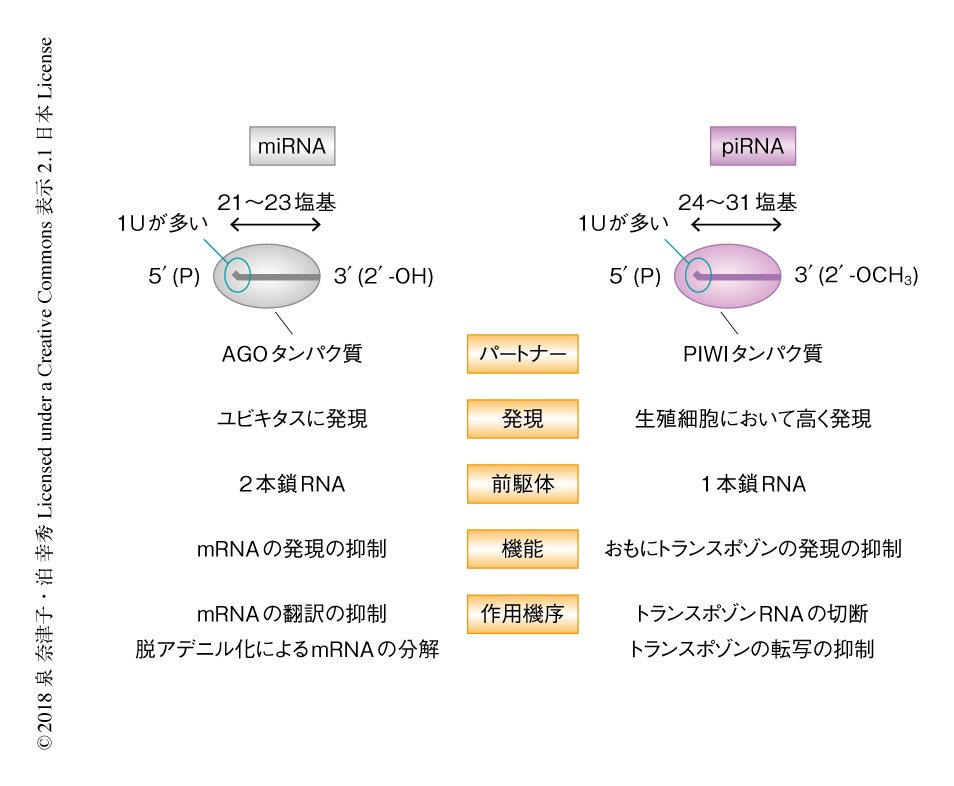
piRNAは動物の生殖細胞に高く発現する24~31塩基の小分子RNAであり,Argonaute(AGO)ファミリーに属するPIWIタンパク質と複合体を形成して機能する(図1).piRNAの配列は多様で,その配列に種間での保存性はみられないが,1塩基目がウリジンのものが多く(1Uバイアス),3'末端のリボースは2'-O-メチル化修飾されているという特徴がある.piRNAの1Uバイアスは,AGOと同様に,piRNAの1塩基目を認識するPIWIのMIDドメインとの結合の安定性により説明される1,2)(文献1) は新着論文レビュー でも掲載,文献2) は新着論文レビュー でも掲載).同様に,piRNAの3'末端を認識するPIWIのPAZドメインは,AGOのPAZドメインに比べ,2'-O-メチル化修飾をもつ塩基とより安定に結合しやすい構造をしており3,4),piRNAの3'末端はこの2'-O-メチル化修飾によりヌクレアーゼによる分解から保護されると考えられている.
piRNAはトランスポゾンに対し相補的な配列をもつものが多く,piRNA-PIWI複合体の主要な役割はトランスポゾンの発現の抑制にある.PIWIを欠損した変異体やpiRNAを生成できない変異体においては,トランスポゾンの脱抑制とともに配偶子の形成が損なわれ不妊になる.これらのことから,piRNA経路はトランスポゾンの活性を抑制することにより生殖細胞のゲノムをまもる役割があると考えられている.
PIWIはAGOと同様にスライサー活性をもち,piRNAと結合したPIWIは,piRNAと相補的な配列をもつトランスポゾンRNAを認識すると,piRNAの10塩基目と11塩基目とのあいだで標的RNAを切断する.これにより,トランスポゾンRNAの転写後の抑制に機能する.piRNAの長さはsiRNAと比較するとやや長いが,PIWIによる標的RNAの切断には2~21塩基目までの相補性で十分であることが示されている5).また,のちに述べるように,PIWIによる標的RNAの切断は,標的RNAの発現を抑制するだけでなく新たなpiRNAの生成とも共役している.一方,ショウジョウバエのPiwiやマウスのMIWI2など核に局在する一部のPIWIは,スライサー活性に非依存的に標的となるトランスポゾンの転写の抑制に機能する6-8).
piRNAはおもにpiRNAクラスターからの転写産物やトランスポゾンに由来するRNAから生成される.piRNAクラスターとは機能を失ったトランスポゾンの残骸が蓄積したゲノム領域のことで,長いものでは数百kbpにおよぶものもある9).また,piRNAの一部はmRNAのおもに3'側非翻訳領域からも生成されており,これらはgenic piRNAとよばれている10).
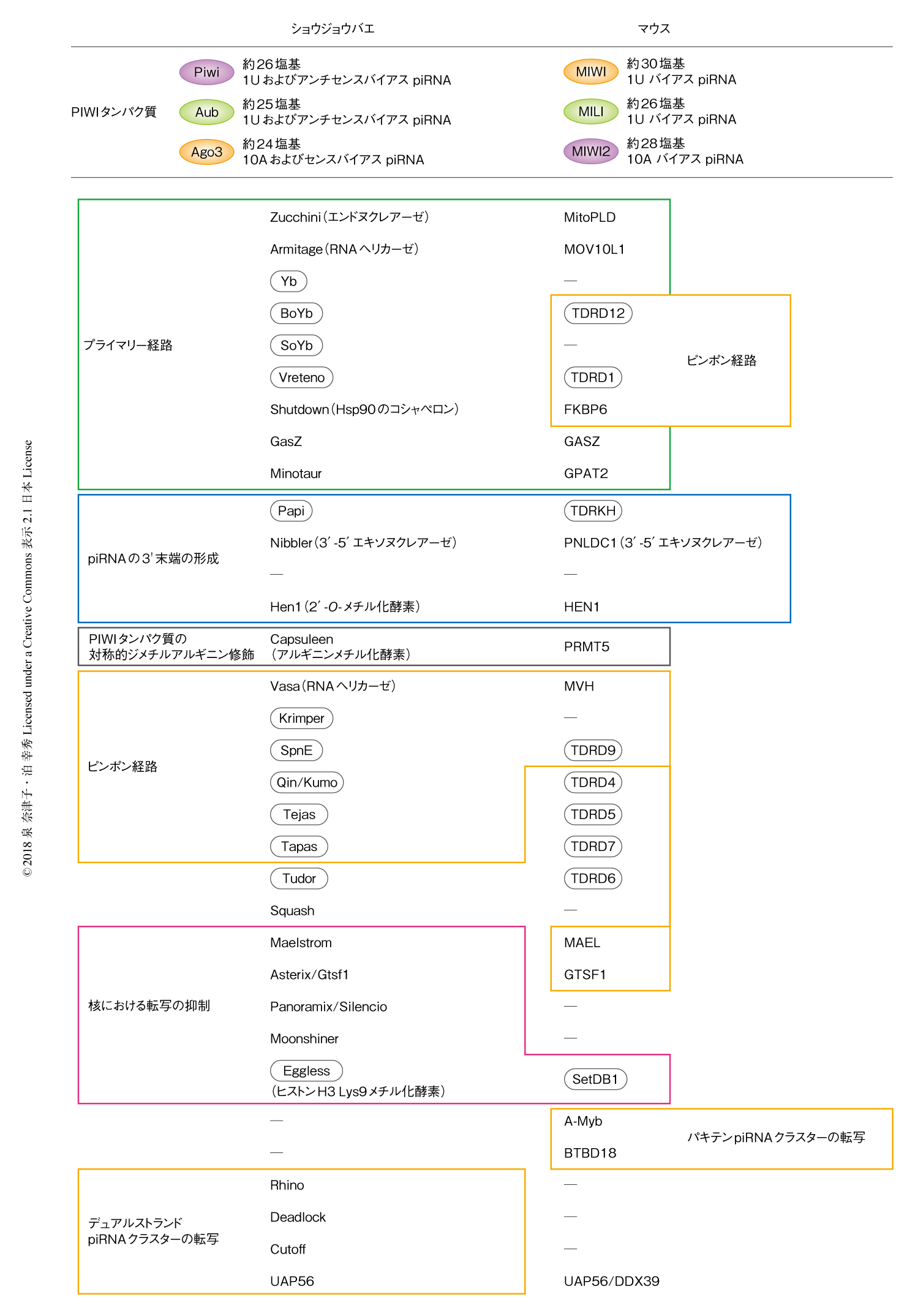
miRNAやsiRNAとは異なり,piRNAは中間体として2本鎖の状態を介さず,長い1本鎖の転写産物が細胞質においてプロセシングされることにより生成される.その過程には,多数のTudorドメインタンパク質,RNAヘリカーゼ,ヌクレアーゼが必要である11)(図2).PIWIのおもにN末端領域のアルギニン残基は,Capsuleen/PRMT5により対称的ジメチルアルギニン修飾されることが知られている12).Tudorドメインタンパク質は,TudorドメインによりPIWIの対称的ジメチルアルギニン修飾を認識して結合する.Tudorドメインタンパク質とPIWIとの相互作用は,piRNAの生成およびPIWIの局在に重要であることが示されている.
piRNAの生成経路は,既存のpiRNAを必要としないプライマリー経路と,PIWIのスライサー活性に依存したピンポン経路とに大別される.ピンポン経路においては,PIWIによる標的RNAの切断と共役して新たなpiRNAが生成される.以下,ショウジョウバエ,マウス,線虫それぞれのpiRNA経路について解説する.
ショウジョウバエにはPiwi,Aubergine(Aub),Argonaute3(Ago3)の3つのPIWIタンパク質が存在し,そのいずれの欠損でもトランスポゾンの脱抑制や配偶子の形成の異常がみられ,雌雄とも不妊になる.piRNAの研究はショウジョウバエの卵巣においてもっとも進んでおり,以降は,ショウジョウバエの卵巣における解析から明らかにされたことについて述べる.
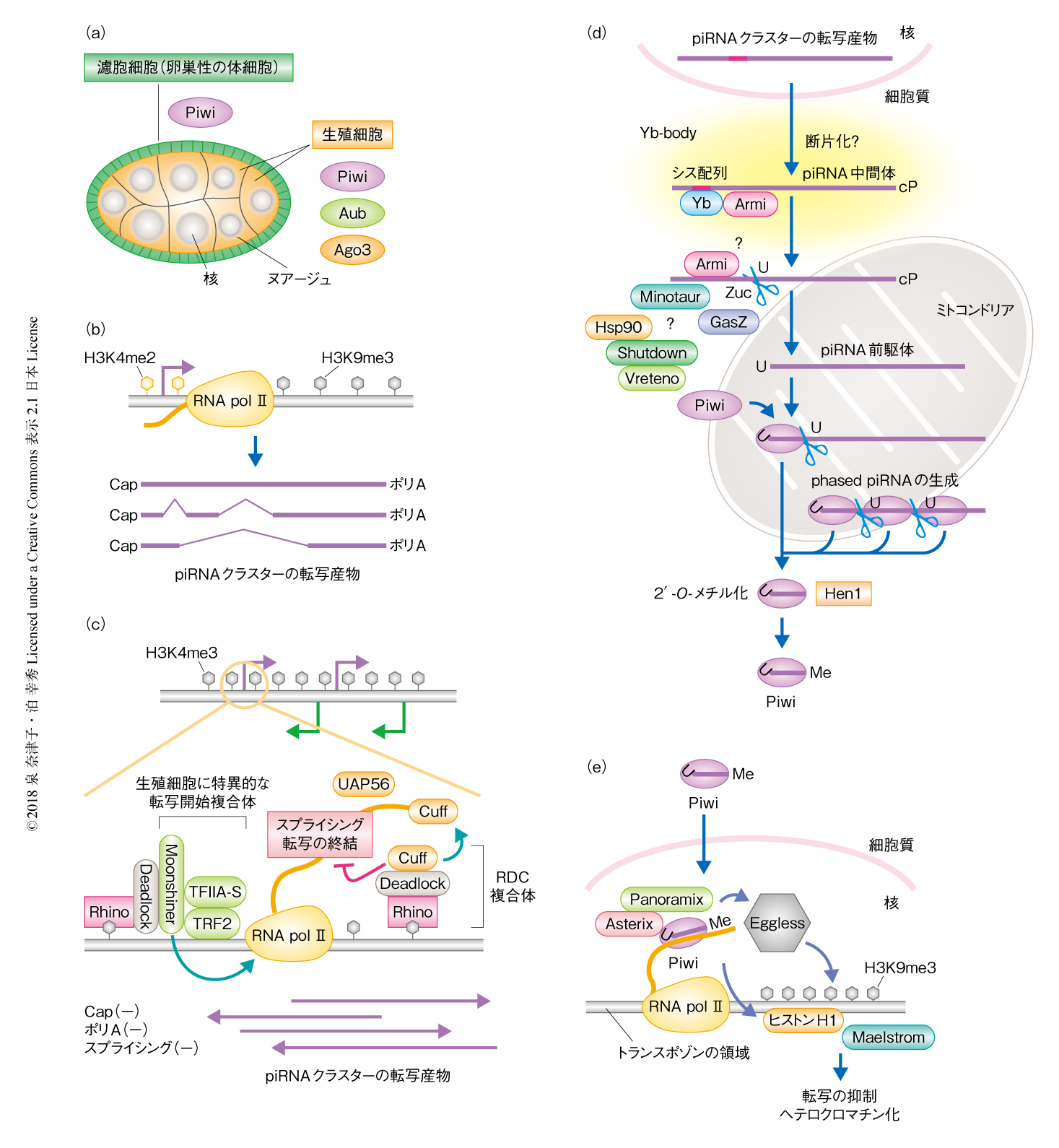
ショウジョウバエの卵巣を構成する卵室は,生殖細胞である哺育細胞および卵母細胞が卵巣性の体細胞である濾胞細胞にかこまれた構造をとる(図3a).3つのPIWIのうち,Piwiは生殖細胞および濾胞細胞の両方に発現するが,AubおよびAgo3は生殖細胞のみに発現する9).また,Piwiは核に局在し転写の抑制に機能するのに対し,AubおよびAgo3は細胞質の核の周囲の顆粒構造“ヌアージュ”(フランス語で“雲”の意)に局在し,標的RNAを切断することにより転写後の抑制に機能する9).さきに述べたように,piRNAの生成経路はプライマリー経路とピンポン経路とに大別されるが,濾胞細胞においてはプライマリー経路のみが,生殖細胞においてはプライマリー経路およびピンポン経路が機能する.
piRNAクラスターおよびpiRNAクラスターからの転写:ショウジョウバエのpiRNAクラスターはセントロメアやテロメアの近傍に多く,転写の様式から,ゲノムの片方の鎖から転写産物を生成するflamencoなどのユニストランドクラスター(図3b)と,ゲノムの両方の鎖から転写産物を生成する42ABなどのデュアルストランドクラスター(図3c)とに分類される.濾胞細胞において発現するpiRNAはユニストランドクラスターに由来するものが多く,その転写産物はトランスポゾンに対しアンチセンス方向のトランスポゾンの断片を多く含むことが知られている.一方,生殖細胞において発現するpiRNAはデュアルストランドクラスターに由来するものが多い.いずれのpiRNAクラスターにも,ヘテロクロマチン化のシグナルであるヒストンH3のLys9のトリメチル化修飾がみられ,piRNAクラスターからのpiRNAの生成にはヒストンH3のLys9のメチル化酵素が必要である13).ヒストンH3のLys9のトリメチル化修飾は抑制性のヒストン修飾として知られるが,デュアルストランドクラスターにおいてはその転写に不可欠である.デュアルストランドクラスターは明確なプロモーター領域をもたず,その転写はヒストンH3のLys9のトリメチル化修飾と結合するヘテロクロマチンタンパク質HP1のホモログであるRhinoに依存する14).Rhinoは,生殖細胞に特異的に発現する基本転写因子のサブユニットのホモログであるMoonshinerをリクルートすることにより転写開始複合体の形成をひき起こし,piRNAクラスターの内部からの転写を活性化する15,16)(図3c).デュアルストランドクラスターからは,キャップ構造をもたずスプライシングをうけない特殊な転写産物がRNAポリメラーゼIIにより生成される16,17).RhinoはさらにDeadlockを介してCutoff(Cuff)とRDC複合体を形成する16).Cuffは,piRNAクラスターからの転写産物の5'末端と結合することにより転写産物を安定化するとともに,RNAのプロセシングや転写の終結を抑制し,デュアルストランドクラスターからの転写を促進すると考えられている17,18).デュアルストランドクラスターからの転写産物には,核においてUAP56を含むTREX複合体がCuffに依存的に結合し,細胞質のヌアージュへと輸送される19,20).一方,ユニストランドクラスターからの転写はヒストンH3のLys4のジメチル化修飾をもつ推定上のプロモーター領域からRhinoに非依存的に開始される16).その転写産物はmRNAと同様にキャップ構造およびポリA鎖をもち,選択的スプライシングをうけうることから,デュアルストランドクラスターとは転写の機構が明確に区別される16)(図3b).
プライマリー経路におけるpiRNAの生成:プライマリー経路は濾胞細胞および生殖細胞の両方において機能しており,Piwiと結合するpiRNAおよび一部のAubと結合するpiRNAを生成する.以下,濾胞細胞においてプライマリー経路によりPiwiと結合するpiRNAが生成される過程について述べる.piRNAクラスターからの転写産物がプライマリー経路に入るには,転写産物のもつシス配列の関与が示唆されている.たとえば,濾胞細胞における主要なpiRNAクラスターであるflamencoからの転写産物には,5'末端に下流からのpiRNAの生成をひき起こすことのできるpiRNAトリガー配列(piRNA-trigger sequence:PTS)が同定されている21).また,濾胞細胞においてgenic piRNAを生成する主要なmRNAであるtraffic jamの3'側非翻訳領域にも,下流からのpiRNAの生成をひき起こす約100塩基のシス配列が同定されており,このシス配列にTudorドメインタンパク質であるYbが結合することによりpiRNAの生成がひき起こされる22).さらに,濾胞細胞において任意の転写産物にYbやArmiを結合させることによっても,下流の領域からpiRNAの生成がひき起こされる23,24).これらのことから,濾胞細胞においては,YbがpiRNAクラスターからの転写産物をプライマリーpiRNA経路にみちびくと考えられている(図3d).piRNAクラスターからの長い転写産物は,細胞質においてなんらかのヌクレアーゼによりプロセシングされ,5'末端にリン酸基がなく3'末端が2',3'-環状リン酸となったpiRNA中間体になると予想されている8).ArmiはATP依存性のRNAヘリカーゼで,Ybに依存的にpiRNA中間体と結合する8).Armiと結合したpiRNA中間体は,ミトコンドリア外膜に存在するエンドヌクレアーゼZucchini(Zuc)へとリクルートされ切断される25,26)(文献25) は新着論文レビュー でも掲載).バイオインフォマティクス解析により,ZucによるpiRNA中間体の切断はウリジンのまえで起こりやすいと考えられているが,精製したZucのエンドヌクレアーゼ活性はウリジンへの嗜好性を示さないことから,なんらかの補因子の存在が想定されている25-28).Zucによる切断により生じた1Uバイアスのある3'側のRNA断片はpiRNA前駆体としてPiwiに取り込まれる.PIWIへのpiRNA前駆体の取り込みには,Hsp90およびコシャペロンであるShutdownの関与が示唆されている29,30).Piwiに取り込まれたpiRNA前駆体は3'側がふたたびZucにより切断され27,28),Hen1による2'-O-メチル化修飾をうけて成熟したpiRNAになる31,32).ZucによるpiRNAの3'末端の形成は,同時に下流に新たなpiRNA前駆体を生じ,3'方向へと連続的にPiwiと結合したphased piRNAを生成する27,28).このようにして,ひとつの転写産物から多様な配列をもつ多くのpiRNAが生成される.プライマリー経路におけるpiRNAの生成には,ほかにもTudorドメインタンパク質であるVreteno 33),ミトコンドリア外膜タンパク質であるGasZやMinotaurが必要であることが示されており34-36),GasZはArmiの局在の制御にかかわることが示唆されている35,36).濾胞細胞において,Ybは核膜の近傍のYb-bodyとよばれる構造体に局在する8)(図3d).Yb-bodyにはYbやArmiをはじめ,プライマリー経路に必要なVretenoやShutdownなどが局在し,ミトコンドリアの近傍に観察されることから,Yb-bodyは濾胞細胞におけるpiRNAの生成の場であると考えられている.一方,生殖細胞にYbは発現しておらず,YbのホモログであるBrother of YbやSister of Ybがその機能を代替することが示唆されているが33),Yb-bodyに相当する構造体はなく,ヌアージュがpiRNAの生成の場であると考えられている.
Piwiと結合したpiRNAの核における機能:PiwiはpiRNAと結合すると核移行シグナルに依存して核へと移行し,piRNAの配列に相補的なトランスポゾンRNAと結合する.さらに,PiwiはAsterix/Gtsf1やPanoramix/Silencioと複合体を形成し,ヒストンH3のLys9のメチル化酵素であるEgglessをリクルートすることによりトランスポゾンの領域においてヒストンH3のLys9のトリメチル化をひき起こす37-41)(図3e).Piwiは,ヒストンH3のLys9のトリメチル化修飾と並行して,リンカーヒストンH1を介したクロマチン構造の変化をひき起こすことによっても標的となる領域のヘテロクロマチン化を促進し,トランスポゾンの発現を転写レベルで抑制する42)(新着論文レビュー でも掲載).Piwiによるトランスポゾンの転写の抑制にはMaelstromも必要であるが,MaelstromはヒストンH3のLys9のトリメチル化には関与せず,その作用点は明らかにされていない7).最近,Piwi,Asterix,Panoramix,Maelstromなど,Piwiとともに核において転写の抑制に機能するpiRNA因子の消失により,トランスポゾンmRNAのスプライシングのパターンが変化することが明らかにされた43).くわしい分子機構は不明であるが,PIWIやこれらのpiRNA因子はpiRNAを介してクロマチンの状態を変化させることにより,トランスポゾンmRNAのスプライシングを負に制御してトランスポゾンの活性を抑制するようである.
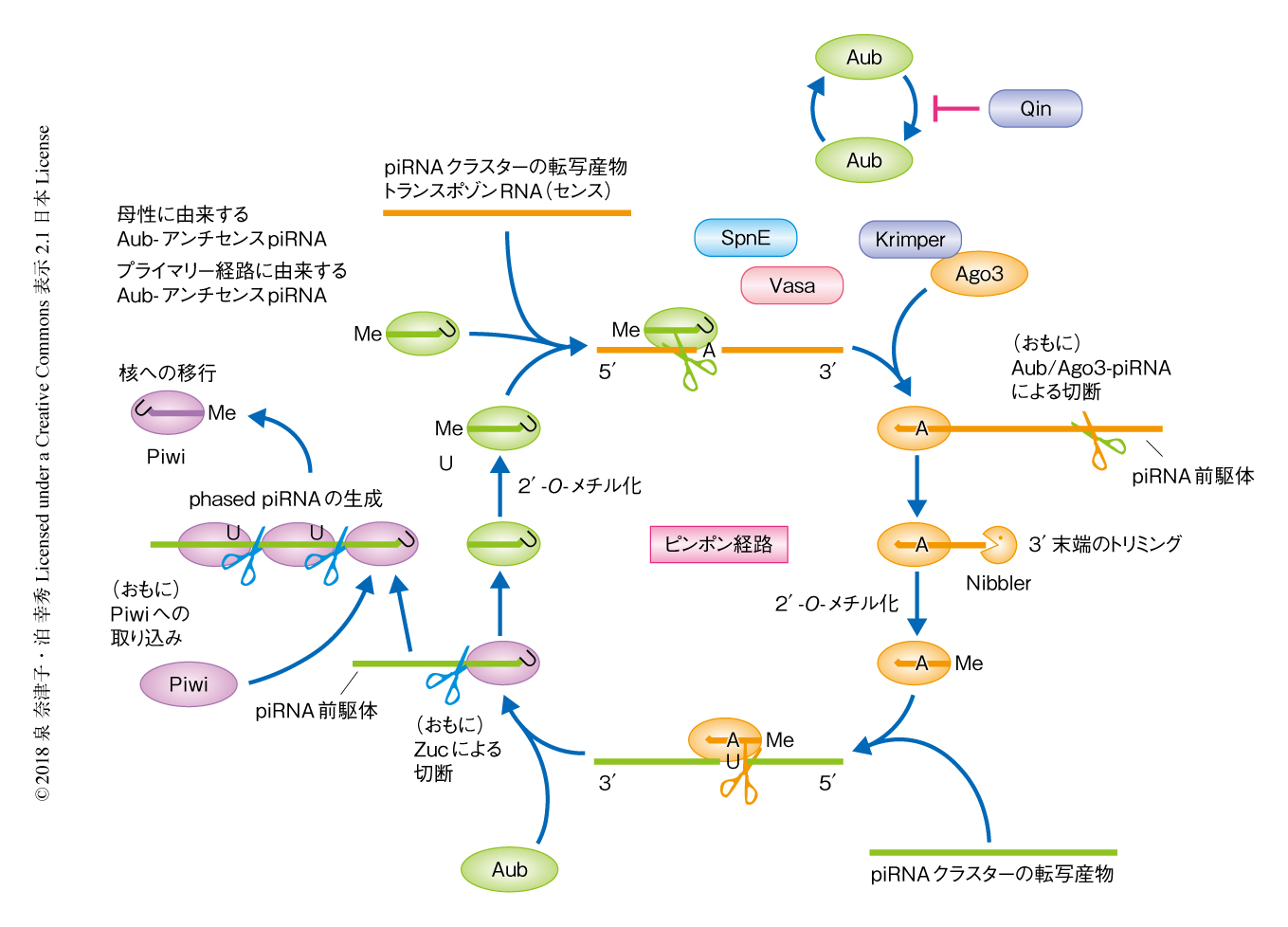
ピンポン経路におけるpiRNAの生成および転写後の抑制:ピンポン経路によるpiRNAの生成機構は,Aubと結合するpiRNAとAgo3と結合するpiRNAの5'側の10塩基が互いに相補的であるものが多いという事実から提唱された9,44).ピンポン経路においては,まず,母性に由来するあるいはプライマリー経路において生成されたAubと結合するアンチセンスpiRNAがそのトリガーとなり,相補的な配列をもつpiRNAクラスターからの転写産物やトランスポゾンRNAを切断する.これにより生じたセンス方向の3'側のRNA断片はpiRNA前駆体としてAgo3に取り込まれる(図4).このとき,KrimperはpiRNAと結合していないAgo3をヌアージュへとリクルートし,Ago3と結合したセンスpiRNAの生成に機能する45,46)(文献45) は新着論文レビュー でも掲載).一方,Qin/Kumoは,Aubによる切断により生じたRNA断片がAubやPiwiに取り込まれるのを抑制することにより,AubとAgo3とのあいだのピンポン反応を促進する47).また,Aubによる切断により生じたRNA断片がAubからAgo3へとうけわたされる過程には,VasaのATPase活性が必要であることが示唆されている48,49).これらピンポン経路におけるpiRNAの生成に必要な因子も,AubやAgo3と同様にヌアージュに局在することが知られている.ショウジョウバエにおけるpiRNAの3'末端の形成機構には,Zucによる切断,および,PIWIによる切断とそれにつづく3'-5'エキソヌクレアーゼNibblerによるトリミング,という2つの様式が知られている50).Ago3に取り込まれたpiRNA前駆体は,下流の領域においてPIWIにより切断されNibblerによりトリミングされる場合が多い50)(図4).一方,Aubに取り込まれたpiRNA前駆体は,多くの場合,Zucによる切断により直接的に3'末端が形成される50).いずれの場合も,piRNAの3'末端はさらにHen1により2'-O-メチル化修飾をうけて成熟したpiRNAになる.ZucによるpiRNA前駆体の切断は,プライマリー経路と同様,3'末端の形成と同時に新たなpiRNA前駆体の5'末端を生じる21,27,28)(図4).これらのphased piRNAはおもにPiwiに取り込まれ,核に移行して転写の抑制に機能する.phased piRNAの生成により,配列が多様化したトランスポゾンにも対応したpiRNAが生成される.このように,ピンポン経路においては,標的となるトランスポゾンRNAの切断による発現の抑制と共役して,活性化したトランスポゾンの抑制にはたらくアンチセンスpiRNAが増幅される.また,ピンポン経路により生成されたAub結合piRNAとAgo3結合piRNAは互いに5'側の10塩基が相補的であり,逆のストランドバイアスを示す.ピンポン経路が提唱された当初,Ago3結合piRNAの10Aバイアスは,1UバイアスのあるAub結合piRNAが標的RNAを切断したことにより生じたものと考えられていた.しかし,のちの解析により,piRNAの1塩基目はPIWIのMIDドメインの5'結合ポケットにより認識されており,標的RNAとの塩基の対合には寄与しないこと1),また,Aubは標的RNAを認識し切断する際,piRNAと対合する標的RNAの1塩基目がAであるもの(t1A)を好む傾向のあることが示された51).これらのことから,現在では,Ago3結合piRNAの10AバイアスはAub結合piRNAの1Uバイアスに起因するのではなく,Aubのt1A嗜好性によるものだと考えられている.一方,Ago3はAubのような標的RNAの1塩基目の嗜好性をもたないことが示唆されている.
piRNA結合Aubは,後期の卵細胞においてTudorやCapsuleenなどとともに,次世代の生殖細胞を形成する卵細胞の後極の極顆粒に局在する.さきに述べたように,このAub結合piRNAは抑制すべきトランスポゾンの配列の記憶として母親からうけつがれ,次世代の生殖細胞におけるpiRNAの生成のトリガーとしてはたらく52).母親にないトランスポゾンを父親が保持していた場合,交配により生じた個体はそのトランスポゾンに対するpiRNAを生成できず不妊の表現型を呈する.この現象はhybrid dysgenesisとよばれている.
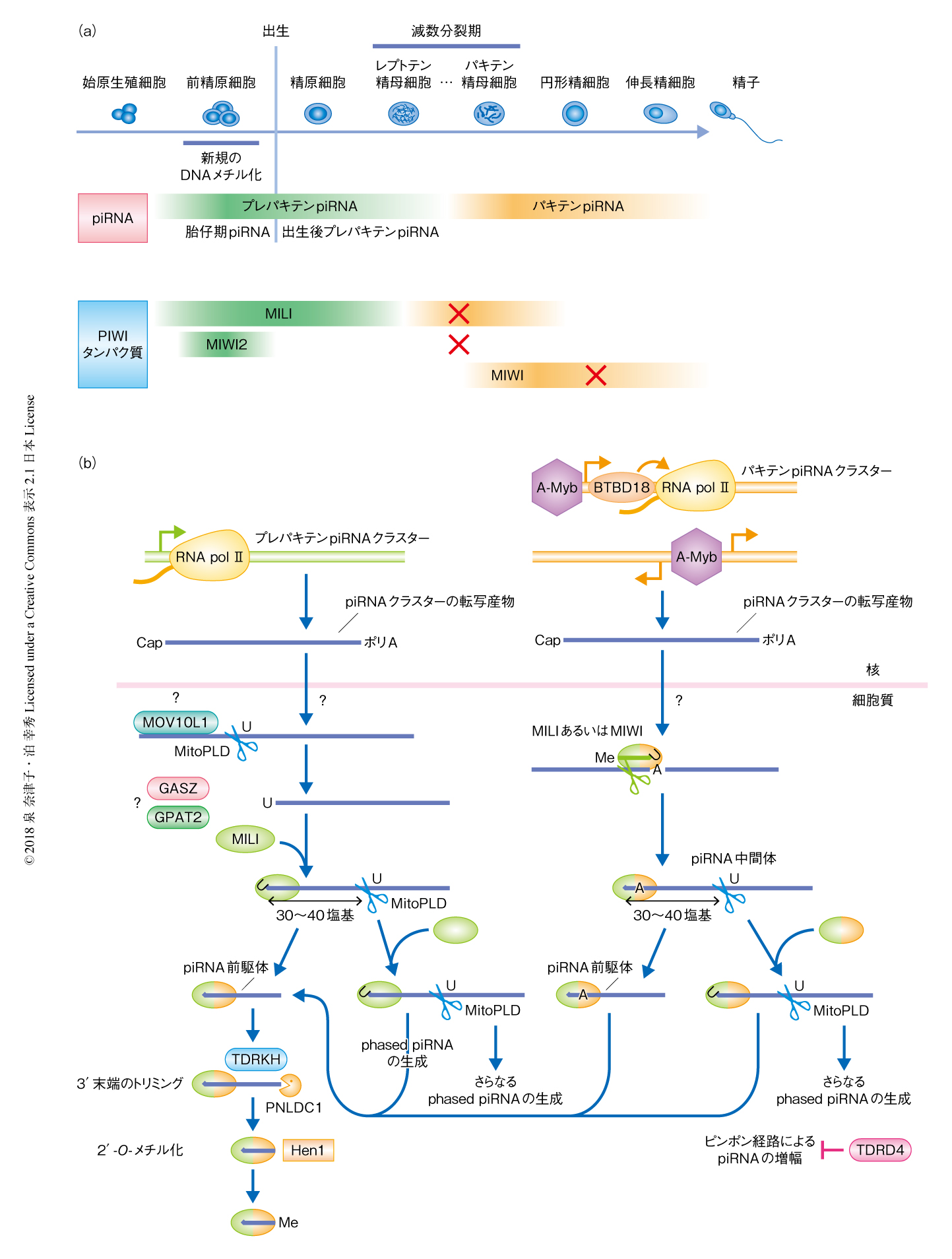
マウスにおいてPIWIおよびpiRNAは精巣に高く発現しており,正常な精子の形成に不可欠である.一方,卵巣におけるPIWIの発現は低く,その欠損による明らかな影響は報告されていない.マウスにおいてはMILI,MIWI2,MIWIという3つのPIWIタンパク質が精巣の発達にともない時期特異的に発現する(図5a).MILIは胎仔期から円形精子期にわたり発現するが53),MIWI2は出生の前後のごくかぎられた時期にのみ発現する54).この時期は,前精原細胞の分裂が一時的に停止し新規のDNAメチル化が起こる時期にあたる.一方,MIWIは生後の精子が形成される時期である減数分裂パキテン期から伸長精子期にかけて発現する55).MILIあるいはMIWI2のノックアウトマウスは減数分裂パキテン期のまえに精子の形成が停止し不妊になる53,56).同様な表現型は,piRNAの生成にかかわるMVH,MitoPLD,TDRKH,TDRD9,TDRD12,GASZ,MOV10L1などのノックアウトマウスにおいても観察されている57-64).一方,MIWIの欠損では,減数分裂は正常に進行するものの,円形精子のアポトーシスにより精子の形成が損なわれ不妊になる55).同様に,TDRD1,TDRD5,TDRD7,RNF17などのpiRNA因子のノックアウトマウスにおいても減数分裂ののち精子の形成は不全になることが知られている65-68).
マウスのpiRNAは,その発現の時期によりプレパキテンpiRNAとパキテンpiRNAにと大別される(図5a).プレパキテンpiRNAはMILIおよびMIWI2と結合しており,パキテンpiRNAはMILIおよびMIWIと結合する.また,MILI,MIWI2,MIWIそれぞれに結合するpiRNAの長さのピークは,26塩基,28塩基,30塩基と多少の違いのあることが知られている(図2).
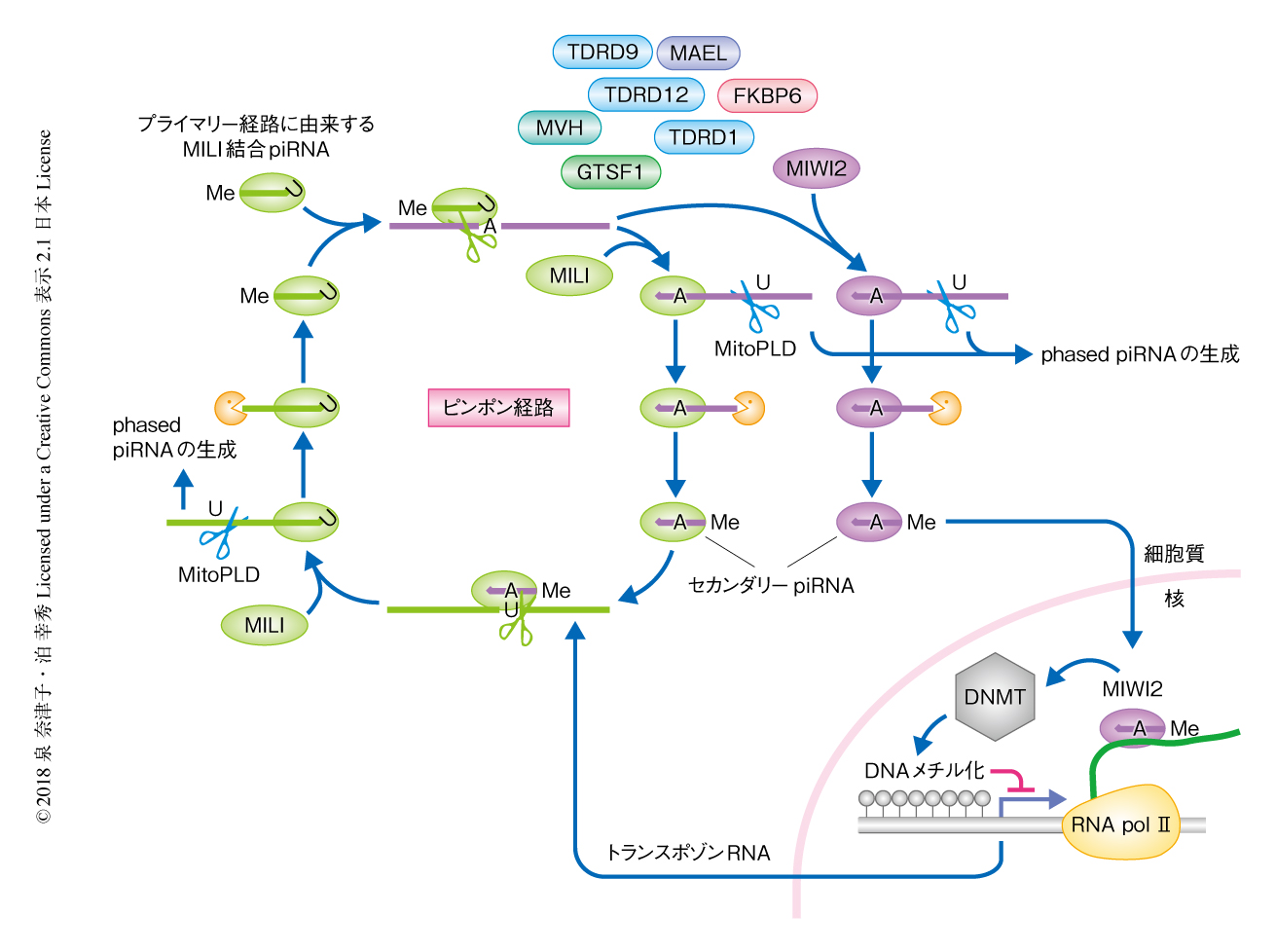
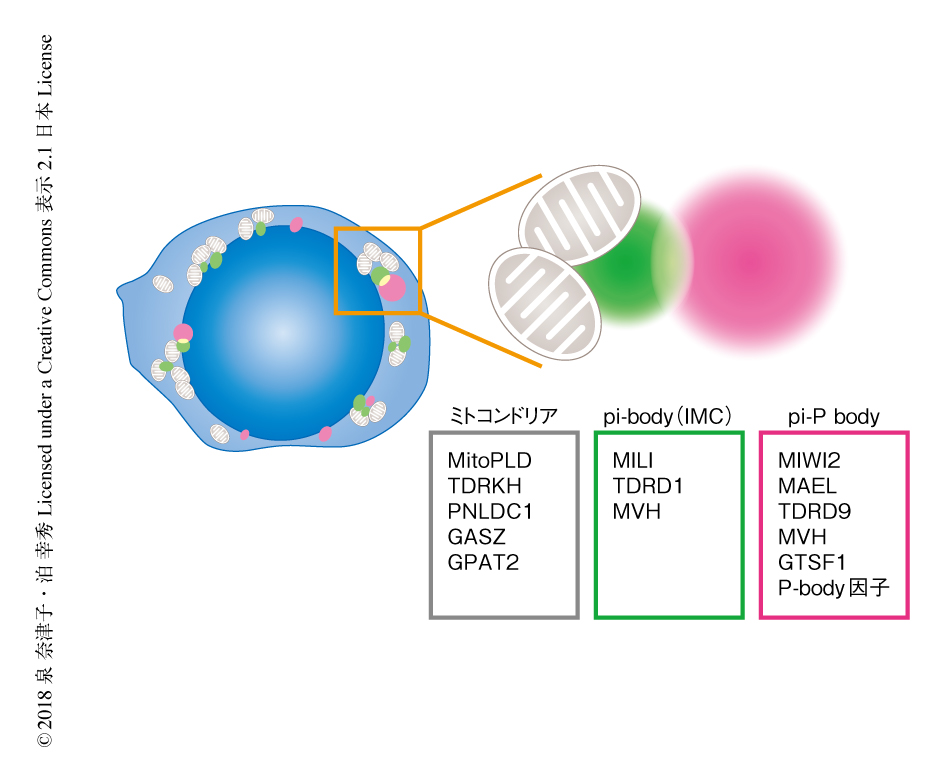
プレパキテンpiRNAの生成およびその役割:プレパキテンpiRNAはさらに,胎仔期プレパキテンpiRNAと,出生後からパキテン期よりまえに発現する出生後プレパキテンpiRNAとに区別されるが,いずれもパキテンpiRNAに比べ,トランスポゾンに由来するものが多いという特徴がある69).また,転写産物から生成されるgenic piRNAの割合は胎仔期より出生後のほうが高いことが報告されている.以降は,解析の進んでいる胎仔期プレパキテンpiRNAの生成機構について述べる.胎仔期piRNAは,piRNAクラスターに由来する転写産物,あるいは,トランスポゾンRNAからプライマリー経路およびピンポン経路を介して生成される.マウスのpiRNAクラスターは,ゲノムの片方の鎖からpiRNAを生成するユニストランドクラスターが多く,RNAポリメラーゼIIによりキャップ構造およびポリA鎖の付加されたmRNA様の長い転写産物が生成される(図5b).piRNAクラスターからの転写産物は,ショウジョウバエと同様のプライマリー経路によりpiRNAへとプロセシングされる.細胞質へと輸送された転写産物は,まず,ZucのホモログであるMitoPLD 59,60)(文献60) は新着論文レビュー でも掲載)により(おもに)ウリジンのまえで切断されると考えられている.この過程には,ATP依存性のRNAヘリカーゼであるMOV10L1(Armiのホモログ)の介在が示唆されている70).これにより生じた5'末端がウリジンのRNA断片はMILIに取り込まれたのち,さらに30~40塩基ほど下流の領域の(おもに)ウリジンのまえでMitoPLDにより切断され,piRNA前駆体の3'末端が形成される27,28).ショウジョウバエのZucと同様に,このMitoPLDによる切断は,piRNA前駆体の3'末端の形成と同時に下流に新たなpiRNA前駆体の5'末端を生じることによりphased piRNAの生成をひき起こす27,28)(図5b).MILIに取り込まれたpiRNA前駆体は,そののち,TDRKHによりミトコンドリア膜へとリクルートされ57),カイコのTrimmerのホモログである3'-5'エキソヌクレアーゼPNLDC1 71)(新着論文レビュー でも掲載)により3'末端がトリミングされ72-74),メチル化酵素Hen1による2'-O-メチル化修飾をうけて75),成熟したpiRNAになる.このようにプライマリー経路により生成されたMILI結合piRNAは1Uバイアスを示す.また,MILI結合piRNAの生成にはミトコンドリア外膜タンパク質であるGASZやGPAT2も必要であることが報告されている63,76).piRNAと結合したMILIは,その配列と相補的なトランスポゾンRNAを切断することによりその発現を抑制する.MILIによる切断により生じた3'側のRNA断片は,さらにピンポン経路によりMILIあるいはMIWI2に取り込まれる6)(図6).一方,MIWI2のスライサー活性はpiRNAの生成やトランスポゾンの抑制には必要ではなく6),MIWI2からMILIへのピンポン反応は起こらないと考えられている.MILIあるいはMIWI2に取り込まれた3'側のRNA断片は,プライマリー経路と同様に,30~40塩基ほど下流の領域でMitoPLDにより切断されてpiRNA前駆体となり,PNLDC1により3'末端がプロセシングされ成熟したpiRNAになる.このようにピンポン経路により生成されたセカンダリーpiRNAは10Aバイアスを示し,5'側の10塩基がプライマリー経路により生成されたpiRNAと相補的になる.ショウジョウバエと同様に,マウスのピンポン経路においても,MitoPLDの活性はpiRNA前駆体の3'末端の形成と同時に,下流に新たなpiRNA前駆体の5'末端を生じることでphased piRNAを生成し,これらはMIWI2あるいはMILIに取り込まれる27,28,77).このように,piRNAと結合したMILIは細胞質においてトランスポゾンRNAの切断を介してその発現を抑制するとともに,ピンポン経路によるpiRNAの生成およびphased piRNAの生成をひき起こすことによりトランスポゾンに対するpiRNAを増幅する.一方,piRNAと結合したMIWI2は核へと移行し,LINE1やIAPなどのレトロトランスポゾンの発現制御領域のCpGメチル化をひき起こすことにより転写を抑制する54,78)(図6).くわしい分子機構は明らかにされていないが,MIWI2がpiRNAを介して転写されたトランスポゾンRNAと結合し,DNAメチル化酵素DNMTなどをリクルートするモデルが考えられている.MILIの欠損により,MIWI2へのpiRNAの結合および核への局在が顕著に減少し,トランスポゾンの発現制御領域のCpGメチル化が低下する54,78).これらのことから,MIWI2へのpiRNAの取り込みの大部分は,MILIによる標的RNAの切断に依存したピンポン経路を介すると考えられている.一方で,最近の研究から,MIWI2を介さずにMILIがトランスポゾンの発現制御領域のCpGメチル化をひき起こす経路の存在も示唆されている79).胎仔期piRNAが生成される前精原細胞において,MILIはTDRD1およびMVHとともにミトコンドリアの間隙にみられるpi-bodyあるいはIMC(intermitochondrial cement)とよばれる構造体に局在する80)(図7).また,ミトコンドリア外膜にはMitoPLD,TDRKH,PNLDC1,GASZ,GPAT2など,さまざまなpiRNA因子が局在することが知られている.一方で,MIWI2はTDRD9,MAEL,MVH,GTSF1といったpiRNA因子やP-bodyの構成因子とともにpiP-bodyという場を形成する80)(図7).pi-bodyおよびpiP-bodyは核の近傍で互いに近接した局在を示すことから,これらの構造体においてピンポン経路によるpiRNAの生成が起こると考えられている.実際,pi-bodyおよびpiP-bodyの両方に局在するMVHのATPase活性は,MILIにより切断されたRNA断片がMIWI2へとうけわたされるのに必要であり,MVHの変異体においてMIWI2結合piRNAは生成されない81,82).また,piP-bodyに局在するGTSF1は,MILIによる標的RNAの切断に関与することが示唆されており,GSTF1の変異体においてはピンポン経路を介したMILIおよびMIWI2へのpiRNAの取り込みがみられなくなることが報告されている83).同様に,pi-bodyの構成因子であるTDRD1と相互作用することの知られているHsp90のコシャペロンFKBP6(Shutdownのホモログ)も,MIWI2結合piRNAの生成に必要であることが示されている84).
パキテンpiRNAの生成およびその役割:パキテンpiRNAは,プレパキテンpiRNAに比べ,トランスポゾンに由来するものが少なく,遺伝子間領域など非特定の配列に由来するものが多い69).また,精細胞の小分子RNAの大部分をしめるほど大量に発現する.パキテンpiRNAを生成するpiRNAクラスターからの転写の多くは,転写因子A-MYBにより正に制御されることが明らかにされている85)(図5b).A-MYBはまた,MIWIやほかのpiRNA因子をコードする遺伝子の転写も促進することから,この時期において,piRNAクラスターの転写とpiRNA因子の発現は協調的に制御される.また,一部のパキテンpiRNAを生成するpiRNAクラスターには核タンパク質BTBD18が結合し,RNAポリメラーゼIIによる転写の伸長を促進することによりパキテンpiRNAの生成に寄与する86).パキテン期においてはプライマリー経路によってのみpiRNAが生成されると考えられてきたが,最近の研究から,MILIやMIWIによる切断がpiRNAの生成のトリガーになることが示唆されている(図5b).MILIあるいはMIWIにより切断されたpiRNAクラスターに由来する転写産物はMILIあるいはMIWIに取り込まれ,さらに,3'側がMitoPLDにより切断されpiRNA前駆体になる.さらに,piRNA前駆体はプレパキテンpiRNAと同様な様式により3'末端がプロセシングされ成熟したpiRNAになる.この場合も,MitoPLDによる切断により生じた3'側のRNA断片からは,MILIあるいはMIWIに取り込まれたphased piRNAが3'方向へ連続的に生成される73).このようにして生成されたMILIあるいはMIWIと結合したパキテンpiRNAは1Uバイアスを示す.piRNA因子のひとつであるRNF17(Qin/Kumoのホモログ)は,この時期にピンポン経路によるpiRNAの増幅を抑制し,パキテンpiRNAクラスターの転写産物から適切にpiRNAが生成されるのに必要だと考えられている87).パキテンpiRNAがおもに生成される円形精細胞においては,piP-bodyは消失し,MILIおよびMIWIはMVH,TDRD6,TDRD7などのTudorドメインタンパク質とともに核膜の近傍のchromatoid bodyに局在する.chromatoid bodyには,piRNA因子だけでなく,RNA結合タンパク質,miRNA関連因子,ナンセンスmRNA分解因子,翻訳関連因子などさまざまなタンパク質が局在することから,mRNAの転写後の制御にかかわる構造体であると考えられている.パキテンpiRNAの機能のひとつは,転写後の制御によるレトロトランスポゾンLINE1の発現の抑制にあるが5),遺伝子間領域などに由来する大量のパキテンpiRNAは,相補的な配列をもつ明確な標的RNAが存在しないことから,その役割は謎につつまれている.最近の研究において,パキテンpiRNAの一部がMIWIのスライサー活性により精子の形成にかかわるmRNAの発現を制御する可能性や88),伸長精細胞において脱アデニル化酵素CAF1とともに内在性のmRNAの排除にはたらく可能性が示唆されている89).piRNAの主要な機能はトランスポゾンの抑制にあるが,このようなpiRNAがトランスポゾン以外の標的の発現を制御することはほかの生物においても知られている.たとえば,カイコにおいては,雌の性染色体であるW染色体から発現するfem piRNAが,雄化をひき起こすMasc mRNAを切断することによりその発現を抑制し,雌化をひき起こすことが明らかにされている90)(新着論文レビュー でも掲載).
これまでみてきたように,ショウジョウバエおよびマウスにおいて,プライマリー経路におけるpiRNAの生成,および,ピンポン経路におけるpiRNAの増幅,さらに,それらに付随したphased piRNAの生成など,piRNAの生成機構の大枠は保存されており,これらはゼブラフィッシュやカイコなどでも類似することが明らかにされている.
線虫のpiRNAはショウジョウバエやマウスより長さが短く,21塩基で1塩基目がウリジンではじまるものが多いことから21U-RNAとよばれている.線虫にはPRG-1およびPRG-2の2つのPIWIタンパク質が存在するが,piRNA経路においてはおもにPRG-1が機能する.ショウジョウバエやマウスのpiRNAとは異なり,21U-RNAはそれぞれが個別にゲノム(遺伝子間領域あるいはイントロン領域)にコードされており,約28~29塩基の短い21U-RNA前駆体がRNAポリメラーゼIIにより転写される91-93)(図8a).21U-RNAの遺伝子座の多くは転写ユニットの40塩基ほど上流にFKHモチーフをもち,転写因子であるFKHファミリータンパク質に依存的に転写される91,94).21U-RNA前駆体の生成に必要な因子としてTOFU-3,TOFU-4,TOFU-5,PRDE-1が同定されている92,93).このうち,PRDE-1はFKHファミリータンパク質に依存的な21U-RNA前駆体の生成にのみ関与する92).21U-RNA前駆体は21U-RNAの正確に2塩基上流から転写されることから,28~29塩基の21U-RNA前駆体は,キャップ構造が削除されたのち,さらに5'末端の2塩基が除去され,PRG-1に取り込まれると考えられている.この過程に必要な因子としてPID-1,TOFU-1,TOFU-2が同定されている93,95).PRG-1に取り込まれた21U-RNA前駆体は,PARNのホモログであるPARN-1により3'末端がトリミングされ96),メチル化酵素HENN-1により2'-O-メチル化修飾をうけ97-99),成熟した21U-RNAになる.さきに述べたように,線虫は独自のpiRNA経路をもっており,それを反映するように,線虫には保存されていないpiRNA因子も多く,さきにあげたpiRNAの生成にかかわる因子も線虫に特有のものである.
21U-RNAと相補的な配列をもつトランスポゾンはTc3しか知られておらず,21U-RNAの標的はながらく不明であったが,のちの解析により,21U-RNAはmiRNAのように数塩基のミスマッチを許容して標的RNAを認識することが明らかにされた100-102).標的の認識においては2~7塩基目のシード領域が重要であり,14~19塩基目の3'サプリメント領域の相補性も21U-RNAによる発現の抑制に寄与する101,102).また,9~13塩基目のセントラル領域のミスマッチが許容されるのと一致して,21U-RNA:PRG-1複合体による標的RNAの発現の抑制にはPRG-1のスライサー活性は必要でないことが示されている100,103).
これまでの研究から,21U-RNA:PRG-1複合体は,発現抑制のトリガーとしてのみ機能し,内在性の22G-siRNA経路を活性化することにより標的RNAの発現を抑制することが明らかにされている100,103).21U-RNA:PRG-1複合体は生殖細胞において発現する種々のRNAをスキャンし,標的RNAを認識するとRNA依存性のRNAポリメラーゼをリクルートすることにより2本鎖RNAを形成し,新たな22G-RNAの生成をひき起こす(図8b).この22G-RNAは線虫に特異的なAGOであるWAGOに取り込まれる.22G-RNA:WAGO複合体は相補的な配列をもつRNAを切断することによりその発現を抑制する.さらに,WAGOのひとつであるHRDE-1は22G-RNAと結合すると核へと移行し,NRDE-1,NRDE-2,NRDE-4に依存してヒストンH3のLys9のトリメチル化修飾を介して標的となる配列の転写を抑制する104,105).発現の抑制のシグナルがどのように伝達されるのか,その詳細は明らかにされていないが,核に局在するWAGOによる転写の抑制の効果は世代をこえてうけつがれ,いちど核において発現の抑制が確立されると,その維持には21U-RNAは必要ないことが示されている100,104-106)(文献106) は新着論文レビュー でも掲載).
21U-RNA:PRG-1複合体は数塩基のミスマッチを許容して標的RNAを認識するため,外来性の多様な配列にも対応する一方で,内在性のmRNAもその標的になりうる.これまでに,内在性のmRNAが21U-RNAによる発現の抑制を回避する2つの分子機構が報告されている(図8b).ひとつは,CSR-1の内在性のmRNAへの結合である.線虫の生殖細胞においては,WAGOにくわえ,CSR-1というAGOが発現する.CSR-1は内在性のmRNAを標的とする22G-RNAと結合するが,その発現は抑制しない.CSR-1が標的とする内在性のmRNAにおいては,21U-RNA:PRG-1複合体の結合および下流の22G-RNAの生成が抑制されることが示されており,21U-RNAの内在性のmRNAへの結合をCSR-1が阻止するというモデルが提唱されている101,106,107)(文献107) は新着論文レビュー でも掲載).また,線虫のゲノムにはperiodic An/Tn cluster(PATC)とよばれる,連続したAやTを周期的にくり返す線虫に特有の非コード配列が存在する.PATCはイントロンや遺伝子間領域に多くみられ,生殖細胞に発現する内在性の遺伝子はイントロンにおけるPATCの割合が高い傾向がある.さらに,イントロンにPATCをもつmRNAは,21U-RNAの標的となる配列が存在していても,21U-RNAによる発現の抑制をまぬがれることが示されている102).これらのことから,イントロンに存在するPATCがなんらかのかたちで自己のライセンスとしてはたらき,21U-RNAによる発現の抑制を回避すると考えられている.
piRNAの発見から10年以上が経過した.次世代シークエンサー技術の発展とともにpiRNAの研究は大きく進展し,piRNA経路にかかわる因子の同定およびその生成機構や作用機序の大枠が明らかにされつつある.一方で,piRNA経路にかかわる個々の因子の分子機能や相互関係についての理解はいまだ途上にあり,さらなる解明が望まれる.このレビューにおいてはほとんど言及しなかったが,piRNAの機能は,トランスポゾンの発現の抑制にとどまらず,mRNAの発現や局在の制御,さらに,体細胞における機能へと大きな拡がりをみせている.また,がん細胞におけるPIWIやpiRNAの異所的な発現から,piRNA経路とがんとの関係も注目されている.今後,piRNAの生成機構および作用機序の理解がさらに進むとともに,体細胞におけるpiRNAの機能の解明,また,その制御のしくみが明らかにされることを期待したい.
略歴:2009年 横浜市立大学大学院医学研究科博士課程 修了,2010年 東京大学分子細胞生物学研究所 研究員を経て,2014年より同 助教(現 東京大学定量生命科学研究所 助教).
研究テーマ:piRNAの生成機構.
泊 幸秀(Yukihide Tomari)
東京大学定量生命科学研究所 教授.
研究室URL:http://www.iam.u-tokyo.ac.jp/tomari/
© 2018 泉 奈津子・泊 幸秀 Licensed under CC 表示 2.1 日本
(東京大学定量生命科学研究所 RNA機能研究分野)
email:泊 幸秀
領域融合レビュー, 7, e003 (2018) DOI: 10.7875/leading.author.7.e003
Natsuko Izumi & Yukihide Tomari: The biogenesis and function of PIWI-interacting RNAs.
要 約
RNAサイレンシング機構のひとつとして知られるpiRNA経路は,動物の生殖巣において,おもにトランスポゾンの発現の抑制に機能する.piRNAは約30塩基の小分子RNAで,配列に相補性のあるトランスポゾンRNAへとPIWIタンパク質をガイドすることにより,転写レベルおよび転写後レベルにおいてその発現を抑制する.piRNA経路にかかわる因子の欠損はトランスポゾンの活性化や配偶子の形成不全をひき起こすことから,piRNA経路は生殖細胞のゲノムの完全性を維持するうえできわめて重要な役割を担うと考えられている.これまでの精力的な研究により,多数の因子が関与する複雑なpiRNA経路の全体像がしだいに明らかにされてきた.自らのゲノムに保持されるトランスポゾンの情報をもとに,piRNAはどのように生成され機能するのか,その分子機構について最近の知見をまじえて解説する.
はじめに
ヒトのゲノムの約45%は,トランスポゾンとよばれる転移因子やその残骸からなるくり返し配列によりしめられることが明らかにされている.トランスポゾンは,自らの配列を切り出し別の部位に転移するDNA型トランスポゾンと,自らの配列からの転写産物をDNAへと逆転写し,新たな部位へと挿入することにより増幅するレトロトランスポゾンとに分類される.これらトランスポゾンの活性は,遺伝子の多様性の獲得やゲノムにダイナミックな変化をもたらすことにより生物の進化に寄与してきたと考えられている.その一方で,トランスポゾンの転移は遺伝子の発現を撹乱したり遺伝子の変異をひき起こしたりするため,正確な遺伝情報の維持のため,その活性は適切に制御されなければならない.通常,トランスポゾンの領域はDNAメチル化や抑制性のヒストン修飾により転写が抑制されているが,動物の生殖細胞には,さらにRNAサイレンシング機構によりトランスポゾンを抑制するしくみとしてpiRNA経路が存在する.その中心となるのが,piRNA(PIWI-interacting RNA)およびそのパートナーであるPIWIタンパク質(PIWI)である.piRNAとPIWIは複合体を形成し,主として標的となるトランスポゾンの発現を転写レベルおよび転写後レベルで抑制する.これによりトランスポゾンの脅威から生殖細胞のゲノムを保護し,次世代への正確な遺伝情報の継承を保障する.また,piRNA経路により抑制されるべきトランスポゾンなどの非自己に由来する配列の一部は,生殖細胞を介して次世代へとうけつがれることも明らかにされている.
このレビューにおいては,これまでの研究により明らかにされてきたショウジョウバエおよびマウスにおけるpiRNAの生成機構および作用機序を,最近の知見をまじえて解説する.また,ショウジョウバエやマウスとは異なる方法で非自己に由来する配列を識別し抑制する機構を獲得してきた線虫のpiRNA経路についても言及する.
なお,RNAサイレンシングの構造基盤については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001 (2012) も参照されたい.
1.piRNAとPIWIタンパク質
piRNAは動物の生殖細胞に高く発現する24~31塩基の小分子RNAであり,Argonaute(AGO)ファミリーに属するPIWIタンパク質と複合体を形成して機能する(図1).piRNAの配列は多様で,その配列に種間での保存性はみられないが,1塩基目がウリジンのものが多く(1Uバイアス),3'末端のリボースは2'-O-メチル化修飾されているという特徴がある.piRNAの1Uバイアスは,AGOと同様に,piRNAの1塩基目を認識するPIWIのMIDドメインとの結合の安定性により説明される1,2)(文献1) は新着論文レビュー でも掲載,文献2) は新着論文レビュー でも掲載).同様に,piRNAの3'末端を認識するPIWIのPAZドメインは,AGOのPAZドメインに比べ,2'-O-メチル化修飾をもつ塩基とより安定に結合しやすい構造をしており3,4),piRNAの3'末端はこの2'-O-メチル化修飾によりヌクレアーゼによる分解から保護されると考えられている.
piRNAはトランスポゾンに対し相補的な配列をもつものが多く,piRNA-PIWI複合体の主要な役割はトランスポゾンの発現の抑制にある.PIWIを欠損した変異体やpiRNAを生成できない変異体においては,トランスポゾンの脱抑制とともに配偶子の形成が損なわれ不妊になる.これらのことから,piRNA経路はトランスポゾンの活性を抑制することにより生殖細胞のゲノムをまもる役割があると考えられている.
PIWIはAGOと同様にスライサー活性をもち,piRNAと結合したPIWIは,piRNAと相補的な配列をもつトランスポゾンRNAを認識すると,piRNAの10塩基目と11塩基目とのあいだで標的RNAを切断する.これにより,トランスポゾンRNAの転写後の抑制に機能する.piRNAの長さはsiRNAと比較するとやや長いが,PIWIによる標的RNAの切断には2~21塩基目までの相補性で十分であることが示されている5).また,のちに述べるように,PIWIによる標的RNAの切断は,標的RNAの発現を抑制するだけでなく新たなpiRNAの生成とも共役している.一方,ショウジョウバエのPiwiやマウスのMIWI2など核に局在する一部のPIWIは,スライサー活性に非依存的に標的となるトランスポゾンの転写の抑制に機能する6-8).
2.piRNAの生成にかかわる因子
piRNAはおもにpiRNAクラスターからの転写産物やトランスポゾンに由来するRNAから生成される.piRNAクラスターとは機能を失ったトランスポゾンの残骸が蓄積したゲノム領域のことで,長いものでは数百kbpにおよぶものもある9).また,piRNAの一部はmRNAのおもに3'側非翻訳領域からも生成されており,これらはgenic piRNAとよばれている10).
miRNAやsiRNAとは異なり,piRNAは中間体として2本鎖の状態を介さず,長い1本鎖の転写産物が細胞質においてプロセシングされることにより生成される.その過程には,多数のTudorドメインタンパク質,RNAヘリカーゼ,ヌクレアーゼが必要である11)(図2).PIWIのおもにN末端領域のアルギニン残基は,Capsuleen/PRMT5により対称的ジメチルアルギニン修飾されることが知られている12).Tudorドメインタンパク質は,TudorドメインによりPIWIの対称的ジメチルアルギニン修飾を認識して結合する.Tudorドメインタンパク質とPIWIとの相互作用は,piRNAの生成およびPIWIの局在に重要であることが示されている.
piRNAの生成経路は,既存のpiRNAを必要としないプライマリー経路と,PIWIのスライサー活性に依存したピンポン経路とに大別される.ピンポン経路においては,PIWIによる標的RNAの切断と共役して新たなpiRNAが生成される.以下,ショウジョウバエ,マウス,線虫それぞれのpiRNA経路について解説する.
3.ショウジョウバエにおけるpiRNA経路
ショウジョウバエにはPiwi,Aubergine(Aub),Argonaute3(Ago3)の3つのPIWIタンパク質が存在し,そのいずれの欠損でもトランスポゾンの脱抑制や配偶子の形成の異常がみられ,雌雄とも不妊になる.piRNAの研究はショウジョウバエの卵巣においてもっとも進んでおり,以降は,ショウジョウバエの卵巣における解析から明らかにされたことについて述べる.
ショウジョウバエの卵巣を構成する卵室は,生殖細胞である哺育細胞および卵母細胞が卵巣性の体細胞である濾胞細胞にかこまれた構造をとる(図3a).3つのPIWIのうち,Piwiは生殖細胞および濾胞細胞の両方に発現するが,AubおよびAgo3は生殖細胞のみに発現する9).また,Piwiは核に局在し転写の抑制に機能するのに対し,AubおよびAgo3は細胞質の核の周囲の顆粒構造“ヌアージュ”(フランス語で“雲”の意)に局在し,標的RNAを切断することにより転写後の抑制に機能する9).さきに述べたように,piRNAの生成経路はプライマリー経路とピンポン経路とに大別されるが,濾胞細胞においてはプライマリー経路のみが,生殖細胞においてはプライマリー経路およびピンポン経路が機能する.
piRNAクラスターおよびpiRNAクラスターからの転写:ショウジョウバエのpiRNAクラスターはセントロメアやテロメアの近傍に多く,転写の様式から,ゲノムの片方の鎖から転写産物を生成するflamencoなどのユニストランドクラスター(図3b)と,ゲノムの両方の鎖から転写産物を生成する42ABなどのデュアルストランドクラスター(図3c)とに分類される.濾胞細胞において発現するpiRNAはユニストランドクラスターに由来するものが多く,その転写産物はトランスポゾンに対しアンチセンス方向のトランスポゾンの断片を多く含むことが知られている.一方,生殖細胞において発現するpiRNAはデュアルストランドクラスターに由来するものが多い.いずれのpiRNAクラスターにも,ヘテロクロマチン化のシグナルであるヒストンH3のLys9のトリメチル化修飾がみられ,piRNAクラスターからのpiRNAの生成にはヒストンH3のLys9のメチル化酵素が必要である13).ヒストンH3のLys9のトリメチル化修飾は抑制性のヒストン修飾として知られるが,デュアルストランドクラスターにおいてはその転写に不可欠である.デュアルストランドクラスターは明確なプロモーター領域をもたず,その転写はヒストンH3のLys9のトリメチル化修飾と結合するヘテロクロマチンタンパク質HP1のホモログであるRhinoに依存する14).Rhinoは,生殖細胞に特異的に発現する基本転写因子のサブユニットのホモログであるMoonshinerをリクルートすることにより転写開始複合体の形成をひき起こし,piRNAクラスターの内部からの転写を活性化する15,16)(図3c).デュアルストランドクラスターからは,キャップ構造をもたずスプライシングをうけない特殊な転写産物がRNAポリメラーゼIIにより生成される16,17).RhinoはさらにDeadlockを介してCutoff(Cuff)とRDC複合体を形成する16).Cuffは,piRNAクラスターからの転写産物の5'末端と結合することにより転写産物を安定化するとともに,RNAのプロセシングや転写の終結を抑制し,デュアルストランドクラスターからの転写を促進すると考えられている17,18).デュアルストランドクラスターからの転写産物には,核においてUAP56を含むTREX複合体がCuffに依存的に結合し,細胞質のヌアージュへと輸送される19,20).一方,ユニストランドクラスターからの転写はヒストンH3のLys4のジメチル化修飾をもつ推定上のプロモーター領域からRhinoに非依存的に開始される16).その転写産物はmRNAと同様にキャップ構造およびポリA鎖をもち,選択的スプライシングをうけうることから,デュアルストランドクラスターとは転写の機構が明確に区別される16)(図3b).
プライマリー経路におけるpiRNAの生成:プライマリー経路は濾胞細胞および生殖細胞の両方において機能しており,Piwiと結合するpiRNAおよび一部のAubと結合するpiRNAを生成する.以下,濾胞細胞においてプライマリー経路によりPiwiと結合するpiRNAが生成される過程について述べる.piRNAクラスターからの転写産物がプライマリー経路に入るには,転写産物のもつシス配列の関与が示唆されている.たとえば,濾胞細胞における主要なpiRNAクラスターであるflamencoからの転写産物には,5'末端に下流からのpiRNAの生成をひき起こすことのできるpiRNAトリガー配列(piRNA-trigger sequence:PTS)が同定されている21).また,濾胞細胞においてgenic piRNAを生成する主要なmRNAであるtraffic jamの3'側非翻訳領域にも,下流からのpiRNAの生成をひき起こす約100塩基のシス配列が同定されており,このシス配列にTudorドメインタンパク質であるYbが結合することによりpiRNAの生成がひき起こされる22).さらに,濾胞細胞において任意の転写産物にYbやArmiを結合させることによっても,下流の領域からpiRNAの生成がひき起こされる23,24).これらのことから,濾胞細胞においては,YbがpiRNAクラスターからの転写産物をプライマリーpiRNA経路にみちびくと考えられている(図3d).piRNAクラスターからの長い転写産物は,細胞質においてなんらかのヌクレアーゼによりプロセシングされ,5'末端にリン酸基がなく3'末端が2',3'-環状リン酸となったpiRNA中間体になると予想されている8).ArmiはATP依存性のRNAヘリカーゼで,Ybに依存的にpiRNA中間体と結合する8).Armiと結合したpiRNA中間体は,ミトコンドリア外膜に存在するエンドヌクレアーゼZucchini(Zuc)へとリクルートされ切断される25,26)(文献25) は新着論文レビュー でも掲載).バイオインフォマティクス解析により,ZucによるpiRNA中間体の切断はウリジンのまえで起こりやすいと考えられているが,精製したZucのエンドヌクレアーゼ活性はウリジンへの嗜好性を示さないことから,なんらかの補因子の存在が想定されている25-28).Zucによる切断により生じた1Uバイアスのある3'側のRNA断片はpiRNA前駆体としてPiwiに取り込まれる.PIWIへのpiRNA前駆体の取り込みには,Hsp90およびコシャペロンであるShutdownの関与が示唆されている29,30).Piwiに取り込まれたpiRNA前駆体は3'側がふたたびZucにより切断され27,28),Hen1による2'-O-メチル化修飾をうけて成熟したpiRNAになる31,32).ZucによるpiRNAの3'末端の形成は,同時に下流に新たなpiRNA前駆体を生じ,3'方向へと連続的にPiwiと結合したphased piRNAを生成する27,28).このようにして,ひとつの転写産物から多様な配列をもつ多くのpiRNAが生成される.プライマリー経路におけるpiRNAの生成には,ほかにもTudorドメインタンパク質であるVreteno 33),ミトコンドリア外膜タンパク質であるGasZやMinotaurが必要であることが示されており34-36),GasZはArmiの局在の制御にかかわることが示唆されている35,36).濾胞細胞において,Ybは核膜の近傍のYb-bodyとよばれる構造体に局在する8)(図3d).Yb-bodyにはYbやArmiをはじめ,プライマリー経路に必要なVretenoやShutdownなどが局在し,ミトコンドリアの近傍に観察されることから,Yb-bodyは濾胞細胞におけるpiRNAの生成の場であると考えられている.一方,生殖細胞にYbは発現しておらず,YbのホモログであるBrother of YbやSister of Ybがその機能を代替することが示唆されているが33),Yb-bodyに相当する構造体はなく,ヌアージュがpiRNAの生成の場であると考えられている.
Piwiと結合したpiRNAの核における機能:PiwiはpiRNAと結合すると核移行シグナルに依存して核へと移行し,piRNAの配列に相補的なトランスポゾンRNAと結合する.さらに,PiwiはAsterix/Gtsf1やPanoramix/Silencioと複合体を形成し,ヒストンH3のLys9のメチル化酵素であるEgglessをリクルートすることによりトランスポゾンの領域においてヒストンH3のLys9のトリメチル化をひき起こす37-41)(図3e).Piwiは,ヒストンH3のLys9のトリメチル化修飾と並行して,リンカーヒストンH1を介したクロマチン構造の変化をひき起こすことによっても標的となる領域のヘテロクロマチン化を促進し,トランスポゾンの発現を転写レベルで抑制する42)(新着論文レビュー でも掲載).Piwiによるトランスポゾンの転写の抑制にはMaelstromも必要であるが,MaelstromはヒストンH3のLys9のトリメチル化には関与せず,その作用点は明らかにされていない7).最近,Piwi,Asterix,Panoramix,Maelstromなど,Piwiとともに核において転写の抑制に機能するpiRNA因子の消失により,トランスポゾンmRNAのスプライシングのパターンが変化することが明らかにされた43).くわしい分子機構は不明であるが,PIWIやこれらのpiRNA因子はpiRNAを介してクロマチンの状態を変化させることにより,トランスポゾンmRNAのスプライシングを負に制御してトランスポゾンの活性を抑制するようである.
ピンポン経路におけるpiRNAの生成および転写後の抑制:ピンポン経路によるpiRNAの生成機構は,Aubと結合するpiRNAとAgo3と結合するpiRNAの5'側の10塩基が互いに相補的であるものが多いという事実から提唱された9,44).ピンポン経路においては,まず,母性に由来するあるいはプライマリー経路において生成されたAubと結合するアンチセンスpiRNAがそのトリガーとなり,相補的な配列をもつpiRNAクラスターからの転写産物やトランスポゾンRNAを切断する.これにより生じたセンス方向の3'側のRNA断片はpiRNA前駆体としてAgo3に取り込まれる(図4).このとき,KrimperはpiRNAと結合していないAgo3をヌアージュへとリクルートし,Ago3と結合したセンスpiRNAの生成に機能する45,46)(文献45) は新着論文レビュー でも掲載).一方,Qin/Kumoは,Aubによる切断により生じたRNA断片がAubやPiwiに取り込まれるのを抑制することにより,AubとAgo3とのあいだのピンポン反応を促進する47).また,Aubによる切断により生じたRNA断片がAubからAgo3へとうけわたされる過程には,VasaのATPase活性が必要であることが示唆されている48,49).これらピンポン経路におけるpiRNAの生成に必要な因子も,AubやAgo3と同様にヌアージュに局在することが知られている.ショウジョウバエにおけるpiRNAの3'末端の形成機構には,Zucによる切断,および,PIWIによる切断とそれにつづく3'-5'エキソヌクレアーゼNibblerによるトリミング,という2つの様式が知られている50).Ago3に取り込まれたpiRNA前駆体は,下流の領域においてPIWIにより切断されNibblerによりトリミングされる場合が多い50)(図4).一方,Aubに取り込まれたpiRNA前駆体は,多くの場合,Zucによる切断により直接的に3'末端が形成される50).いずれの場合も,piRNAの3'末端はさらにHen1により2'-O-メチル化修飾をうけて成熟したpiRNAになる.ZucによるpiRNA前駆体の切断は,プライマリー経路と同様,3'末端の形成と同時に新たなpiRNA前駆体の5'末端を生じる21,27,28)(図4).これらのphased piRNAはおもにPiwiに取り込まれ,核に移行して転写の抑制に機能する.phased piRNAの生成により,配列が多様化したトランスポゾンにも対応したpiRNAが生成される.このように,ピンポン経路においては,標的となるトランスポゾンRNAの切断による発現の抑制と共役して,活性化したトランスポゾンの抑制にはたらくアンチセンスpiRNAが増幅される.また,ピンポン経路により生成されたAub結合piRNAとAgo3結合piRNAは互いに5'側の10塩基が相補的であり,逆のストランドバイアスを示す.ピンポン経路が提唱された当初,Ago3結合piRNAの10Aバイアスは,1UバイアスのあるAub結合piRNAが標的RNAを切断したことにより生じたものと考えられていた.しかし,のちの解析により,piRNAの1塩基目はPIWIのMIDドメインの5'結合ポケットにより認識されており,標的RNAとの塩基の対合には寄与しないこと1),また,Aubは標的RNAを認識し切断する際,piRNAと対合する標的RNAの1塩基目がAであるもの(t1A)を好む傾向のあることが示された51).これらのことから,現在では,Ago3結合piRNAの10AバイアスはAub結合piRNAの1Uバイアスに起因するのではなく,Aubのt1A嗜好性によるものだと考えられている.一方,Ago3はAubのような標的RNAの1塩基目の嗜好性をもたないことが示唆されている.
piRNA結合Aubは,後期の卵細胞においてTudorやCapsuleenなどとともに,次世代の生殖細胞を形成する卵細胞の後極の極顆粒に局在する.さきに述べたように,このAub結合piRNAは抑制すべきトランスポゾンの配列の記憶として母親からうけつがれ,次世代の生殖細胞におけるpiRNAの生成のトリガーとしてはたらく52).母親にないトランスポゾンを父親が保持していた場合,交配により生じた個体はそのトランスポゾンに対するpiRNAを生成できず不妊の表現型を呈する.この現象はhybrid dysgenesisとよばれている.
4.マウスにおけるpiRNA経路
マウスにおいてPIWIおよびpiRNAは精巣に高く発現しており,正常な精子の形成に不可欠である.一方,卵巣におけるPIWIの発現は低く,その欠損による明らかな影響は報告されていない.マウスにおいてはMILI,MIWI2,MIWIという3つのPIWIタンパク質が精巣の発達にともない時期特異的に発現する(図5a).MILIは胎仔期から円形精子期にわたり発現するが53),MIWI2は出生の前後のごくかぎられた時期にのみ発現する54).この時期は,前精原細胞の分裂が一時的に停止し新規のDNAメチル化が起こる時期にあたる.一方,MIWIは生後の精子が形成される時期である減数分裂パキテン期から伸長精子期にかけて発現する55).MILIあるいはMIWI2のノックアウトマウスは減数分裂パキテン期のまえに精子の形成が停止し不妊になる53,56).同様な表現型は,piRNAの生成にかかわるMVH,MitoPLD,TDRKH,TDRD9,TDRD12,GASZ,MOV10L1などのノックアウトマウスにおいても観察されている57-64).一方,MIWIの欠損では,減数分裂は正常に進行するものの,円形精子のアポトーシスにより精子の形成が損なわれ不妊になる55).同様に,TDRD1,TDRD5,TDRD7,RNF17などのpiRNA因子のノックアウトマウスにおいても減数分裂ののち精子の形成は不全になることが知られている65-68).
マウスのpiRNAは,その発現の時期によりプレパキテンpiRNAとパキテンpiRNAにと大別される(図5a).プレパキテンpiRNAはMILIおよびMIWI2と結合しており,パキテンpiRNAはMILIおよびMIWIと結合する.また,MILI,MIWI2,MIWIそれぞれに結合するpiRNAの長さのピークは,26塩基,28塩基,30塩基と多少の違いのあることが知られている(図2).
プレパキテンpiRNAの生成およびその役割:プレパキテンpiRNAはさらに,胎仔期プレパキテンpiRNAと,出生後からパキテン期よりまえに発現する出生後プレパキテンpiRNAとに区別されるが,いずれもパキテンpiRNAに比べ,トランスポゾンに由来するものが多いという特徴がある69).また,転写産物から生成されるgenic piRNAの割合は胎仔期より出生後のほうが高いことが報告されている.以降は,解析の進んでいる胎仔期プレパキテンpiRNAの生成機構について述べる.胎仔期piRNAは,piRNAクラスターに由来する転写産物,あるいは,トランスポゾンRNAからプライマリー経路およびピンポン経路を介して生成される.マウスのpiRNAクラスターは,ゲノムの片方の鎖からpiRNAを生成するユニストランドクラスターが多く,RNAポリメラーゼIIによりキャップ構造およびポリA鎖の付加されたmRNA様の長い転写産物が生成される(図5b).piRNAクラスターからの転写産物は,ショウジョウバエと同様のプライマリー経路によりpiRNAへとプロセシングされる.細胞質へと輸送された転写産物は,まず,ZucのホモログであるMitoPLD 59,60)(文献60) は新着論文レビュー でも掲載)により(おもに)ウリジンのまえで切断されると考えられている.この過程には,ATP依存性のRNAヘリカーゼであるMOV10L1(Armiのホモログ)の介在が示唆されている70).これにより生じた5'末端がウリジンのRNA断片はMILIに取り込まれたのち,さらに30~40塩基ほど下流の領域の(おもに)ウリジンのまえでMitoPLDにより切断され,piRNA前駆体の3'末端が形成される27,28).ショウジョウバエのZucと同様に,このMitoPLDによる切断は,piRNA前駆体の3'末端の形成と同時に下流に新たなpiRNA前駆体の5'末端を生じることによりphased piRNAの生成をひき起こす27,28)(図5b).MILIに取り込まれたpiRNA前駆体は,そののち,TDRKHによりミトコンドリア膜へとリクルートされ57),カイコのTrimmerのホモログである3'-5'エキソヌクレアーゼPNLDC1 71)(新着論文レビュー でも掲載)により3'末端がトリミングされ72-74),メチル化酵素Hen1による2'-O-メチル化修飾をうけて75),成熟したpiRNAになる.このようにプライマリー経路により生成されたMILI結合piRNAは1Uバイアスを示す.また,MILI結合piRNAの生成にはミトコンドリア外膜タンパク質であるGASZやGPAT2も必要であることが報告されている63,76).piRNAと結合したMILIは,その配列と相補的なトランスポゾンRNAを切断することによりその発現を抑制する.MILIによる切断により生じた3'側のRNA断片は,さらにピンポン経路によりMILIあるいはMIWI2に取り込まれる6)(図6).一方,MIWI2のスライサー活性はpiRNAの生成やトランスポゾンの抑制には必要ではなく6),MIWI2からMILIへのピンポン反応は起こらないと考えられている.MILIあるいはMIWI2に取り込まれた3'側のRNA断片は,プライマリー経路と同様に,30~40塩基ほど下流の領域でMitoPLDにより切断されてpiRNA前駆体となり,PNLDC1により3'末端がプロセシングされ成熟したpiRNAになる.このようにピンポン経路により生成されたセカンダリーpiRNAは10Aバイアスを示し,5'側の10塩基がプライマリー経路により生成されたpiRNAと相補的になる.ショウジョウバエと同様に,マウスのピンポン経路においても,MitoPLDの活性はpiRNA前駆体の3'末端の形成と同時に,下流に新たなpiRNA前駆体の5'末端を生じることでphased piRNAを生成し,これらはMIWI2あるいはMILIに取り込まれる27,28,77).このように,piRNAと結合したMILIは細胞質においてトランスポゾンRNAの切断を介してその発現を抑制するとともに,ピンポン経路によるpiRNAの生成およびphased piRNAの生成をひき起こすことによりトランスポゾンに対するpiRNAを増幅する.一方,piRNAと結合したMIWI2は核へと移行し,LINE1やIAPなどのレトロトランスポゾンの発現制御領域のCpGメチル化をひき起こすことにより転写を抑制する54,78)(図6).くわしい分子機構は明らかにされていないが,MIWI2がpiRNAを介して転写されたトランスポゾンRNAと結合し,DNAメチル化酵素DNMTなどをリクルートするモデルが考えられている.MILIの欠損により,MIWI2へのpiRNAの結合および核への局在が顕著に減少し,トランスポゾンの発現制御領域のCpGメチル化が低下する54,78).これらのことから,MIWI2へのpiRNAの取り込みの大部分は,MILIによる標的RNAの切断に依存したピンポン経路を介すると考えられている.一方で,最近の研究から,MIWI2を介さずにMILIがトランスポゾンの発現制御領域のCpGメチル化をひき起こす経路の存在も示唆されている79).胎仔期piRNAが生成される前精原細胞において,MILIはTDRD1およびMVHとともにミトコンドリアの間隙にみられるpi-bodyあるいはIMC(intermitochondrial cement)とよばれる構造体に局在する80)(図7).また,ミトコンドリア外膜にはMitoPLD,TDRKH,PNLDC1,GASZ,GPAT2など,さまざまなpiRNA因子が局在することが知られている.一方で,MIWI2はTDRD9,MAEL,MVH,GTSF1といったpiRNA因子やP-bodyの構成因子とともにpiP-bodyという場を形成する80)(図7).pi-bodyおよびpiP-bodyは核の近傍で互いに近接した局在を示すことから,これらの構造体においてピンポン経路によるpiRNAの生成が起こると考えられている.実際,pi-bodyおよびpiP-bodyの両方に局在するMVHのATPase活性は,MILIにより切断されたRNA断片がMIWI2へとうけわたされるのに必要であり,MVHの変異体においてMIWI2結合piRNAは生成されない81,82).また,piP-bodyに局在するGTSF1は,MILIによる標的RNAの切断に関与することが示唆されており,GSTF1の変異体においてはピンポン経路を介したMILIおよびMIWI2へのpiRNAの取り込みがみられなくなることが報告されている83).同様に,pi-bodyの構成因子であるTDRD1と相互作用することの知られているHsp90のコシャペロンFKBP6(Shutdownのホモログ)も,MIWI2結合piRNAの生成に必要であることが示されている84).
パキテンpiRNAの生成およびその役割:パキテンpiRNAは,プレパキテンpiRNAに比べ,トランスポゾンに由来するものが少なく,遺伝子間領域など非特定の配列に由来するものが多い69).また,精細胞の小分子RNAの大部分をしめるほど大量に発現する.パキテンpiRNAを生成するpiRNAクラスターからの転写の多くは,転写因子A-MYBにより正に制御されることが明らかにされている85)(図5b).A-MYBはまた,MIWIやほかのpiRNA因子をコードする遺伝子の転写も促進することから,この時期において,piRNAクラスターの転写とpiRNA因子の発現は協調的に制御される.また,一部のパキテンpiRNAを生成するpiRNAクラスターには核タンパク質BTBD18が結合し,RNAポリメラーゼIIによる転写の伸長を促進することによりパキテンpiRNAの生成に寄与する86).パキテン期においてはプライマリー経路によってのみpiRNAが生成されると考えられてきたが,最近の研究から,MILIやMIWIによる切断がpiRNAの生成のトリガーになることが示唆されている(図5b).MILIあるいはMIWIにより切断されたpiRNAクラスターに由来する転写産物はMILIあるいはMIWIに取り込まれ,さらに,3'側がMitoPLDにより切断されpiRNA前駆体になる.さらに,piRNA前駆体はプレパキテンpiRNAと同様な様式により3'末端がプロセシングされ成熟したpiRNAになる.この場合も,MitoPLDによる切断により生じた3'側のRNA断片からは,MILIあるいはMIWIに取り込まれたphased piRNAが3'方向へ連続的に生成される73).このようにして生成されたMILIあるいはMIWIと結合したパキテンpiRNAは1Uバイアスを示す.piRNA因子のひとつであるRNF17(Qin/Kumoのホモログ)は,この時期にピンポン経路によるpiRNAの増幅を抑制し,パキテンpiRNAクラスターの転写産物から適切にpiRNAが生成されるのに必要だと考えられている87).パキテンpiRNAがおもに生成される円形精細胞においては,piP-bodyは消失し,MILIおよびMIWIはMVH,TDRD6,TDRD7などのTudorドメインタンパク質とともに核膜の近傍のchromatoid bodyに局在する.chromatoid bodyには,piRNA因子だけでなく,RNA結合タンパク質,miRNA関連因子,ナンセンスmRNA分解因子,翻訳関連因子などさまざまなタンパク質が局在することから,mRNAの転写後の制御にかかわる構造体であると考えられている.パキテンpiRNAの機能のひとつは,転写後の制御によるレトロトランスポゾンLINE1の発現の抑制にあるが5),遺伝子間領域などに由来する大量のパキテンpiRNAは,相補的な配列をもつ明確な標的RNAが存在しないことから,その役割は謎につつまれている.最近の研究において,パキテンpiRNAの一部がMIWIのスライサー活性により精子の形成にかかわるmRNAの発現を制御する可能性や88),伸長精細胞において脱アデニル化酵素CAF1とともに内在性のmRNAの排除にはたらく可能性が示唆されている89).piRNAの主要な機能はトランスポゾンの抑制にあるが,このようなpiRNAがトランスポゾン以外の標的の発現を制御することはほかの生物においても知られている.たとえば,カイコにおいては,雌の性染色体であるW染色体から発現するfem piRNAが,雄化をひき起こすMasc mRNAを切断することによりその発現を抑制し,雌化をひき起こすことが明らかにされている90)(新着論文レビュー でも掲載).
これまでみてきたように,ショウジョウバエおよびマウスにおいて,プライマリー経路におけるpiRNAの生成,および,ピンポン経路におけるpiRNAの増幅,さらに,それらに付随したphased piRNAの生成など,piRNAの生成機構の大枠は保存されており,これらはゼブラフィッシュやカイコなどでも類似することが明らかにされている.
5.線虫におけるpiRNA経路
線虫のpiRNAはショウジョウバエやマウスより長さが短く,21塩基で1塩基目がウリジンではじまるものが多いことから21U-RNAとよばれている.線虫にはPRG-1およびPRG-2の2つのPIWIタンパク質が存在するが,piRNA経路においてはおもにPRG-1が機能する.ショウジョウバエやマウスのpiRNAとは異なり,21U-RNAはそれぞれが個別にゲノム(遺伝子間領域あるいはイントロン領域)にコードされており,約28~29塩基の短い21U-RNA前駆体がRNAポリメラーゼIIにより転写される91-93)(図8a).21U-RNAの遺伝子座の多くは転写ユニットの40塩基ほど上流にFKHモチーフをもち,転写因子であるFKHファミリータンパク質に依存的に転写される91,94).21U-RNA前駆体の生成に必要な因子としてTOFU-3,TOFU-4,TOFU-5,PRDE-1が同定されている92,93).このうち,PRDE-1はFKHファミリータンパク質に依存的な21U-RNA前駆体の生成にのみ関与する92).21U-RNA前駆体は21U-RNAの正確に2塩基上流から転写されることから,28~29塩基の21U-RNA前駆体は,キャップ構造が削除されたのち,さらに5'末端の2塩基が除去され,PRG-1に取り込まれると考えられている.この過程に必要な因子としてPID-1,TOFU-1,TOFU-2が同定されている93,95).PRG-1に取り込まれた21U-RNA前駆体は,PARNのホモログであるPARN-1により3'末端がトリミングされ96),メチル化酵素HENN-1により2'-O-メチル化修飾をうけ97-99),成熟した21U-RNAになる.さきに述べたように,線虫は独自のpiRNA経路をもっており,それを反映するように,線虫には保存されていないpiRNA因子も多く,さきにあげたpiRNAの生成にかかわる因子も線虫に特有のものである.
21U-RNAと相補的な配列をもつトランスポゾンはTc3しか知られておらず,21U-RNAの標的はながらく不明であったが,のちの解析により,21U-RNAはmiRNAのように数塩基のミスマッチを許容して標的RNAを認識することが明らかにされた100-102).標的の認識においては2~7塩基目のシード領域が重要であり,14~19塩基目の3'サプリメント領域の相補性も21U-RNAによる発現の抑制に寄与する101,102).また,9~13塩基目のセントラル領域のミスマッチが許容されるのと一致して,21U-RNA:PRG-1複合体による標的RNAの発現の抑制にはPRG-1のスライサー活性は必要でないことが示されている100,103).
これまでの研究から,21U-RNA:PRG-1複合体は,発現抑制のトリガーとしてのみ機能し,内在性の22G-siRNA経路を活性化することにより標的RNAの発現を抑制することが明らかにされている100,103).21U-RNA:PRG-1複合体は生殖細胞において発現する種々のRNAをスキャンし,標的RNAを認識するとRNA依存性のRNAポリメラーゼをリクルートすることにより2本鎖RNAを形成し,新たな22G-RNAの生成をひき起こす(図8b).この22G-RNAは線虫に特異的なAGOであるWAGOに取り込まれる.22G-RNA:WAGO複合体は相補的な配列をもつRNAを切断することによりその発現を抑制する.さらに,WAGOのひとつであるHRDE-1は22G-RNAと結合すると核へと移行し,NRDE-1,NRDE-2,NRDE-4に依存してヒストンH3のLys9のトリメチル化修飾を介して標的となる配列の転写を抑制する104,105).発現の抑制のシグナルがどのように伝達されるのか,その詳細は明らかにされていないが,核に局在するWAGOによる転写の抑制の効果は世代をこえてうけつがれ,いちど核において発現の抑制が確立されると,その維持には21U-RNAは必要ないことが示されている100,104-106)(文献106) は新着論文レビュー でも掲載).
21U-RNA:PRG-1複合体は数塩基のミスマッチを許容して標的RNAを認識するため,外来性の多様な配列にも対応する一方で,内在性のmRNAもその標的になりうる.これまでに,内在性のmRNAが21U-RNAによる発現の抑制を回避する2つの分子機構が報告されている(図8b).ひとつは,CSR-1の内在性のmRNAへの結合である.線虫の生殖細胞においては,WAGOにくわえ,CSR-1というAGOが発現する.CSR-1は内在性のmRNAを標的とする22G-RNAと結合するが,その発現は抑制しない.CSR-1が標的とする内在性のmRNAにおいては,21U-RNA:PRG-1複合体の結合および下流の22G-RNAの生成が抑制されることが示されており,21U-RNAの内在性のmRNAへの結合をCSR-1が阻止するというモデルが提唱されている101,106,107)(文献107) は新着論文レビュー でも掲載).また,線虫のゲノムにはperiodic An/Tn cluster(PATC)とよばれる,連続したAやTを周期的にくり返す線虫に特有の非コード配列が存在する.PATCはイントロンや遺伝子間領域に多くみられ,生殖細胞に発現する内在性の遺伝子はイントロンにおけるPATCの割合が高い傾向がある.さらに,イントロンにPATCをもつmRNAは,21U-RNAの標的となる配列が存在していても,21U-RNAによる発現の抑制をまぬがれることが示されている102).これらのことから,イントロンに存在するPATCがなんらかのかたちで自己のライセンスとしてはたらき,21U-RNAによる発現の抑制を回避すると考えられている.
おわりに
piRNAの発見から10年以上が経過した.次世代シークエンサー技術の発展とともにpiRNAの研究は大きく進展し,piRNA経路にかかわる因子の同定およびその生成機構や作用機序の大枠が明らかにされつつある.一方で,piRNA経路にかかわる個々の因子の分子機能や相互関係についての理解はいまだ途上にあり,さらなる解明が望まれる.このレビューにおいてはほとんど言及しなかったが,piRNAの機能は,トランスポゾンの発現の抑制にとどまらず,mRNAの発現や局在の制御,さらに,体細胞における機能へと大きな拡がりをみせている.また,がん細胞におけるPIWIやpiRNAの異所的な発現から,piRNA経路とがんとの関係も注目されている.今後,piRNAの生成機構および作用機序の理解がさらに進むとともに,体細胞におけるpiRNAの機能の解明,また,その制御のしくみが明らかにされることを期待したい.
文献
- Matsumoto, N., Nishimasu, H., Sakakibara, K. et al.: Crystal structure of silkworm PIWI-clade Argonaute Siwi bound to piRNA. Cell, 167, 484-497.e9 (2016)[PubMed] [新着論文レビュー]
- Kawaoka, S., Izumi, N., Katsuma, S. et al.: 3' end formation of PIWI-interacting RNAs in vitro. Mol. Cell, 43, 1015-1022 (2011)[PubMed] [新着論文レビュー]
- Simon, B., Kirkpatrick, J. P., Eckhardt, S. et al.: Recognition of 2'-O-methylated 3'-end of piRNA by the PAZ domain of a Piwi protein. Structure, 19, 172-180 (2011)[PubMed]
- Tian, Y., Simanshu, D. K., Ma, J. B. et al.: Structural basis for piRNA 2'-O-methylated 3'-end recognition by Piwi PAZ (Piwi/Argonaute/Zwille) domains. Proc. Natl. Acad. Sci. USA, 108, 903-910 (2011)[PubMed]
- Reuter, M., Berninger, P., Chuma, S. et al.: Miwi catalysis is required for piRNA amplification-independent LINE1 transposon silencing. Nature, 480, 264-267 (2011)[PubMed]
- De Fazio, S., Bartonicek, N., Di Giacomo, M. et al.: The endonuclease activity of Mili fuels piRNA amplification that silences LINE1 elements. Nature, 480, 259-263 (2011)[PubMed]
- Sienski, G., Donertas, D. & Brennecke, J.: Transcriptional silencing of transposons by Piwi and maelstrom and its impact on chromatin state and gene expression. Cell, 151, 964-80 (2012)[PubMed]
- Saito, K., Ishizu, H., Komai, M. et al.: Roles for the Yb body components Armitage and Yb in primary piRNA biogenesis in Drosophila. Genes Dev., 24, 2493-2498 (2010)[PubMed]
- Brennecke, J., Aravin, A. A., Stark, A. et al.: Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell, 128, 1089-1103 (2007)[PubMed]
- Robine, N., Lau, N. C., Balla, S. et al.: A broadly conserved pathway generates 3'UTR-directed primary piRNAs. Curr. Biol., 19, 2066-2076 (2009)[PubMed]
- Siomi, M. C., Mannen, T. & Siomi, H.: How does the royal family of Tudor rule the PIWI-interacting RNA pathway? Genes Dev., 24, 636-646 (2010)[PubMed]
- Kirino, Y., Kim, N., de Planell-Saguer, M. et al.: Arginine methylation of Piwi proteins catalysed by dPRMT5 is required for Ago3 and Aub stability. Nat. Cell Biol., 11, 652-658 (2009)[PubMed]
- Rangan, P., Malone, C. D., Navarro, C. et al.: piRNA production requires heterochromatin formation in Drosophila. Curr. Biol., 21, 1373-1379 (2011)[PubMed]
- Klattenhoff, C., Xi, H., Li, C. et al.: The Drosophila HP1 homolog Rhino is required for transposon silencing and piRNA production by dual-strand clusters. Cell, 138, 1137-1149 (2009)[PubMed]
- Andersen, P. R., Tirian, L., Vunjak, M. et al.: A heterochromatin-dependent transcription machinery drives piRNA expression. Nature, 549, 54-59 (2017)[PubMed]
- Mohn, F., Sienski, G., Handler, D. et al.: The Rhino-Deadlock-Cutoff complex licenses noncanonical transcription of dual-strand piRNA clusters in Drosophila. Cell, 157, 1364-1379 (2014)[PubMed]
- Zhang, Z., Wang, J., Schultz, N. et al.: The HP1 homolog Rhino anchors a nuclear complex that suppresses piRNA precursor splicing. Cell, 157, 1353-1363 (2014)[PubMed]
- Chen, Y. A., Stuwe, E., Luo, Y. et al.: Cutoff suppresses RNA polymerase II termination to ensure expression of piRNA precursors. Mol. Cell, 63, 97-109 (2016)[PubMed]
- Zhang, F., Wang, J., Xu, J. et al.: UAP56 couples piRNA clusters to the perinuclear transposon silencing machinery. Cell, 151, 871-884 (2012)[PubMed]
- Hur, J. K., Luo, Y., Moon, S. et al.: Splicing-independent loading of TREX on nascent RNA is required for efficient expression of dual-strand piRNA clusters in Drosophila. Genes Dev., 30, 840-855 (2016)[PubMed]
- Homolka, D., Pandey, R. R., Goriaux, C. et al.: PIWI slicing and RNA elements in precursors instruct directional primary piRNA biogenesis. Cell Rep., 12, 418-428 (2015)[PubMed]
- Ishizu, H., Iwasaki, Y. W., Hirakata, S. et al.: Somatic primary piRNA biogenesis driven by cis-acting RNA elements and trans-acting Yb. Cell Rep., 12, 429-440 (2015)[PubMed]
- Pandey, R. R., Homolka, D., Chen, K. M. et al.: Recruitment of Armitage and Yb to a transcript triggers its phased processing into primary piRNAs in Drosophila ovaries. PLoS Genet., 13, e1006956 (2017)[PubMed]
- Rogers, A. K., Situ, K., Perkins, E. M. et al.: Zucchini-dependent piRNA processing is triggered by recruitment to the cytoplasmic processing machinery. Genes Dev., 31, 1858-1869 (2017)[PubMed]
- Nishimasu, H., Ishizu, H., Saito, K. et al.: Structure and function of Zucchini endoribonuclease in piRNA biogenesis. Nature, 491, 284-287 (2012)[PubMed] [新着論文レビュー]
- Ipsaro, J. J., Haase, A. D., Knott, S. R. et al.: The structural biochemistry of Zucchini implicates it as a nuclease in piRNA biogenesis. Nature, 491, 279-283 (2012)[PubMed]
- Han, B. W., Wang, W., Li, C. et al.: piRNA-guided transposon cleavage initiates Zucchini-dependent, phased piRNA production. Science, 348, 817-821 (2015)[PubMed]
- Mohn, F., Handler, D. & Brennecke, J.: piRNA-guided slicing specifies transcripts for Zucchini-dependent, phased piRNA biogenesis. Science, 348, 812-817 (2015)[PubMed]
- Olivieri, D., Senti, K. A., Subramanian, S. et al.: The cochaperone Shutdown defines a group of biogenesis factors essential for all piRNA populations in Drosophila. Mol. Cell, 47, 954-969 (2012)[PubMed]
- Preall, J. B., Czech, B., Guzzardo, P. M. et al.: shutdown is a component of the Drosophila piRNA biogenesis machinery. RNA, 18, 1446-1457 (2012)[PubMed]
- Saito, K., Sakaguchi, Y., Suzuki, T. et al.: Pimet, the Drosophila homolog of HEN1, mediates 2'-O-methylation of Piwi- interacting RNAs at their 3' ends. Genes Dev., 21, 1603-1608 (2007)[PubMed]
- Horwich, M. D., Li, C., Matranga, C. et al.: The Drosophila RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC. Curr. Biol., 17, 1265-1272 (2007)[PubMed]
- Handler, D., Olivieri, D., Novatchkova, M. et al.: A systematic analysis of Drosophila TUDOR domain-containing proteins identifies Vreteno and the Tdrd12 family as essential primary piRNA pathway factors. EMBO J., 30, 3977-3993 (2011)[PubMed]
- Vagin, V. V., Yu, Y., Jankowska, A. et al.: Minotaur is critical for primary piRNA biogenesis. RNA, 19, 1064-1077 (2013)[PubMed]
- Handler, D., Meixner, K., Pizka, M. et al.: The genetic makeup of the Drosophila piRNA pathway. Mol. Cell, 50, 762-777 (2013)[PubMed]
- Czech, B., Preall, J. B., McGinn, J. et al.: A transcriptome-wide RNAi screen in the Drosophila ovary reveals factors of the germline piRNA pathway. Mol. Cell, 50, 749-761 (2013)[PubMed]
- Sienski, G., Batki, J., Senti, K. A. et al.: Silencio/CG9754 connects the Piwi-piRNA complex to the cellular heterochromatin machinery. Genes Dev., 29, 2258-2271 (2015)[PubMed]
- Yu, Y., Gu, J., Jin, Y. et al.: Panoramix enforces piRNA-dependent cotranscriptional silencing. Science, 350, 339-342 (2015)[PubMed]
- Muerdter, F., Guzzardo, P. M., Gillis, J. et al.: A genome-wide RNAi screen draws a genetic framework for transposon control and primary piRNA biogenesis in Drosophila. Mol. Cell, 50, 736-748 (2013)[PubMed]
- Donertas, D., Sienski, G. & Brennecke, J.: Drosophila Gtsf1 is an essential component of the Piwi-mediated transcriptional silencing complex. Genes Dev., 27, 1693-1705 (2013)[PubMed]
- Ohtani, H., Iwasaki, Y. W., Shibuya, A. et al.: DmGTSF1 is necessary for Piwi-piRISC-mediated transcriptional transposon silencing in the Drosophila ovary. Genes Dev., 27, 1656-1661 (2013)[PubMed]
- Iwasaki, Y. W., Murano, K., Ishizu, H. et al.: Piwi modulates chromatin accessibility by regulating multiple factors including histone H1 to repress transposons. Mol. Cell, 63, 408-419 (2016)[PubMed] [新着論文レビュー]
- Teixeira, F. K., Okuniewska, M., Malone, C. D. et al.: piRNA-mediated regulation of transposon alternative splicing in the soma and germ line. Nature, 552, 268-272 (2017)[PubMed]
- Gunawardane, L. S., Saito, K., Nishida, K. M. et al.: A slicer-mediated mechanism for repeat-associated siRNA 5' end formation in Drosophila. Science, 315, 1587-1590 (2007)[PubMed]
- Sato, K., Iwasaki, Y. W., Shibuya, A. et al.: Krimper enforces an antisense bias on piRNA pools by binding AGO3 in the Drosophila germline. Mol. Cell, 59, 553-563 (2015)[PubMed] [新着論文レビュー]
- Webster, A., Li, S., Hur, J. K. et al.: Aub and Ago3 are recruited to nuage through two mechanisms to form a Ping-Pong complex assembled by Krimper. Mol. Cell, 59, 564-575 (2015)[PubMed]
- Zhang, Z., Xu, J., Koppetsch, B. S. et al.: Heterotypic piRNA Ping-Pong requires qin, a protein with both E3 ligase and Tudor domains. Mol. Cell, 44, 572-584 (2011)[PubMed]
- Xiol, J., Spinelli, P., Laussmann, M. A. et al.: RNA clamping by Vasa assembles a piRNA amplifier complex on transposon transcripts. Cell, 157, 1698-1711 (2014)[PubMed]
- Nishida, K. M., Iwasaki. Y. W., Murota, Y. et al.: Respective functions of two distinct Siwi complexes assembled during PIWI-interacting RNA biogenesis in Bombyx germ cells. Cell Rep., 10, 193-203 (2015)[PubMed]
- Hayashi, R., Schnabl, J., Handler, D. et al.: Genetic and mechanistic diversity of piRNA 3'-end formation. Nature, 539, 588-592 (2016)[PubMed]
- Wang, W., Yoshikawa, M., Han, B. W. et al.: The initial uridine of primary piRNAs does not create the tenth adenine that is the hallmark of secondary piRNAs. Mol. Cell, 56, 708-716 (2014)[PubMed]
- Brennecke, J., Malone, C. D., Aravin, A. A. et al.: An epigenetic role for maternally inherited piRNAs in transposon silencing. Science, 322, 1387-1392 (2008)[PubMed]
- Kuramochi-Miyagawa, S., Kimura, T., Ijiri, T. W. et al.: Mili, a mammalian member of piwi family gene, is essential for spermatogenesis. Development, 131, 839-849 (2004)[PubMed]
- Aravin, A. A., Sachidanandam, R., Bourc'his, D. et al.: A piRNA pathway primed by individual transposons is linked to de novo DNA methylation in mice. Mol. Cell, 31, 785-799 (2008)[PubMed]
- Deng, W. & Lin, H.: miwi, a murine homolog of piwi, encodes a cytoplasmic protein essential for spermatogenesis. Dev. Cell, 2, 819-830 (2002)[PubMed]
- Carmell, M. A., Girard, A., van de Kant, H. J. et al.: MIWI2 is essential for spermatogenesis and repression of transposons in the mouse male germline. Dev. Cell, 12, 503-514 (2007)[PubMed]
- Saxe, J. P., Chen, M., Zhao, H. et al.: Tdrkh is essential for spermatogenesis and participates in primary piRNA biogenesis in the germline. EMBO J., 32, 1869-1885 (2013)[PubMed]
- Tanaka, S. S., Toyooka, Y., Akasu, R. et al.: The mouse homolog of Drosophila Vasa is required for the development of male germ cells. Genes Dev., 14, 841-853 (2000)[PubMed]
- Huang, H., Gao, Q., Peng, X. et al.: piRNA-associated germline nuage formation and spermatogenesis require MitoPLD profusogenic mitochondrial-surface lipid signaling. Dev. Cell, 20, 376-387 (2011)[PubMed]
- Watanabe, T., Chuma, S., Yamamoto, Y. et al.: MITOPLD is a mitochondrial protein essential for nuage formation and piRNA biogenesis in the mouse germline. Dev. Cell, 20, 364-375 (2011)[PubMed] [新着論文レビュー]
- Shoji, M., Tanaka, T., Hosokawa, M. et al.: The TDRD9-MIWI2 complex is essential for piRNA-mediated retrotransposon silencing in the mouse male germline. Dev. Cell, 17, 775-787 (2009)[PubMed]
- Pandey, R. R., Tokuzawa, Y., Yang, Z. et al.: Tudor domain containing 12 (TDRD12) is essential for secondary PIWI interacting RNA biogenesis in mice. Proc. Natl. Acad. Sci. USA, 110, 16492-16497 (2013)[PubMed]
- Ma, L., Buchold, G. M., Greenbaum, M. P. et al.: GASZ is essential for male meiosis and suppression of retrotransposon expression in the male germline. PLoS Genet., 5, e1000635 (2009)[PubMed]
- Zheng, K., Xiol, J., Reuter, M. et al.: Mouse MOV10L1 associates with Piwi proteins and is an essential component of the Piwi-interacting RNA (piRNA) pathway. Proc. Natl. Acad. Sci. USA, 107, 11841-11846 (2010)[PubMed]
- Pan, J., Goodheart, M., Chuma, S. et al.: RNF17, a component of the mammalian germ cell nuage, is essential for spermiogenesis. Development, 132, 4029-4039 (2005)[PubMed]
- Tanaka, T., Hosokawa, M., Vagin, V. V. et al.: Tudor domain containing 7 (Tdrd7) is essential for dynamic ribonucleoprotein (RNP) remodeling of chromatoid bodies during spermatogenesis. Proc. Natl. Acad. Sci. USA, 108, 10579-10584 (2011)[PubMed]
- Yabuta, Y., Ohta, H., Abe, T. et al.: TDRD5 is required for retrotransposon silencing, chromatoid body assembly, and spermiogenesis in mice. J. Cell Biol., 192, 781-795 (2011)[PubMed]
- Chuma, S., Hosokawa, M., Kitamura, K. et al.: Tdrd1/Mtr-1, a tudor-related gene, is essential for male germ-cell differentiation and nuage/germinal granule formation in mice. Proc. Natl. Acad. Sci. USA, 103, 15894-15899 (2006)[PubMed]
- Aravin, A. A., Sachidanandam, R., Girard, A. et al.: Developmentally regulated piRNA clusters implicate MILI in transposon control. Science, 316, 744-747 (2007)[PubMed]
- Vourekas, A., Zheng, K., Fu, Q. et al.: The RNA helicase MOV10L1 binds piRNA precursors to initiate piRNA processing. Genes Dev., 29, 617-629 (2015)[PubMed]
- Izumi, N., Shoji, K., Sakaguchi, Y. et al.: Identification and functional analysis of the pre-piRNA 3' trimmer in silkworms. Cell, 164, 962-973 (2016)[PubMed] [新着論文レビュー]
- Nishimura, T., Nagamori, I., Nakatani, T. et al.: PNLDC1, mouse pre-piRNA Trimmer, is required for meiotic and post-meiotic male germ cell development. EMBO Rep., 19, e44957 (2018)[PubMed]
- Ding, D., Liu, J., Dong, K. et al.: PNLDC1 is essential for piRNA 3' end trimming and transposon silencing during spermatogenesis in mice. Nat. Commun., 8, 819 (2017)[PubMed]
- Zhang, Y., Guo, R., Cui, Y. et al.: An essential role for PNLDC1 in piRNA 3' end trimming and male fertility in mice. Cell Res., 27, 1392-1396 (2017)[PubMed]
- Kirino, Y. & Mourelatos, Z.: The mouse homolog of HEN1 is a potential methylase for Piwi-interacting RNAs. RNA, 13, 1397-1401 (2007)[PubMed]
- Shiromoto, Y., Kuramochi-Miyagawa, S., Daiba, A. et al.: GPAT2, a mitochondrial outer membrane protein, in piRNA biogenesis in germline stem cells. RNA, 19, 803-810 (2013)[PubMed]
- Yang, Z., Chen, K. M., Pandey, R. R. et al.: PIWI slicing and EXD1 drive biogenesis of nuclear piRNAs from cytosolic targets of the mouse piRNA pathway. Mol. Cell, 61, 138-152 (2016)[PubMed]
- Kuramochi-Miyagawa, S., Watanabe, T., Gotoh, K. et al.: DNA methylation of retrotransposon genes is regulated by Piwi family members MILI and MIWI2 in murine fetal testes. Genes Dev., 22, 908-917 (2008)[PubMed]
- Manakov, S. A., Pezic, D., Marinov, G. K. et al.: MIWI2 and MILI have differential effects on piRNA biogenesis and DNA methylation. Cell Rep., 12, 1234-1243 (2015)[PubMed]
- Aravin, A. A., van der Heijden, G. W., Castaneda, J. et al.: Cytoplasmic compartmentalization of the fetal piRNA pathway in mice. PLoS Genet., 5, e1000764 (2009)[PubMed]
- Wenda, J. M., Homolka, D., Yang, Z. et al.: Distinct roles of RNA helicases MVH and TDRD9 in PIWI slicing-triggered mammalian piRNA biogenesis and function. Dev. Cell, 41, 623-637.e9 (2017)[PubMed]
- Kuramochi-Miyagawa, S., Watanabe, T., Gotoh, K. et al.: MVH in piRNA processing and gene silencing of retrotransposons. Genes Dev., 24, 887-892 (2010)[PubMed]
- Yoshimura, T., Watanabe, T., Kuramochi-Miyagawa, S. et al.: Mouse GTSF1 is an essential factor for secondary piRNA biogenesis. EMBO Rep., 19, e42054 (2018)[PubMed]
- Xiol, J., Cora, E., Koglgruber, R. et al.: A role for Fkbp6 and the chaperone machinery in piRNA amplification and transposon silencing. Mol. Cell, 47, 970-979 (2012)[PubMed]
- Li, X. Z., Roy, C. K., Dong, X. et al.: An ancient transcription factor initiates the burst of piRNA production during early meiosis in mouse testes. Mol. Cell, 50, 67-81 (2013)[PubMed]
- Zhou, L., Canagarajah, B., Zhao, Y. et al.: BTBD18 regulates a subset of piRNA-generating loci through transcription elongation in mice. Dev. Cell, 40, 453-466.e5 (2017)[PubMed]
- Wasik, K. A., Tam, O. H., Knott, S. R. et al.: RNF17 blocks promiscuous activity of PIWI proteins in mouse testes. Genes Dev., 29, 1403-1415 (2015)[PubMed]
- Zhang, P., Kang, J. Y., Gou, L. T. et al.: MIWI and piRNA-mediated cleavage of messenger RNAs in mouse testes. Cell Res., 25, 193-207 (2015)[PubMed]
- Gou, L. T., Dai, P., Yang, J. H. et al.: Pachytene piRNAs instruct massive mRNA elimination during late spermiogenesis. Cell Res., 24, 680-700 (2014)[PubMed]
- Kiuchi, T., Koga, H., Kawamoto, M. et al.: A single female-specific piRNA is the primary determiner of sex in the silkworm. Nature, 509, 633-636 (2014)[PubMed] [新着論文レビュー]
- Ruby, J. G., Jan, C., Player, C. et al.: Large-scale sequencing reveals 21U-RNAs and additional microRNAs and endogenous siRNAs in C. elegans. Cell, 127, 1193-1207 (2006)[PubMed]
- Weick, E. M., Sarkies, P., Silva, N. et al.: PRDE-1 is a nuclear factor essential for the biogenesis of Ruby motif-dependent piRNAs in C. elegans. Genes Dev., 28, 783-796 (2014)[PubMed]
- Goh, W. S., Seah, J. W., Harrison, E. J. et al.: A genome-wide RNAi screen identifies factors required for distinct stages of C. elegans piRNA biogenesis. Genes Dev., 28, 797-807 (2014)[PubMed]
- Cecere, G., Zheng, G. X., Mansisidor, A. R. et al.: Promoters recognized by forkhead proteins exist for individual 21U-RNAs. Mol. Cell, 47, 734-745 (2012)[PubMed]
- de Albuquerque, B. F., Luteijn, M. J., Cordeiro Rodrigues, R. J. et al.: PID-1 is a novel factor that operates during 21U-RNA biogenesis in Caenorhabditis elegans. Genes Dev., 28, 683-688 (2014)[PubMed]
- Tang, W., Tu, S., Lee, H. C. et al.: The RNase PARN-1 trims piRNA 3' ends to promote transcriptome surveillance in C. elegans. Cell, 164, 974-984 (2016)[PubMed]
- Kamminga, L. M., van Wolfswinkel, J. C., Luteijn, M. J. et al.: Differential impact of the HEN1 homolog HENN-1 on 21U and 26G RNAs in the germline of Caenorhabditis elegans. PLoS Genet., 8, e1002702 (2012)[PubMed]
- Billi, A. C., Alessi, A. F., Khivansara, V. et al.: The Caenorhabditis elegans HEN1 ortholog, HENN-1, methylates and stabilizes select subclasses of germline small RNAs. PLoS Genet., 8, e1002617 (2012)[PubMed]
- Montgomery, T. A., Rim, Y. S., Zhang, C. et al.: PIWI associated siRNAs and piRNAs specifically require the Caenorhabditis elegans HEN1 ortholog henn-1. PLoS Genet., 8, e1002616 (2012)[PubMed]
- Lee, H. C., Gu, W., Shirayama, M. et al.: C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell, 150, 78-87 (2012)[PubMed]
- Shen, E. Z., Chen, H., Ozturk, A. R. et al.: Identification of piRNA binding sites reveals the Argonaute regulatory landscape of the C. elegans germline. Cell, 172, 937-951.e18 (2018)[PubMed]
- Zhang, D., Tu, S., Stubna, M. et al.: The piRNA targeting rules and the resistance to piRNA silencing in endogenous genes. Science, 359, 587-592 (2018)[PubMed]
- Bagijn, M. P., Goldstein, L. D., Sapetschnig, A. et al.: Function, targets, and evolution of Caenorhabditis elegans piRNAs. Science, 337, 574-578 (2012)[PubMed]
- Ashe, A., Sapetschnig, A., Weick, E. M. et al.: piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell, 150, 88-99 (2012)[PubMed]
- Buckley, B. A., Burkhart, K. B., Gu, S. G. et al.: A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality. Nature, 489, 447-451 (2012)[PubMed]
- Shirayama, M., Seth, M., Lee, H. C. et al.: piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell, 150, 65-77 (2012)[PubMed] [新着論文レビュー]
- Seth, M., Shirayama, M., Gu, W. et al.: The C. elegans CSR-1 Argonaute pathway counteracts epigenetic silencing to promote germline gene expression. Dev. Cell, 27, 656-663 (2013)[PubMed] [新着論文レビュー]
著者プロフィール
略歴:2009年 横浜市立大学大学院医学研究科博士課程 修了,2010年 東京大学分子細胞生物学研究所 研究員を経て,2014年より同 助教(現 東京大学定量生命科学研究所 助教).
研究テーマ:piRNAの生成機構.
泊 幸秀(Yukihide Tomari)
東京大学定量生命科学研究所 教授.
研究室URL:http://www.iam.u-tokyo.ac.jp/tomari/
© 2018 泉 奈津子・泊 幸秀 Licensed under CC 表示 2.1 日本