植物において減数分裂期に高発現するphasiRNAの産生経路および機能
2019/01/23
小野聖二郎・野々村賢一
(国立遺伝学研究所 植物細胞遺伝研究室)
email:野々村賢一
領域融合レビュー, 8, e002 (2019) DOI: 10.7875/leading.author.8.e002
Seijiro Ono & Ken-Ichi Nonomura: Biogenesis and function of plant reproductive phasiRNAs abundantly expressed in meiotic stages.

減数分裂は,両親に由来する染色体から新たな遺伝子の組合せを創出し,染色体の数や構造を監視する役割を担う,遺伝や育種の根幹をなす生命現象である.これまでに,不対合領域に分布する遺伝子の発現抑制や,piRNAによる生殖細胞系列におけるトランスポゾンの抑制など,生殖細胞の発生や減数分裂の進行と小分子RNAに依存的なエピジェネティックな機構との関連性が多くのモデル生物において報告されている.植物にpiRNAは保存されていないが,近年,単子葉植物の生殖器官において高発現する21塩基長および24塩基長のsiRNAとしてphasiRNAが発見され,生殖細胞の初期の発生を制御する可能性のある小分子RNAとして注目されている.phasiRNAの前駆体となるlncRNAは,植物のゲノムにおいて数百か所から数千か所の遺伝子領域あるいは遺伝子間領域に分布する.前駆体RNAがRNAポリメラーゼIIにより転写されたのち,22塩基長のmiRNAとArgonauteとの複合体による1次切断,RNA依存的RNAポリメラーゼによる2本鎖化,リボヌクレアーゼIII様酵素であるDCLによるmiRNAによる切断部位を起点とした21塩基長あるいは24塩基長の間隔での切断をへて,成熟phasiRNAが産生される.このレビューにおいては,おもに減数分裂期に高発現するphasiRNAについて,産生経路や発現の時空間的な制御,相互作用するArgonauteの種類および機能などを解説し,動物におけるpiRNAの産生経路との比較をつうじ,植物に特徴的な生殖システムをあぶりだすことを試みる.
減数分裂は,有性生殖を行うすべての真核生物に保存される,生殖の過程における中心的なイベントのひとつである.減数分裂における組換えは両親に由来する染色体から新たな遺伝子の組合せを創出し次世代へと伝達する.さらに,減数分裂は相同染色体の対合をつうじ染色体の数および構造の監視も担う.たとえば,ヒョウとライオンの1代雑種であるレオポンに生殖能力がないのは,両親の染色体の数および構造の違いによる減数分裂における染色体の対合の異常が一因とされる.このように,減数分裂は種内の遺伝的な多様化に機能するとともに,染色体の数および構造の認識をつうじ種間の遺伝子の交換を制限し種の維持に貢献する.
このレビューにおけるおもなトピックである小分子RNAと減数分裂との関連が知られた最初のきっかけは,アカパンカビの不対合領域における遺伝子の発現抑制の研究であり1,2),マウスにおいても類似の現象が報告された3,4).植物においては,最近,生殖の過程において機能する小分子RNAに関する報告があいついだ.とくに,花粉において先駆的な研究が多いが,それについてはほかのすぐれたレビューを参照いただき5),このレビューにおいては,減数分裂期に高発現する小分子RNAのひとつphasiRNAの産生経路について,植物に特徴的な生殖システムの説明をおりまじえて解説し,動物のpiRNAとの比較から,いまだに多くの謎を残す植物のphasiRNAの役割について考察する.
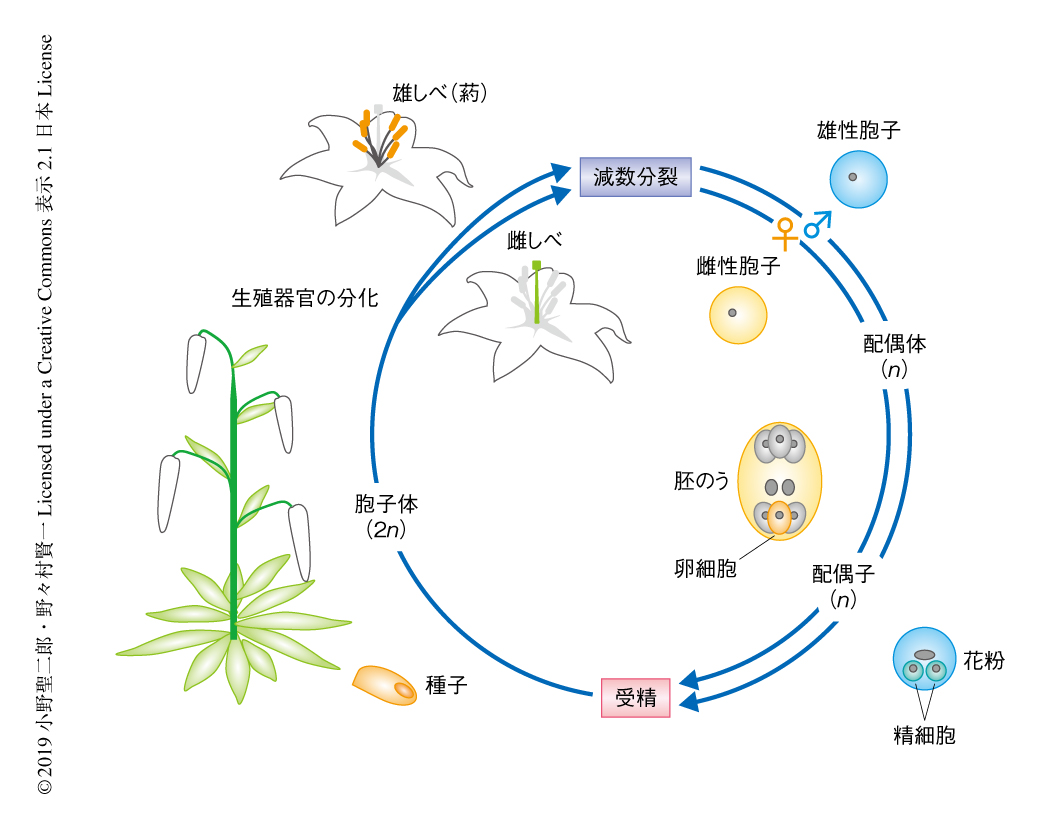
“世代”という言葉の生殖生物学における真の意味をご存知だろうか? 生殖生物学においては,親子世代といった一般的な用法とは異なり,同一の生活環に出現する異なる生殖様式のそれぞれをさす6,7).ヒトも含め多くの動物においては,減数分裂により生じた細胞はそのまま配偶子へと成熟するため,動物の体は配偶子を生み出す配偶体と定義され,生活環に現われる生殖世代は配偶体(有性生殖)世代のみである.一方,陸上植物の減数分裂により生じた細胞(胞子)は,体細胞分裂をへて多細胞の配偶体(被子植物においては,花粉および胚のう)となり,その内部に配偶子を形成する(図1).配偶体は,動物および植物においてともに有性生殖世代に分類される.一方,われわれが通常目にする被子植物の体は,配偶体(胞子に由来)を無性的につくりだす体,すなわち,胞子体として無性生殖世代に定義される.このように,陸上植物は無性と有性の2種類の生殖世代が同一の生活環に交互に現われる世代交代をするという特徴をもつ.つまり,生殖様式の観点から,動物の生殖細胞の発生と比較される植物の発生の過程は,減数分裂ののちから受精にかけての配偶体の形成の過程であり,陸上植物にとり,減数分裂は無性生殖世代の最後のイベントである.
このレビューにおける焦点である生殖に特異的な小分子RNAとして,動物においてはpiRNA(PIWI-interacting RNA)が同定され,さかんに研究されている.piRNAは,生殖細胞系列においてトランスポゾンを不活性化し完全なゲノムを次世代に伝達することが一義的な役割と考えられている.このようなシステムはすべての生物に不可欠なはずだが,植物はpiRNAの産生経路に類似した小分子RNAの産生経路,および,piRNAと結合するPIWIファミリーに属するArgonaute(AGO)を保存していない8-10).この事実は,植物に特有の生殖世代を反映しているのかもしれない.その一方で,植物において減数分裂細胞およびその原基細胞には,減数分裂あるいはそののちの有性生殖(配偶体の形成)の運命を左右する遺伝子,および,小分子RNAを含む非コードRNAが多く発現しており11-13),それらがpiRNAに相当する機能を担う可能性は十分に考えられる.
小分子RNAは20~30塩基長のRNAであり,ガイドRNAとしてArgonauteに取り込まれてRISC(RNA-induced silencing complex)とよばれる複合体を形成し,配列に依存的に転写後あるいは転写のレベルにおいて遺伝子の発現抑制に機能する.転写後抑制においては,RISCが標的となるmRNAと結合して切断や翻訳の抑制を行う.転写抑制は,RISCが標的となるクロマチン領域にRNAあるいはDNAを介して結合し,その領域のクロマチンに修飾をひき起こす機構である.植物における代表的な転写抑制としてRNA依存的DNAメチル化が知られている14).一般に,転写後抑制は細胞質に局在するArgonauteが,転写抑制は核に局在するArgonauteが,それぞれ実行する15).動物の小分子RNAは,miRNA(microRNA),siRNA(small interfering RNA),そして,生殖器官において機能するpiRNAの3つに大別される.とくに,piRNAを含む小分子RNAの産生については,泉 奈津子・泊 幸秀, 領域融合レビュー, 7, e003 (2018) を参照されたい.
植物はpiRNAをもたない一方で,phasiRNA(phased secondary siRNA)とよばれる独自のsiRNAを発達させた.phasiRNAは,遺伝子領域あるいは遺伝子間領域からRNAポリメラーゼIIにより転写されるlncRNA(long non-coding RNA)を前駆体とする10)(図2a).この前駆体RNAをPHAS RNA,由来するゲノム領域をPHAS座とよぶ.phasiRNAの産生は,PHAS RNAの5’末端あるいは両方の末端に保存される標的配列を,22塩基長のmiRNAとAGO1あるいはAGO7との複合体が切断する1次切断にはじまる.切断された1本鎖のPHAS RNAは,RNA依存的RNAポリメラーゼであるRDR6により2本鎖化される.そののち,miRNAの切断部位を起点として,リボヌクレアーゼIII様の酵素であるDCL4あるいはDCL5が,2本鎖のPHAS RNAを21塩基長(DCL4)あるいは24塩基長(DCL5)の等間隔で2次切断することにより,phasiRNAが産生される.
phasiRNAは,PHAS座のセンス鎖およびアンチセンス鎖の両方に,21塩基長あるいは24塩基長の位相をもってマップされている.よくタンデムリピートと誤解されるが,同一のPHAS座においてもphasiRNAのあいだに配列の保存性はなく,それぞれがユニークな配列としてマップされる場合が多い.24塩基長のphasiRNAの前駆体をコードする24-PHAS座はイネのゲノムに100座以上,21塩基長のphasiRNAの前駆体をコードする21-PHAS座にいたっては1000座以上が同定されている16-18)(図2b).同様のパターンを示すものにtasiRNA(trans-acting siRNA)がある.tasiRNAは植物のjuvenile-adult相転換を含む発生の過程において重要な役割をはたすsiRNAとして,以前に報告されていた19).その産生経路はphasiRNAと類似するが,tasiRNAの産生経路においては前駆体となるlncRNAが約10種類と少なく,特定の遺伝子のmRNAにトランスに作用する20).phasiRNAはシスおよびトランスの両方で作用するsiRNAを含む用語であることから,tasiRNAをphasiRNAの一種として定義することが提案されている10).
phasiRNAは花序組織に豊富に存在するsiRNAとして,イネにおいて最初に報告された16).そののち,花序のうちとくに雄しべ(葯)において豊富に発現すること,1次切断がAGO1および22塩基長のmiRNA(miR2118あるいはmiR2275),2次切断がRDR6およびDCL4あるいはDCL5に依存することが報告され21,22),phasiRNAとして定義された.なお,生殖器官以外にも発現するphasiRNAの報告があるが,とくに単子葉植物では生殖器官において高い発現を示す場合が多いため,このレビューでは,とくに断らないかぎり生殖phasiRNA(reproductive phasiRNA)をphasiRNAとよぶ.
トウモロコシおよびイネにおいては,葯の発達段階にそったphasiRNAの時空間的な発現パターンが明らかにされている23,24).21塩基長のphasiRNAは減数分裂のまえから減数分裂期にかけて葯に豊富に発現する一方,24塩基長のphasiRNAは減数分裂期においてピンポイントに発現する(図3).イネの減数分裂期の葯において検出される小分子RNAのうち,トランスポゾンをふくむくり返し配列に由来するsiRNAが全体の約75%をしめるが,それにつづいて21塩基長のphasiRNAおよび24塩基長のphasiRNAがそれぞれ15%および6%存在する18).
葉などの表皮に発現するHD-ZIP IVファミリーの転写因子であるOCL4の機能欠損変異体の葯において,21塩基長のphasiRNAの発現量が劇的に減少したことから,21塩基長のphasiRNAおよびその前駆体は葯の表皮にて発現する可能性が示された23).24塩基長のphasiRNAは,葯のうち雄性の減数分裂細胞をとりかこむタペート層とよばれる体細胞組織において特異的に発現することが示された18,23)(図3).タペート細胞は減数分裂ののちに形成される胞子に花粉殻の材料や栄養を供給するが,減数分裂期における機能は不明であった.のちに述べるように,最近,筆者らの研究グループにより,タペート層において特異的に発現し機能するbHLH型の転写因子が,減数分裂に特異的な24塩基長のphasiRNAの産生を活性化することが明らかにされた.
生殖phasiRNAは,イネやトウモロコシにくわえ,近年では,カモジグサ25,26),ユリ,アスパラガス27) などの単子葉植物においてあいついで報告されている.一方,マメ科など双子葉植物においては,栄養成長期に特定の遺伝子領域からphasiRNAが産生されるが,生殖器官における豊富な発現は報告されておらず,また,代表的なモデル植物であるシロイヌナズナにおいてはtasiRNA以外に明瞭なphasiRNAは検出されないため28),生殖phasiRNAの研究はおもに単子葉植物において進んでいる.
生殖phasiRNAの分子機能についてはいまだによくわかっていない.phasiRNAの配列の種類が膨大であることが標的となるRNAの予測がむずかしい一因である.イネのゲノムに21-PHAS座は1000以上も存在し16,17),そこからは合計で2万種類以上のphasiRNAの産生が見込まれる.24-PHAS座は100座あまりだが18),それでも見込まれる24塩基長のphasiRNAの種類は膨大である.標的となるRNAのゲノムワイドな予測29) や,機械学習による予測30) も試みられているが,phasiRNAの配列からは標的となるRNAの明瞭な同定にはいたっていない.また,ほかのsiRNAと比較して,phasiRNAはユニークな遺伝子間領域に由来する傾向が示された18,23,30).phasiRNAのこのような性質は,RNAポリメラーゼIVに依存的なsiRNA や31),epigenetically activated siRNA(easiRNA)など32),特定の遺伝子あるいはトランスポゾンを標的とする植物のほかのsiRNAの性質とは一線を画す.
これらの知見を総合すると,phasiRNAは核において自らの由来する領域,すなわち,PHAS座の転写抑制に作用する可能性がきわめて高い.事実,トウモロコシの21-PHAS座および24-PHAS座の周辺は,葯および雄性減数分裂細胞(花粉母細胞)におけるDNAのメチル化,とくにCHHメチル化(H:A,T,G)が成葉などの栄養成長器官より高い傾向が示されている33).植物のsiRNAは,RNA依存的DNAメチル化に代表される転写抑制に機能することが知られており14),phasiRNAも生殖の過程において同様の経路に組み込まれている可能性は十分に考えられる(図4).
phasiRNAの“secondary”とは,miRNAによる1次切断をへてDCLによる2次切断により産生されることにちなむ.しかし,イネやトウモロコシの21-PHAS座のなかには,miR2118による1次切断部位からはじまる位相におさまるphasiRNAのほか,その位相から9塩基ずれた位相をもつsiRNAが多く見い出される34).これらは,phasiRNAによる21-PHAS RNAの3次切断が起こり,そこからさらにDCL4による連続切断がはじまると考えるとうまく説明される.すなわち,phasiRNAは転写制御への関与の可能性にくわえ,PHAS RNAを標的とした転写後抑制にかかわり,それ自体の増幅サイクルを促進する可能性が提唱されている(図4).
Argonauteは,シロイヌナズナにおいて10種類,イネにおいて19種類,トウモロコシにおいて17種類が保存され,それらはAGO1/5/10ファミリー,AGO2/3/7ファミリー,AGO4/6/9ファミリーに分類される35).生殖の過程における関与の知られるArgonauteはAGO4/6/9ファミリーおよびAGO1/5/10ファミリーに多く含まれるが(phasiRNAの産生において1次切断を実行するArgonauteは除く),それらはいずれも動物のPIWIファミリーには属さない.
シロイヌナズナのAGO9(AtAGO9)は,おもにトランスポゾンに由来する24塩基長のsiRNAと結合する36).AtAGO9は雌性減数分裂細胞の核および雌性減数分裂細胞をつつむ珠心表皮の細胞質に局在し36,37),隣接する体細胞が雌性減数分裂細胞になることを抑制する36).トウモロコシのAGO104(ZmAGO104)はAtAGO9のホモログであり,変異体の葯においては減数分裂の停止や減数分裂染色体の脱凝縮などの異常が,また,雌しべの側では減数分裂の異常にともなう非還元性の配偶子の形成が観察される38).シロイヌナズナのAGO4(AtAGO4)は生活環をつうじて恒常的に発現するが,変異体においては花粉母細胞における減数分裂染色体の脱凝縮,分配の遅滞,架橋などの異常が起こる39).AtAGO4,AtAGO9,ZmAGO104はいずれもAGO4/6/9ファミリーに属し,とくにAtAGO4はRNA依存的DNAメチル化経路において中心的な役割を担うことから,減数分裂とRNA依存的DNAメチル化経路との密接な関連性が示唆される.
イネMEL1(OsMEL1)は,雌雄の胞子原基細胞と胞子形成細胞(いずれも,減数分裂細胞に分化する細胞)および減数分裂細胞において特異的に発現するArgonauteであり,その変異体においては減数分裂染色体の凝縮が早期に停止して完全不稔になる8).OsMEL1のオーソログであるシロイヌナズナのAGO5(AtAGO5)については,花粉の精細胞および珠心表皮における発現が報告されている40,41).しかし,AtAGO5のドミナントネガティブ変異体は減数分裂の直後において胞子の発生が異常となるが,機能欠損型変異体は減数分裂において顕著な表現型を示さない41).AGO1/5/10ファミリーに属するイネの5つのArgonauteのうち,OsMEL1は葯および雌しべ,OsAGO12は葯に特異的に,OsAGO14は雌しべに特異的に発現するなど9),AGO1/5/10ファミリーは生殖に特異的に機能するArgonauteとして機能が分化した可能性が高い.
現時点では,生殖phasiRNAとの結合が明確に示されたArgonauteはイネにおけるOsMEL1のみである.OsMEL1は5’末端がCであるsiRNAと結合する傾向を示す17).減数分裂よりまえから減数分裂にかけて,おもに21塩基長のphasiRNAと結合し,その割合は結合するすべての小分子RNAの約60%におよぶ17,18).OsMEL1は減数分裂期に24塩基長のphasiRNAとも結合するが,結合するすべての小分子RNAの1%未満である18).この結果から,OsMEL1は5’末端がCである21塩基長のphasiRNAに高い嗜好性をもつ可能性が示唆される.しかし,これは葯において発現するsiRNAの割合を反映した結果かもしれず,事実,OsMEL1と結合する小分子RNAにはトランスポゾンをふくむくり返し配列に由来するsiRNAも20%程度含まれており17,18),phasiRNA以外のsiRNAとOsMEL1との結合の重要性も否定できない.
根の放射状パターンや葉の表裏パターンの形成において,小分子RNAは細胞のあいだを移動し,細胞非自律的な遺伝子の発現抑制を介した組織の分化に寄与する42,43).また,ウイルスに感染した植物において,小分子RNAは全身抵抗性を確立する際のシグナル因子として維管束を介して長距離輸送される44).細胞のあいだを移行する小分子RNAは転写制御および転写後制御において機能しうることが示されており,siRNAについては,RNA依存的DNAメチル化経路により細胞非自律的にGFP遺伝子の転写抑制をひき起こしうる45).
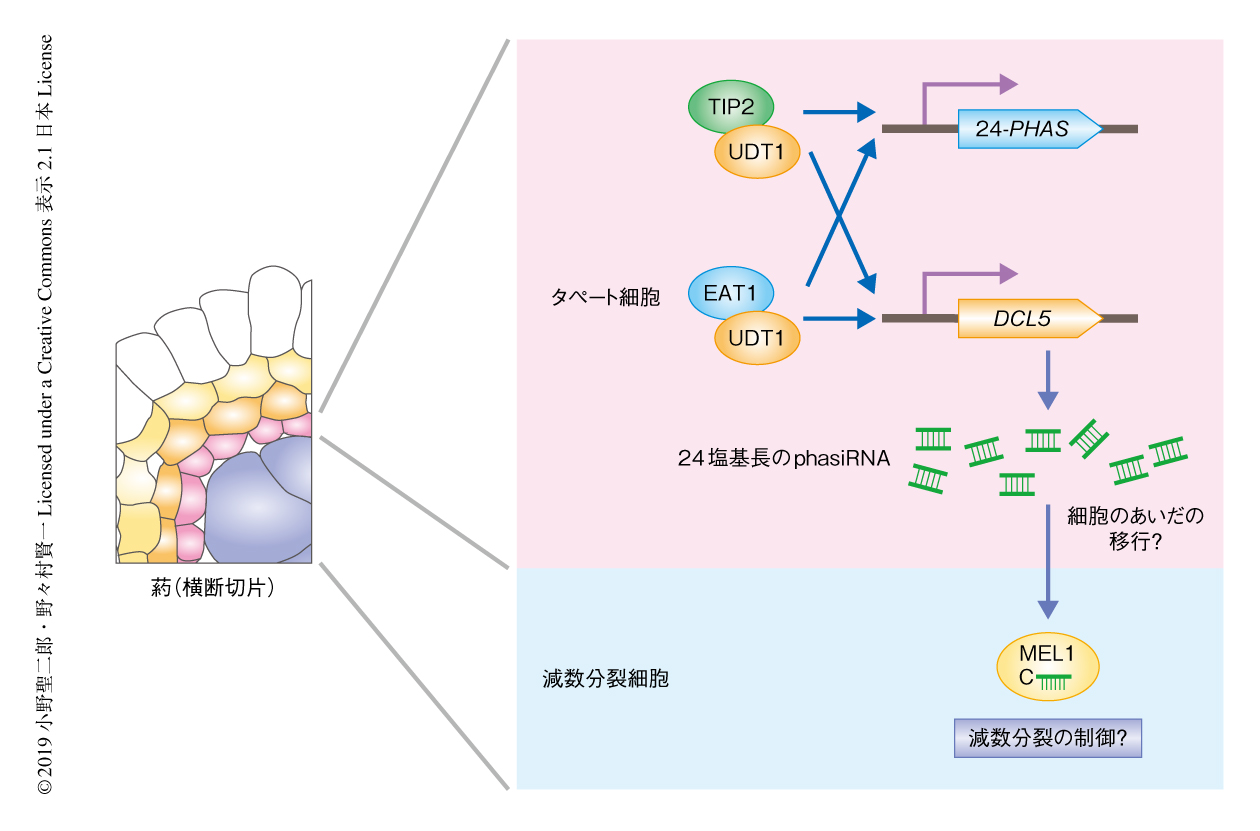
近年,phasiRNAも植物の生殖器官における細胞間シグナル因子として機能する可能性が浮上している.トウモロコシの葯においては,変異体の解析およびphasiRNAの産生経路においてはたらくDCLの遺伝子発現の組織特異性から,21塩基長および24塩基長のphasiRNAはそれぞれ表皮およびタペート層に依存的に産生される可能性が示唆されたが,それにくわえ,前駆体となるPHAS RNAやDCLがほとんど発現しないはずの花粉母細胞において成熟phasiRNAの高い発現が検出されている23).また,機械的に抽出した花粉母細胞を用いたトランスクリプトーム解析においても大量のphasiRNAが検出された33).筆者らの研究グループは,イネにおいてタペート層の分化に重要な役割をはたす転写因子TIP2およびEAT1が,減数分裂期において特異的に発現する24-PHAS RNAの転写を直接的に活性化することを見い出した18).TIP2およびEAT1は,phasiRNAの2次切断にはたらくDCL5をコードする遺伝子の転写も活性化すること,また,TIP2およびEAT1ともおもにタペート細胞の核に特異的に局在することから,24塩基長のphasiRNAの産生は葯のタペート細胞において完結すると推察された(図5).一方,イネにおいて花粉母細胞のみに存在するOsMEL1と結合する小分子RNAのなかに,タペート細胞において産生されるはずの24塩基長のphasiRNAが検出された18).トウモロコシおよびイネにおける結果からは,24塩基長のphasiRNAのタペート細胞から花粉母細胞への細胞のあいだの移行の可能性が支持された.
減数分裂のまえの花粉母細胞においては,細胞質に局在するOsMEL1は24塩基長のphasiRNAの発現が開始する減数分裂への移行期を境に核にも局在すること17,18),また,ヒストンH3のLys9の広範囲なジメチル化を介した減数分裂染色体の凝縮を促進することからも46),OsMEL1が核において機能する可能性が示唆される.AtAGO4は24塩基長のsiRNAと結合すると細胞質から核へと局在を変化させること47),mel1変異体においては染色体の凝縮の異常により減数分裂が早期に停止すること8),eat1変異体においては染色体の部分的な凝縮の異常をともなう減数分裂の進行の遅延が観察されること18),などを考えあわせると,OsMEL1および24塩基長のphasiRNAが減数分裂染色体のRNA依存的DNAメチル化に機能する可能性が暗示される.興味深い仮説だが,花粉母細胞それ自体がphasiRNAを産生し自律的に減数分裂を制御する可能性はいずれの結果からも完全には否定されず,24塩基長のphasiRNAが減数分裂の進行に寄与することを示す直接的な結果も得られていないため,今後,さらなる検証が必要である.
phasiRNAに関する情報は年々増えつづけているが,それらがどのように進化したのかについてはいまだに謎である.21塩基長のphasiRNAの産生に重要なDCL4は,植物において広く保存されている48).裸子植物のオウシュウトウヒ(Picea abies)においては,病害抵抗性にかかわるNB-LRRファミリーなどの遺伝子領域および非コード領域に由来する2種類の21塩基長のphasiRNAが混在し,後者はとくに生殖器官において高発現する49).一方,マメ科やナス科など真正双子葉植物の21塩基長のphasiRNAは多くがNB-LRR遺伝子領域に由来する28,50,51).これらを総合すると,裸子植物あるいは原始的な双子葉類以前は2種類の21塩基長のphasiRNAをあわせもつが,真正双子葉類および単子葉類はその一方のみを受け継ぎ,単子葉類が受け継いだ非コード領域に由来するphasiRNAが生殖細胞に特異的に発現するようになった可能性が示唆される49).しかし,2種類のphasiRNAがそれぞれ独立に進化したのか,あるいは,一方が他方から派生したのかははっきりしない.
一方,24塩基長のphasiRNAは,生殖以外の発生の過程においては一部の双子葉植物においても報告があるが52,53),miR2275およびDCL5ともに単子葉植物以外では見い出されないことから,24塩基長の生殖phasiRNAは単子葉植物の分岐ののちに出現した可能性が高い23).
多くの動物においてpiRNAの産生経路は,1次piRNA産生経路と,ピンポンサイクルとよばれる2次piRNA増幅経路とに区別される(図6).研究が進むショウジョウバエおよびマウスにおいて基本的な経路は共通しているが,以下では,マウスにおけるpiRNAの産生経路と植物における生殖phasiRNAの産生経路を比較する.
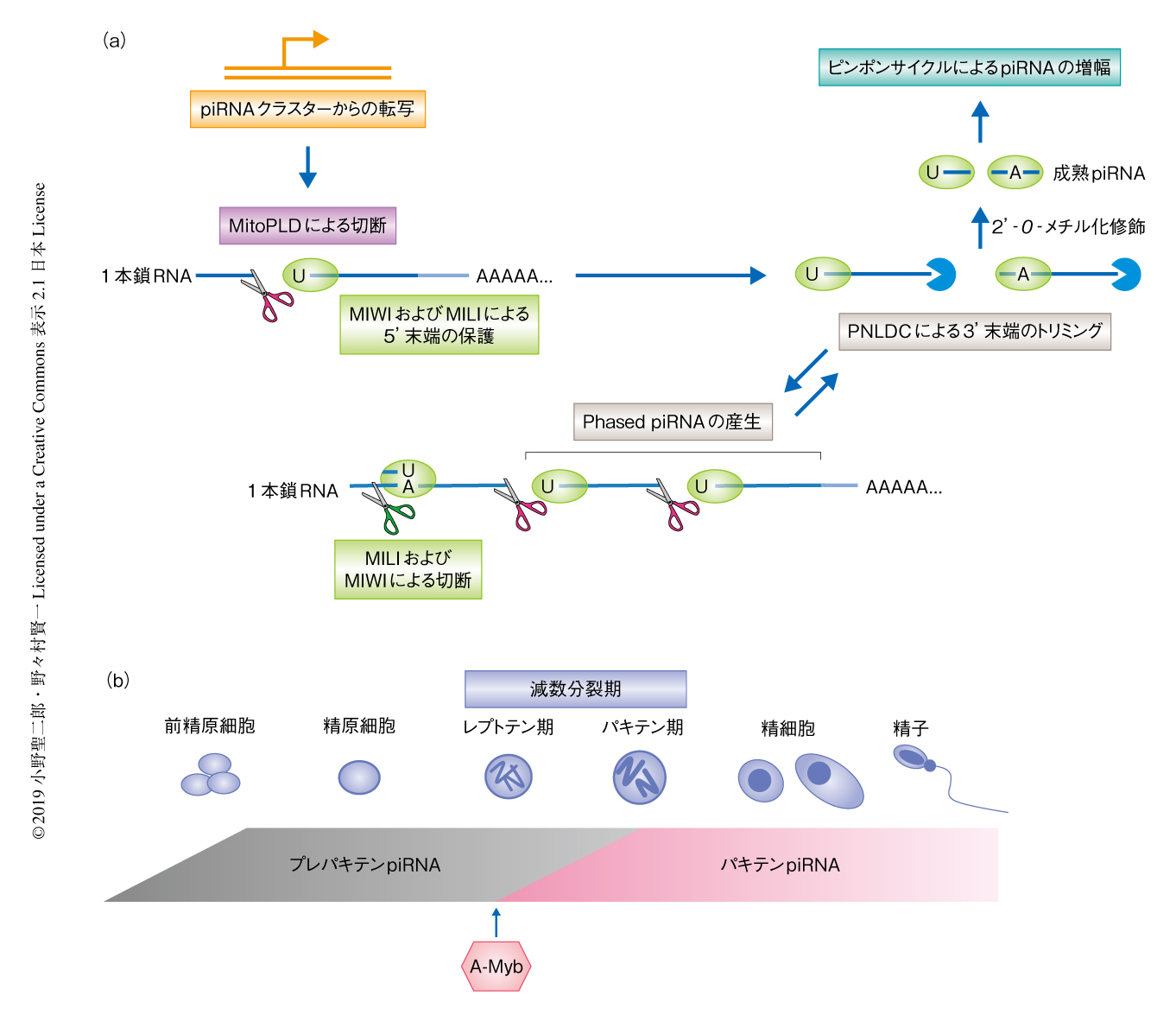
動物のpiRNAの産生経路と比較した際の,植物における生殖phasiRNAの産生経路の根本的な違いは,前駆体となるlncRNAを2本鎖化するRNA依存的RNAポリメラーゼの存在,および,DCLによる2本鎖RNAの連続的かつ等間隔な切断,の2点に集約される.マウスにおける1次piRNA産生経路においては,RNAポリメラーゼIIにより転写された1本鎖のpiRNA前駆体が,エンドヌクレアーゼ活性をもつMitoPLDによりUの直前で切断される(図6).そののち,5’末端がUのRNAに嗜好性をもつMILIあるいはMIWI(生殖細胞において機能するPIWIファミリーのArgonaute)が切断されたRNAの5’末端側を保護し,3’-5’エキソヌクレアーゼであるPNLDC1による3’末端のトリミングをへて,1次piRNAが成熟する54,55).MitoPLDによる切断部位の下流においても同様のことがくり返されるため,ひとつの前駆体RNAから複数のpiRNAが産生される56,57)(phased piRNA経路).ここでは詳細は省くが,ピンポンサイクルはRNA依存的RNAポリメラーゼをもたない生物に特有のpiRNAの増幅システムである.近年,ピンポンサイクルにもphased piRNA経路が寄与する可能性が提唱されている58).
phased piRNA経路は,いっけん,植物における生殖phasiRNAの産生経路と似ている.しかし,piRNAの産生経路においてはArgonauteとエンドヌクレアーゼとが協調して1本鎖RNAを切断するため,piRNAの5’末端はPIWIファミリーのArgonauteの嗜好性を反映する(図6).一方,植物の生殖phasiRNAの産生経路においては,DCLが単独で2本鎖RNAを切断するため,産生されたphasiRNAに5’末端の塩基のかたよりはない.phasiRNA経路においてもArgonauteがmiRNAとともに1次切断を起こすが,あくまでDCLによる切断の起点を決めるだけである.
生物学的な意味ははっきりしないが,phasiRNAの5’末端の配列にかたよりがないという性質は,生殖器官において発現するすべてのタイプのArgonauteがphasiRNAを利用することが可能であることを意味する.植物は季節や気候の変動に応じて地上部のメリステムの任意の細胞から後生的に生殖器官を分化する.植物細胞の高い全能性とも関係する,いわば“いつでもどこでも”生殖器官を分化しうる性質は,胞子体という動物にはない生殖様式がなせる技といえる.生殖成長への相転換をきっかけとして発現を開始する生殖phasiRNAは,生殖細胞において特異的に発現するArgonauteだけでなく,栄養成長から生殖成長期にかけて発現するArgonauteにも取り込まれ,それらのArgonauteの標的となるRNAを一斉に変更し,いつでも細胞運命を体細胞分裂から減数分裂へとふりむけられるような体制をとっているのかもしれない.
マウスの精巣における生殖piRNAのうち,パキテンpiRNAとよばれる減数分裂細胞に豊富なpiRNAは,MYBファミリーの転写因子A-Mybが,piRNAの前駆体RNAやpiRNAの産生経路に必要なTdrd6遺伝子やMiwi遺伝子などの転写を協調的に活性化することにより,はじめて機能しうることが知られる59).この図式は,さきに述べた,イネにおけるbHLH型の転写因子EAT1による24塩基長のphasiRNAの産生経路の制御18) を連想させる.パキテンpiRNAは,植物の生殖phasiRNAと同様,相補的な配列をもつ標的となるRNAは未解明だが,最近の研究から,パキテンpiRNAが精子の形成の過程において父方に由来する内在性のmRNAを分解する可能性なども示唆されている60).
マウスにおいては,ミトコンドリアの間隙に存在するpiPボディや核膜の近傍のchromatoidボディなどの細胞質RNA顆粒がphased piRNAの産生の場として知られる61).ショウジョウバエにおいてもヌアージュとよばれる類似の細胞質RNA顆粒の重要性が報告されるなど62),動物においてはpiRNAの産生に必須の生殖細胞における細胞質顆粒が保存されている.一方,植物においては明瞭な生殖細胞における細胞質顆粒の報告はない.シロイヌナズナにおいてはp4-siRNAのプロセシングの中心が核のCajalボディであること63),生殖器官以外で発現するphasiRNAの産生の初期の段階が小胞膜の膜結合ポリソームに依存すること64),などが知られるが,いずれも生殖細胞に特化した機構ではない.植物に明瞭な生殖細胞における細胞質顆粒がいまだにみつかっていないことは,その特徴的な生殖様式と考えあわせて興味深い.
減数分裂組換えは,相同染色体を鋳型とした相同組換えによる2本鎖DNA切断修復により起こり,その一部は相同染色体の部分的な交換をともなう乗換えとなる.その原理をたくみに応用したのが交雑育種法である.最近の研究から,植物においても減数分裂における2本鎖DNA切断および乗換えの分布は染色体において一様ではなく,DNAメチル化やヒストン修飾などクロマチンの修飾や構造の違い,すなわち,エピジェネティックランドスケープが大きく影響することが明らかにされてきた65,66).クロマチンリモデリングタンパク質などにくわえ,このレビューにおいて紹介したphasiRNAなど生殖器官において高発現する小分子RNAは,転写抑制を介して減数分裂染色体の条件的ヘテロクロマチン化などエピジェネティックランドスケープの形成に一役かっている可能性がきわめて高い.phasiRNAおよびArgonauteの減数分裂における作用機序そのほかの知見が,減数分裂において組換えの位置や頻度の分子遺伝学的な操作による作物の育種に応用される日は,すぐそこまできているのかもしれない.
略歴:2015年 総合研究大学院大学生命科学研究科博士課程 修了,同年より国立遺伝学研究所 特任研究員.
研究テーマ:植物,とくに作物における減数分裂の制御機構およびその育種への応用.
野々村 賢一(Ken-Ichi Nonomura)
国立遺伝学研究所 准教授.
研究室URL:http://nonomuralab-nig.sakura.ne.jp/top_j.html
© 2019 小野聖二郎・野々村賢一 Licensed under CC 表示 2.1 日本
(国立遺伝学研究所 植物細胞遺伝研究室)
email:野々村賢一
領域融合レビュー, 8, e002 (2019) DOI: 10.7875/leading.author.8.e002
Seijiro Ono & Ken-Ichi Nonomura: Biogenesis and function of plant reproductive phasiRNAs abundantly expressed in meiotic stages.
要 約
減数分裂は,両親に由来する染色体から新たな遺伝子の組合せを創出し,染色体の数や構造を監視する役割を担う,遺伝や育種の根幹をなす生命現象である.これまでに,不対合領域に分布する遺伝子の発現抑制や,piRNAによる生殖細胞系列におけるトランスポゾンの抑制など,生殖細胞の発生や減数分裂の進行と小分子RNAに依存的なエピジェネティックな機構との関連性が多くのモデル生物において報告されている.植物にpiRNAは保存されていないが,近年,単子葉植物の生殖器官において高発現する21塩基長および24塩基長のsiRNAとしてphasiRNAが発見され,生殖細胞の初期の発生を制御する可能性のある小分子RNAとして注目されている.phasiRNAの前駆体となるlncRNAは,植物のゲノムにおいて数百か所から数千か所の遺伝子領域あるいは遺伝子間領域に分布する.前駆体RNAがRNAポリメラーゼIIにより転写されたのち,22塩基長のmiRNAとArgonauteとの複合体による1次切断,RNA依存的RNAポリメラーゼによる2本鎖化,リボヌクレアーゼIII様酵素であるDCLによるmiRNAによる切断部位を起点とした21塩基長あるいは24塩基長の間隔での切断をへて,成熟phasiRNAが産生される.このレビューにおいては,おもに減数分裂期に高発現するphasiRNAについて,産生経路や発現の時空間的な制御,相互作用するArgonauteの種類および機能などを解説し,動物におけるpiRNAの産生経路との比較をつうじ,植物に特徴的な生殖システムをあぶりだすことを試みる.
はじめに
減数分裂は,有性生殖を行うすべての真核生物に保存される,生殖の過程における中心的なイベントのひとつである.減数分裂における組換えは両親に由来する染色体から新たな遺伝子の組合せを創出し次世代へと伝達する.さらに,減数分裂は相同染色体の対合をつうじ染色体の数および構造の監視も担う.たとえば,ヒョウとライオンの1代雑種であるレオポンに生殖能力がないのは,両親の染色体の数および構造の違いによる減数分裂における染色体の対合の異常が一因とされる.このように,減数分裂は種内の遺伝的な多様化に機能するとともに,染色体の数および構造の認識をつうじ種間の遺伝子の交換を制限し種の維持に貢献する.
このレビューにおけるおもなトピックである小分子RNAと減数分裂との関連が知られた最初のきっかけは,アカパンカビの不対合領域における遺伝子の発現抑制の研究であり1,2),マウスにおいても類似の現象が報告された3,4).植物においては,最近,生殖の過程において機能する小分子RNAに関する報告があいついだ.とくに,花粉において先駆的な研究が多いが,それについてはほかのすぐれたレビューを参照いただき5),このレビューにおいては,減数分裂期に高発現する小分子RNAのひとつphasiRNAの産生経路について,植物に特徴的な生殖システムの説明をおりまじえて解説し,動物のpiRNAとの比較から,いまだに多くの謎を残す植物のphasiRNAの役割について考察する.
1.減数分裂にみる植物と動物の生殖様式の違い
“世代”という言葉の生殖生物学における真の意味をご存知だろうか? 生殖生物学においては,親子世代といった一般的な用法とは異なり,同一の生活環に出現する異なる生殖様式のそれぞれをさす6,7).ヒトも含め多くの動物においては,減数分裂により生じた細胞はそのまま配偶子へと成熟するため,動物の体は配偶子を生み出す配偶体と定義され,生活環に現われる生殖世代は配偶体(有性生殖)世代のみである.一方,陸上植物の減数分裂により生じた細胞(胞子)は,体細胞分裂をへて多細胞の配偶体(被子植物においては,花粉および胚のう)となり,その内部に配偶子を形成する(図1).配偶体は,動物および植物においてともに有性生殖世代に分類される.一方,われわれが通常目にする被子植物の体は,配偶体(胞子に由来)を無性的につくりだす体,すなわち,胞子体として無性生殖世代に定義される.このように,陸上植物は無性と有性の2種類の生殖世代が同一の生活環に交互に現われる世代交代をするという特徴をもつ.つまり,生殖様式の観点から,動物の生殖細胞の発生と比較される植物の発生の過程は,減数分裂ののちから受精にかけての配偶体の形成の過程であり,陸上植物にとり,減数分裂は無性生殖世代の最後のイベントである.
このレビューにおける焦点である生殖に特異的な小分子RNAとして,動物においてはpiRNA(PIWI-interacting RNA)が同定され,さかんに研究されている.piRNAは,生殖細胞系列においてトランスポゾンを不活性化し完全なゲノムを次世代に伝達することが一義的な役割と考えられている.このようなシステムはすべての生物に不可欠なはずだが,植物はpiRNAの産生経路に類似した小分子RNAの産生経路,および,piRNAと結合するPIWIファミリーに属するArgonaute(AGO)を保存していない8-10).この事実は,植物に特有の生殖世代を反映しているのかもしれない.その一方で,植物において減数分裂細胞およびその原基細胞には,減数分裂あるいはそののちの有性生殖(配偶体の形成)の運命を左右する遺伝子,および,小分子RNAを含む非コードRNAが多く発現しており11-13),それらがpiRNAに相当する機能を担う可能性は十分に考えられる.
2.植物に特徴的なsiRNAであるphasiRNA
小分子RNAは20~30塩基長のRNAであり,ガイドRNAとしてArgonauteに取り込まれてRISC(RNA-induced silencing complex)とよばれる複合体を形成し,配列に依存的に転写後あるいは転写のレベルにおいて遺伝子の発現抑制に機能する.転写後抑制においては,RISCが標的となるmRNAと結合して切断や翻訳の抑制を行う.転写抑制は,RISCが標的となるクロマチン領域にRNAあるいはDNAを介して結合し,その領域のクロマチンに修飾をひき起こす機構である.植物における代表的な転写抑制としてRNA依存的DNAメチル化が知られている14).一般に,転写後抑制は細胞質に局在するArgonauteが,転写抑制は核に局在するArgonauteが,それぞれ実行する15).動物の小分子RNAは,miRNA(microRNA),siRNA(small interfering RNA),そして,生殖器官において機能するpiRNAの3つに大別される.とくに,piRNAを含む小分子RNAの産生については,泉 奈津子・泊 幸秀, 領域融合レビュー, 7, e003 (2018) を参照されたい.
植物はpiRNAをもたない一方で,phasiRNA(phased secondary siRNA)とよばれる独自のsiRNAを発達させた.phasiRNAは,遺伝子領域あるいは遺伝子間領域からRNAポリメラーゼIIにより転写されるlncRNA(long non-coding RNA)を前駆体とする10)(図2a).この前駆体RNAをPHAS RNA,由来するゲノム領域をPHAS座とよぶ.phasiRNAの産生は,PHAS RNAの5’末端あるいは両方の末端に保存される標的配列を,22塩基長のmiRNAとAGO1あるいはAGO7との複合体が切断する1次切断にはじまる.切断された1本鎖のPHAS RNAは,RNA依存的RNAポリメラーゼであるRDR6により2本鎖化される.そののち,miRNAの切断部位を起点として,リボヌクレアーゼIII様の酵素であるDCL4あるいはDCL5が,2本鎖のPHAS RNAを21塩基長(DCL4)あるいは24塩基長(DCL5)の等間隔で2次切断することにより,phasiRNAが産生される.
phasiRNAは,PHAS座のセンス鎖およびアンチセンス鎖の両方に,21塩基長あるいは24塩基長の位相をもってマップされている.よくタンデムリピートと誤解されるが,同一のPHAS座においてもphasiRNAのあいだに配列の保存性はなく,それぞれがユニークな配列としてマップされる場合が多い.24塩基長のphasiRNAの前駆体をコードする24-PHAS座はイネのゲノムに100座以上,21塩基長のphasiRNAの前駆体をコードする21-PHAS座にいたっては1000座以上が同定されている16-18)(図2b).同様のパターンを示すものにtasiRNA(trans-acting siRNA)がある.tasiRNAは植物のjuvenile-adult相転換を含む発生の過程において重要な役割をはたすsiRNAとして,以前に報告されていた19).その産生経路はphasiRNAと類似するが,tasiRNAの産生経路においては前駆体となるlncRNAが約10種類と少なく,特定の遺伝子のmRNAにトランスに作用する20).phasiRNAはシスおよびトランスの両方で作用するsiRNAを含む用語であることから,tasiRNAをphasiRNAの一種として定義することが提案されている10).
3.植物の生殖器官において豊富に発現するphasiRNA
phasiRNAは花序組織に豊富に存在するsiRNAとして,イネにおいて最初に報告された16).そののち,花序のうちとくに雄しべ(葯)において豊富に発現すること,1次切断がAGO1および22塩基長のmiRNA(miR2118あるいはmiR2275),2次切断がRDR6およびDCL4あるいはDCL5に依存することが報告され21,22),phasiRNAとして定義された.なお,生殖器官以外にも発現するphasiRNAの報告があるが,とくに単子葉植物では生殖器官において高い発現を示す場合が多いため,このレビューでは,とくに断らないかぎり生殖phasiRNA(reproductive phasiRNA)をphasiRNAとよぶ.
トウモロコシおよびイネにおいては,葯の発達段階にそったphasiRNAの時空間的な発現パターンが明らかにされている23,24).21塩基長のphasiRNAは減数分裂のまえから減数分裂期にかけて葯に豊富に発現する一方,24塩基長のphasiRNAは減数分裂期においてピンポイントに発現する(図3).イネの減数分裂期の葯において検出される小分子RNAのうち,トランスポゾンをふくむくり返し配列に由来するsiRNAが全体の約75%をしめるが,それにつづいて21塩基長のphasiRNAおよび24塩基長のphasiRNAがそれぞれ15%および6%存在する18).
葉などの表皮に発現するHD-ZIP IVファミリーの転写因子であるOCL4の機能欠損変異体の葯において,21塩基長のphasiRNAの発現量が劇的に減少したことから,21塩基長のphasiRNAおよびその前駆体は葯の表皮にて発現する可能性が示された23).24塩基長のphasiRNAは,葯のうち雄性の減数分裂細胞をとりかこむタペート層とよばれる体細胞組織において特異的に発現することが示された18,23)(図3).タペート細胞は減数分裂ののちに形成される胞子に花粉殻の材料や栄養を供給するが,減数分裂期における機能は不明であった.のちに述べるように,最近,筆者らの研究グループにより,タペート層において特異的に発現し機能するbHLH型の転写因子が,減数分裂に特異的な24塩基長のphasiRNAの産生を活性化することが明らかにされた.
生殖phasiRNAは,イネやトウモロコシにくわえ,近年では,カモジグサ25,26),ユリ,アスパラガス27) などの単子葉植物においてあいついで報告されている.一方,マメ科など双子葉植物においては,栄養成長期に特定の遺伝子領域からphasiRNAが産生されるが,生殖器官における豊富な発現は報告されておらず,また,代表的なモデル植物であるシロイヌナズナにおいてはtasiRNA以外に明瞭なphasiRNAは検出されないため28),生殖phasiRNAの研究はおもに単子葉植物において進んでいる.
4.phasiRNAの分子機能
生殖phasiRNAの分子機能についてはいまだによくわかっていない.phasiRNAの配列の種類が膨大であることが標的となるRNAの予測がむずかしい一因である.イネのゲノムに21-PHAS座は1000以上も存在し16,17),そこからは合計で2万種類以上のphasiRNAの産生が見込まれる.24-PHAS座は100座あまりだが18),それでも見込まれる24塩基長のphasiRNAの種類は膨大である.標的となるRNAのゲノムワイドな予測29) や,機械学習による予測30) も試みられているが,phasiRNAの配列からは標的となるRNAの明瞭な同定にはいたっていない.また,ほかのsiRNAと比較して,phasiRNAはユニークな遺伝子間領域に由来する傾向が示された18,23,30).phasiRNAのこのような性質は,RNAポリメラーゼIVに依存的なsiRNA や31),epigenetically activated siRNA(easiRNA)など32),特定の遺伝子あるいはトランスポゾンを標的とする植物のほかのsiRNAの性質とは一線を画す.
これらの知見を総合すると,phasiRNAは核において自らの由来する領域,すなわち,PHAS座の転写抑制に作用する可能性がきわめて高い.事実,トウモロコシの21-PHAS座および24-PHAS座の周辺は,葯および雄性減数分裂細胞(花粉母細胞)におけるDNAのメチル化,とくにCHHメチル化(H:A,T,G)が成葉などの栄養成長器官より高い傾向が示されている33).植物のsiRNAは,RNA依存的DNAメチル化に代表される転写抑制に機能することが知られており14),phasiRNAも生殖の過程において同様の経路に組み込まれている可能性は十分に考えられる(図4).
phasiRNAの“secondary”とは,miRNAによる1次切断をへてDCLによる2次切断により産生されることにちなむ.しかし,イネやトウモロコシの21-PHAS座のなかには,miR2118による1次切断部位からはじまる位相におさまるphasiRNAのほか,その位相から9塩基ずれた位相をもつsiRNAが多く見い出される34).これらは,phasiRNAによる21-PHAS RNAの3次切断が起こり,そこからさらにDCL4による連続切断がはじまると考えるとうまく説明される.すなわち,phasiRNAは転写制御への関与の可能性にくわえ,PHAS RNAを標的とした転写後抑制にかかわり,それ自体の増幅サイクルを促進する可能性が提唱されている(図4).
5.減数分裂期において機能する植物のArgonaute
Argonauteは,シロイヌナズナにおいて10種類,イネにおいて19種類,トウモロコシにおいて17種類が保存され,それらはAGO1/5/10ファミリー,AGO2/3/7ファミリー,AGO4/6/9ファミリーに分類される35).生殖の過程における関与の知られるArgonauteはAGO4/6/9ファミリーおよびAGO1/5/10ファミリーに多く含まれるが(phasiRNAの産生において1次切断を実行するArgonauteは除く),それらはいずれも動物のPIWIファミリーには属さない.
シロイヌナズナのAGO9(AtAGO9)は,おもにトランスポゾンに由来する24塩基長のsiRNAと結合する36).AtAGO9は雌性減数分裂細胞の核および雌性減数分裂細胞をつつむ珠心表皮の細胞質に局在し36,37),隣接する体細胞が雌性減数分裂細胞になることを抑制する36).トウモロコシのAGO104(ZmAGO104)はAtAGO9のホモログであり,変異体の葯においては減数分裂の停止や減数分裂染色体の脱凝縮などの異常が,また,雌しべの側では減数分裂の異常にともなう非還元性の配偶子の形成が観察される38).シロイヌナズナのAGO4(AtAGO4)は生活環をつうじて恒常的に発現するが,変異体においては花粉母細胞における減数分裂染色体の脱凝縮,分配の遅滞,架橋などの異常が起こる39).AtAGO4,AtAGO9,ZmAGO104はいずれもAGO4/6/9ファミリーに属し,とくにAtAGO4はRNA依存的DNAメチル化経路において中心的な役割を担うことから,減数分裂とRNA依存的DNAメチル化経路との密接な関連性が示唆される.
イネMEL1(OsMEL1)は,雌雄の胞子原基細胞と胞子形成細胞(いずれも,減数分裂細胞に分化する細胞)および減数分裂細胞において特異的に発現するArgonauteであり,その変異体においては減数分裂染色体の凝縮が早期に停止して完全不稔になる8).OsMEL1のオーソログであるシロイヌナズナのAGO5(AtAGO5)については,花粉の精細胞および珠心表皮における発現が報告されている40,41).しかし,AtAGO5のドミナントネガティブ変異体は減数分裂の直後において胞子の発生が異常となるが,機能欠損型変異体は減数分裂において顕著な表現型を示さない41).AGO1/5/10ファミリーに属するイネの5つのArgonauteのうち,OsMEL1は葯および雌しべ,OsAGO12は葯に特異的に,OsAGO14は雌しべに特異的に発現するなど9),AGO1/5/10ファミリーは生殖に特異的に機能するArgonauteとして機能が分化した可能性が高い.
現時点では,生殖phasiRNAとの結合が明確に示されたArgonauteはイネにおけるOsMEL1のみである.OsMEL1は5’末端がCであるsiRNAと結合する傾向を示す17).減数分裂よりまえから減数分裂にかけて,おもに21塩基長のphasiRNAと結合し,その割合は結合するすべての小分子RNAの約60%におよぶ17,18).OsMEL1は減数分裂期に24塩基長のphasiRNAとも結合するが,結合するすべての小分子RNAの1%未満である18).この結果から,OsMEL1は5’末端がCである21塩基長のphasiRNAに高い嗜好性をもつ可能性が示唆される.しかし,これは葯において発現するsiRNAの割合を反映した結果かもしれず,事実,OsMEL1と結合する小分子RNAにはトランスポゾンをふくむくり返し配列に由来するsiRNAも20%程度含まれており17,18),phasiRNA以外のsiRNAとOsMEL1との結合の重要性も否定できない.
6.phasiRNAが細胞間シグナル因子として機能する可能性
根の放射状パターンや葉の表裏パターンの形成において,小分子RNAは細胞のあいだを移動し,細胞非自律的な遺伝子の発現抑制を介した組織の分化に寄与する42,43).また,ウイルスに感染した植物において,小分子RNAは全身抵抗性を確立する際のシグナル因子として維管束を介して長距離輸送される44).細胞のあいだを移行する小分子RNAは転写制御および転写後制御において機能しうることが示されており,siRNAについては,RNA依存的DNAメチル化経路により細胞非自律的にGFP遺伝子の転写抑制をひき起こしうる45).
近年,phasiRNAも植物の生殖器官における細胞間シグナル因子として機能する可能性が浮上している.トウモロコシの葯においては,変異体の解析およびphasiRNAの産生経路においてはたらくDCLの遺伝子発現の組織特異性から,21塩基長および24塩基長のphasiRNAはそれぞれ表皮およびタペート層に依存的に産生される可能性が示唆されたが,それにくわえ,前駆体となるPHAS RNAやDCLがほとんど発現しないはずの花粉母細胞において成熟phasiRNAの高い発現が検出されている23).また,機械的に抽出した花粉母細胞を用いたトランスクリプトーム解析においても大量のphasiRNAが検出された33).筆者らの研究グループは,イネにおいてタペート層の分化に重要な役割をはたす転写因子TIP2およびEAT1が,減数分裂期において特異的に発現する24-PHAS RNAの転写を直接的に活性化することを見い出した18).TIP2およびEAT1は,phasiRNAの2次切断にはたらくDCL5をコードする遺伝子の転写も活性化すること,また,TIP2およびEAT1ともおもにタペート細胞の核に特異的に局在することから,24塩基長のphasiRNAの産生は葯のタペート細胞において完結すると推察された(図5).一方,イネにおいて花粉母細胞のみに存在するOsMEL1と結合する小分子RNAのなかに,タペート細胞において産生されるはずの24塩基長のphasiRNAが検出された18).トウモロコシおよびイネにおける結果からは,24塩基長のphasiRNAのタペート細胞から花粉母細胞への細胞のあいだの移行の可能性が支持された.
減数分裂のまえの花粉母細胞においては,細胞質に局在するOsMEL1は24塩基長のphasiRNAの発現が開始する減数分裂への移行期を境に核にも局在すること17,18),また,ヒストンH3のLys9の広範囲なジメチル化を介した減数分裂染色体の凝縮を促進することからも46),OsMEL1が核において機能する可能性が示唆される.AtAGO4は24塩基長のsiRNAと結合すると細胞質から核へと局在を変化させること47),mel1変異体においては染色体の凝縮の異常により減数分裂が早期に停止すること8),eat1変異体においては染色体の部分的な凝縮の異常をともなう減数分裂の進行の遅延が観察されること18),などを考えあわせると,OsMEL1および24塩基長のphasiRNAが減数分裂染色体のRNA依存的DNAメチル化に機能する可能性が暗示される.興味深い仮説だが,花粉母細胞それ自体がphasiRNAを産生し自律的に減数分裂を制御する可能性はいずれの結果からも完全には否定されず,24塩基長のphasiRNAが減数分裂の進行に寄与することを示す直接的な結果も得られていないため,今後,さらなる検証が必要である.
7.phasiRNAの進化
phasiRNAに関する情報は年々増えつづけているが,それらがどのように進化したのかについてはいまだに謎である.21塩基長のphasiRNAの産生に重要なDCL4は,植物において広く保存されている48).裸子植物のオウシュウトウヒ(Picea abies)においては,病害抵抗性にかかわるNB-LRRファミリーなどの遺伝子領域および非コード領域に由来する2種類の21塩基長のphasiRNAが混在し,後者はとくに生殖器官において高発現する49).一方,マメ科やナス科など真正双子葉植物の21塩基長のphasiRNAは多くがNB-LRR遺伝子領域に由来する28,50,51).これらを総合すると,裸子植物あるいは原始的な双子葉類以前は2種類の21塩基長のphasiRNAをあわせもつが,真正双子葉類および単子葉類はその一方のみを受け継ぎ,単子葉類が受け継いだ非コード領域に由来するphasiRNAが生殖細胞に特異的に発現するようになった可能性が示唆される49).しかし,2種類のphasiRNAがそれぞれ独立に進化したのか,あるいは,一方が他方から派生したのかははっきりしない.
一方,24塩基長のphasiRNAは,生殖以外の発生の過程においては一部の双子葉植物においても報告があるが52,53),miR2275およびDCL5ともに単子葉植物以外では見い出されないことから,24塩基長の生殖phasiRNAは単子葉植物の分岐ののちに出現した可能性が高い23).
8.piRNAとphasiRNAの比較
多くの動物においてpiRNAの産生経路は,1次piRNA産生経路と,ピンポンサイクルとよばれる2次piRNA増幅経路とに区別される(図6).研究が進むショウジョウバエおよびマウスにおいて基本的な経路は共通しているが,以下では,マウスにおけるpiRNAの産生経路と植物における生殖phasiRNAの産生経路を比較する.
動物のpiRNAの産生経路と比較した際の,植物における生殖phasiRNAの産生経路の根本的な違いは,前駆体となるlncRNAを2本鎖化するRNA依存的RNAポリメラーゼの存在,および,DCLによる2本鎖RNAの連続的かつ等間隔な切断,の2点に集約される.マウスにおける1次piRNA産生経路においては,RNAポリメラーゼIIにより転写された1本鎖のpiRNA前駆体が,エンドヌクレアーゼ活性をもつMitoPLDによりUの直前で切断される(図6).そののち,5’末端がUのRNAに嗜好性をもつMILIあるいはMIWI(生殖細胞において機能するPIWIファミリーのArgonaute)が切断されたRNAの5’末端側を保護し,3’-5’エキソヌクレアーゼであるPNLDC1による3’末端のトリミングをへて,1次piRNAが成熟する54,55).MitoPLDによる切断部位の下流においても同様のことがくり返されるため,ひとつの前駆体RNAから複数のpiRNAが産生される56,57)(phased piRNA経路).ここでは詳細は省くが,ピンポンサイクルはRNA依存的RNAポリメラーゼをもたない生物に特有のpiRNAの増幅システムである.近年,ピンポンサイクルにもphased piRNA経路が寄与する可能性が提唱されている58).
phased piRNA経路は,いっけん,植物における生殖phasiRNAの産生経路と似ている.しかし,piRNAの産生経路においてはArgonauteとエンドヌクレアーゼとが協調して1本鎖RNAを切断するため,piRNAの5’末端はPIWIファミリーのArgonauteの嗜好性を反映する(図6).一方,植物の生殖phasiRNAの産生経路においては,DCLが単独で2本鎖RNAを切断するため,産生されたphasiRNAに5’末端の塩基のかたよりはない.phasiRNA経路においてもArgonauteがmiRNAとともに1次切断を起こすが,あくまでDCLによる切断の起点を決めるだけである.
生物学的な意味ははっきりしないが,phasiRNAの5’末端の配列にかたよりがないという性質は,生殖器官において発現するすべてのタイプのArgonauteがphasiRNAを利用することが可能であることを意味する.植物は季節や気候の変動に応じて地上部のメリステムの任意の細胞から後生的に生殖器官を分化する.植物細胞の高い全能性とも関係する,いわば“いつでもどこでも”生殖器官を分化しうる性質は,胞子体という動物にはない生殖様式がなせる技といえる.生殖成長への相転換をきっかけとして発現を開始する生殖phasiRNAは,生殖細胞において特異的に発現するArgonauteだけでなく,栄養成長から生殖成長期にかけて発現するArgonauteにも取り込まれ,それらのArgonauteの標的となるRNAを一斉に変更し,いつでも細胞運命を体細胞分裂から減数分裂へとふりむけられるような体制をとっているのかもしれない.
マウスの精巣における生殖piRNAのうち,パキテンpiRNAとよばれる減数分裂細胞に豊富なpiRNAは,MYBファミリーの転写因子A-Mybが,piRNAの前駆体RNAやpiRNAの産生経路に必要なTdrd6遺伝子やMiwi遺伝子などの転写を協調的に活性化することにより,はじめて機能しうることが知られる59).この図式は,さきに述べた,イネにおけるbHLH型の転写因子EAT1による24塩基長のphasiRNAの産生経路の制御18) を連想させる.パキテンpiRNAは,植物の生殖phasiRNAと同様,相補的な配列をもつ標的となるRNAは未解明だが,最近の研究から,パキテンpiRNAが精子の形成の過程において父方に由来する内在性のmRNAを分解する可能性なども示唆されている60).
マウスにおいては,ミトコンドリアの間隙に存在するpiPボディや核膜の近傍のchromatoidボディなどの細胞質RNA顆粒がphased piRNAの産生の場として知られる61).ショウジョウバエにおいてもヌアージュとよばれる類似の細胞質RNA顆粒の重要性が報告されるなど62),動物においてはpiRNAの産生に必須の生殖細胞における細胞質顆粒が保存されている.一方,植物においては明瞭な生殖細胞における細胞質顆粒の報告はない.シロイヌナズナにおいてはp4-siRNAのプロセシングの中心が核のCajalボディであること63),生殖器官以外で発現するphasiRNAの産生の初期の段階が小胞膜の膜結合ポリソームに依存すること64),などが知られるが,いずれも生殖細胞に特化した機構ではない.植物に明瞭な生殖細胞における細胞質顆粒がいまだにみつかっていないことは,その特徴的な生殖様式と考えあわせて興味深い.
おわりに
減数分裂組換えは,相同染色体を鋳型とした相同組換えによる2本鎖DNA切断修復により起こり,その一部は相同染色体の部分的な交換をともなう乗換えとなる.その原理をたくみに応用したのが交雑育種法である.最近の研究から,植物においても減数分裂における2本鎖DNA切断および乗換えの分布は染色体において一様ではなく,DNAメチル化やヒストン修飾などクロマチンの修飾や構造の違い,すなわち,エピジェネティックランドスケープが大きく影響することが明らかにされてきた65,66).クロマチンリモデリングタンパク質などにくわえ,このレビューにおいて紹介したphasiRNAなど生殖器官において高発現する小分子RNAは,転写抑制を介して減数分裂染色体の条件的ヘテロクロマチン化などエピジェネティックランドスケープの形成に一役かっている可能性がきわめて高い.phasiRNAおよびArgonauteの減数分裂における作用機序そのほかの知見が,減数分裂において組換えの位置や頻度の分子遺伝学的な操作による作物の育種に応用される日は,すぐそこまできているのかもしれない.
文 献
- Shiu, P. K., Raju, N. B., Zickler, D. et al.: Meiotic silencing by unpaired DNA. Cell, 107, 905-916 (2001)[PubMed]
- Hammond, T. M., Spollen, W. G., Decker, L. M. et al.: Identification of small RNAs associated with meiotic silencing by unpaired DNA. Genetics, 194, 279-284 (2013)[PubMed]
- Modzelewski, A. J., Holmes, R. J., Hilz, S. et al.: AGO4 regulates entry into meiosis and influences silencing of sex chromosomes in the male mouse germline. Dev. Cell, 23, 251-264 (2012)[PubMed]
- Turner, J. M.: Meiotic silencing in mammals. Annu. Rev. Genet., 49, 395-412 (2015)[PubMed]
- Martinez, G. & Kohler, C.: Role of small RNAs in epigenetic reprogramming during plant sexual reproduction. Curr. Opin. Plant Biol., 36, 22-28 (2017)[PubMed]
- 山岸 宏: 比較生殖学. 東海大学出版会 (1995)
- Horst, N. A. & Reski, R.: Alternation of generations: unravelling the underlying molecular mechanism of a 165-year-old botanical observation. Plant Biol., 18, 549-551 (2016)[PubMed]
- Nonomura, K. I., Morohoshi, A., Nakano, M. et al.: A germ cell specific gene of the ARGONAUTE family is essential for the progression of premeiotic mitosis and meiosis during sporogenesis in rice. Plant Cell, 19, 2583-2594 (2007)[PubMed]
- Kapoor, M., Arora, R., Lama, T. et al.: Genome-wide identification, organization and phylogenetic analysis of Dicer-like, Argonaute and RNA-dependent RNA polymerase gene families and their expression analysis during reproductive development and stress in rice. BMC Genomics, 9, 451 (2008)[PubMed]
- Fei, Q., Xia, R. & Meyers, B. C.: Phased, secondary, small interfering RNAs in posttranscriptional regulatory networks. Plant Cell, 25, 2400-2415 (2013)[PubMed]
- 野々村賢一: イネ生殖細胞形成過程を制御する遺伝子群の単離と機能解析. 育種学研究, 9, 147-152 (2007)
- Kelliher, T. & Walbot, V.: Maize germinal cell initials accommodate hypoxia and precociously express meiotic genes. Plant J., 77, 639-652 (2014)[PubMed]
- Nonomura, K. I.: Small RNA pathways responsible for non-cell-autonomous regulation of plant reproduction. Plant Reprod., 31, 21-29 (2018)[PubMed]
- Matzke, M. A. & Mosher, R. A.: RNA-directed DNA methylation: an epigenetic pathway of increasing complexity. Nat. Rev. Genet., 15, 394-408 (2014)[PubMed]
- Hock, J. & Meister, G.: The Argonaute protein family. Genome Biol., 9, 210 (2008)[PubMed]
- Johnson, C., Kasprzewska, A., Tennessen, K. et al.: Clusters and superclusters of phased small RNAs in the developing inflorescence of rice. Genome Res., 19, 1429-1440 (2009)[PubMed]
- Komiya, R., Ohyanagi, H., Niihama, M. et al.: Rice germline-specific Argonaute MEL1 protein binds to phasiRNAs generated from more than 700 lincRNAs. Plant J., 78, 385-397 (2014)[PubMed]
- Ono, S., Liu, H., Tsuda, K. et al.: EAT1 transcription factor, a non-cell-autonomous regulator of pollen production, activates meiotic small RNA biogenesis in rice anther tapetum. PLoS Genet., 14, e1007238 (2018)[PubMed]
- Peragine, A., Yoshikawa, M., Wu, G. et al.: SGS3 and SGS2/SDE1/RDR6 are required for juvenile development and the production of trans-acting siRNAs in Arabidopsis. Genes Dev., 18, 2368-2379 (2004)[PubMed]
- Yoshikawa, M., Peragine, A., Park, M. Y. et al.: A pathway for the biogenesis of trans-acting siRNAs in Arabidopsis. Genes Dev., 19, 2164-2175 (2005)[PubMed]
- Song, X., Li, P., Zhai, J. et al.: Roles of DCL4 and DCL3b in rice phased small RNA biogenesis. Plant J., 69, 462-474 (2012)[PubMed]
- Song, X., Wang, D., Ma, L. et al.: Rice RNA-dependent RNA polymerase 6 acts in small RNA biogenesis and spikelet development. Plant J., 71, 378-389 (2012)[PubMed]
- Zhai, J., Zhang, H., Arikit, S. et al.: Spatiotemporally dynamic, cell-type-dependent premeiotic and meiotic phasiRNAs in maize anthers. Proc. Natl. Acad. Sci. USA, 112, 3146-3151 (2015)[PubMed]
- Fei, Q., Yang, L., Liang, W. et al.: Dynamic changes of small RNAs in rice spikelet development reveal specialized reproductive phasiRNA pathways. J. Exp. Bot., 67, 6037-6049 (2016)[PubMed]
- International Brachypodium Initiative: Genome sequencing and analysis of the model grass Brachypodium distachyon. Nature, 463, 763-768 (2010)[PubMed]
- Jeong, D. H., Schmidt, S. A., Rymarquis, L. A. et al.: Parallel analysis of RNA ends enhances global investigation of microRNAs and target RNAs of Brachypodium distachyon. Genome Biol., 14, R145 (2013)[PubMed]
- Kakrana, A., Mathioni, S. M., Huang, K. et al.: Plant 24-nt reproductive phasiRNAs from intramolecular duplex mRNAs in diverse monocots. Genome Res., 28, 1333-1344 (2018)[PubMed]
- Zhai, J., Jeong, D. H., De Paoli, E. et al.: MicroRNAs as master regulators of the plant NB-LRR defense gene family via the production of phased, trans-acting siRNAs. Genes Dev., 25, 2540-2553 (2011)[PubMed]
- Addo-Quaye, C., Eshoo,. T. W., Bartel, D. P. et al.: Endogenous siRNA and miRNA targets identified by sequencing of the Arabidopsis degradome. Curr. Biol., 18, 758-762 (2008)[PubMed]
- Patel, P., Mathioni, S., Kakrana, A. et al.: Reproductive phasiRNAs in grasses are compositionally distinct from other classes of small RNAs. New Phytol., 220, 851-864 (2018)[PubMed]
- Lu, J., Zhang, C., Baulcombe, D. C. et al.: Maternal siRNAs as regulators of parental genome imbalance and gene expression in endosperm of Arabidopsis seeds. Proc. Natl. Acad. Sci. USA, 109, 5529-5534 (2012)[PubMed]
- Creasey, K. M., Zhai, J., Borges, F. et al.: miRNAs trigger widespread epigenetically activated siRNAs from transposons in Arabidopsis. Nature, 508, 411-415 (2014)[PubMed]
- Dukowic-Schulze, S., Sundararajan, A., Ramaraj, T. et al.: Novel meiotic miRNAs and indications for a role of phasiRNAs in meiosis. Front. Plant Sci., 7, 762 (2016)[PubMed]
- Tamim, S., Cai, Z., Mathioni, S. M. et al.: Cis-directed cleavage and nonstoichiometric abundances of 21-nucleotide reproductive phased small interfering RNAs in grasses. New Phytol., 220, 865-877 (2018)[PubMed]
- Zhang, H., Xia, R., Meyers, B. C. et al.: Evolution, functions, and mysteries of plant ARGONAUTE proteins. Curr. Opin. Plant Biol., 27, 84-90 (2015)[PubMed]
- Olmedo-Monfil, V., Duran-Figueroa, N., Arteaga-Vazquez, M. et al.: Control of female gamete formation by a small RNA pathway in Arabidopsis. Nature, 464, 628-632 (2010)[PubMed]
- Rodriguez-Leal, D., Leon-Martinez, G., Abad-Vivero, U. et al.: Natural variation in epigenetic pathways affects the specification of female gamete precursors in Arabidopsis. Plant Cell, 27, 1034-1045 (2015)[PubMed]
- Singh, M., Goel, S., Meeley, R. B. et al.: Production of viable gametes without meiosis in maize deficient for an ARGONAUTE protein. Plant Cell, 23, 443-458 (2011)[PubMed]
- Oliver, C., Santos, J. L. & Pradillo, M.: Accurate chromosome segregation at first meiotic division requires AGO4, a protein involved in RNA-dependent DNA methylation in Arabidopsis thaliana. Genetics, 204, 543-553 (2016)[PubMed]
- Borges, F., Pereira, P. A., Slotkin, R. K. et al.: MicroRNA activity in the Arabidopsis male germline. J. Exp. Bot., 62, 1611-1620 (2011)[PubMed]
- Tucker, M. R., Okada, T., Hu, Y. et al.: Somatic small RNA pathways promote the mitotic events of megagametogenesis during female reproductive development in Arabidopsis. Development, 139, 1399-1404 (2012)[PubMed]
- Nogueira, F. T., Madi, S., Chitwood, D. H. et al.: Two small regulatory RNAs establish opposing fates of a developmental axis. Genes Dev., 21, 750-755 (2007)[PubMed]
- Chitwood, D. H., Nogueira, F. T., Howell, M. D. et al.: Pattern formation via small RNA mobility. Genes Dev., 23, 549-554 (2009)[PubMed]
- 野田口理孝: 植物の全身を長距離移行するRNA. 植物科学最前線, 8, 121 (2017)
- Bai, S., Kasai, A., Yamada, K. et al.: A mobile signal transported over a long distance induces systemic transcriptional gene silencing in a grafted partner. J. Exp. Bot., 62, 4561-4570 (2011)[PubMed]
- Liu, H. & Nonomura, K. I.: A wide reprogramming of histone H3 modifications during male meiosis I in rice is dependent on the Argonaute protein MEL1. J. Cell Sci., 129, 3553-3561 (2016)[PubMed]
- Ye, R., Wang, W., Iki, T. et al.: Cytoplasmic assembly and selective nuclear import of Arabidopsis Argonaute4/siRNA complexes. Mol. Cell, 46, 859-870 (2012)[PubMed]
- You, C., Cui, J., Wang, H. et al.: Conservation and divergence of small RNA pathways and microRNAs in land plants. Genome Biol., 18, 158 (2017)[PubMed]
- Xia, R., Xu, J., Arikit, S. et al.: Extensive families of miRNAs and PHAS loci in Norway spruce demonstrate the origins of complex phasiRNA networks in seed plants. Mol. Biol. Evol., 32, 2905-2918 (2015)[PubMed]
- Shivaprasad, P. V., Chen, H. M., Patel, K. et al.: A microRNA superfamily regulates nucleotide binding site-leucine-rich repeats and other mRNAs. Plant Cell, 24, 859-874 (2012)[PubMed]
- de Vries, S., Kloesges, T. & Rose, L. E.: Evolutionarily dynamic, but robust, targeting of resistance genes by the miR482/2118 gene family in the Solanaceae. Genome Biol. Evol., 7, 3307-3321 (2015)[PubMed]
- Liu, L., Ren, S., Guo, J. et al.: Genome-wide identification and comprehensive analysis of microRNAs and phased small interfering RNAs in watermelon. BMC Genomics, 19, 111 (2018)[PubMed]
- Chen, K., Liu, L., Zhang, X. et al.: Phased secondary small interfering RNAs in Panaxnotoginseng. BMC Genomics, 19, 41 (2018)[PubMed]
- Ding, D., Liu, J., Dong, K. et al.: PNLDC1 is essential for piRNA 3’ end trimming and transposon silencing during spermatogenesis in mice. Nat. Commun., 8, 819 (2017)[PubMed]
- Nishimura, T., Nagamori, I., Nakatani, T. et al.: PNLDC1, mouse pre-piRNA Trimmer, is required for meiotic and post-meiotic male germ cell development. EMBO Rep., 19, e44957 (2018)[PubMed]
- Han, B. W., Wang, W., Li, C. et al.: piRNA-guided transposon cleavage initiates Zucchini-dependent, phased piRNA production. Science, 348, 817-821 (2015)[PubMed]
- Mohn, F., Handler, D. & Brennecke, J.: piRNA-guided slicing specifies transcripts for Zucchini dependent, phased piRNA biogenesis. Science, 348, 812-817 (2015)[PubMed]
- Yang, Z., Chen, K. M., Pandey, R. R. et al.: PIWI slicing and EXD1 drive biogenesis of nuclear piRNAs from cytosolic targets of the mouse piRNA pathway. Mol. Cell, 61, 138-152 (2016)[PubMed]
- Li, X. Z., Roy, C. K., Dong, X. et al.: An ancient transcription factor initiates the burst of piRNA production during early meiosis in mouse testes. Mol. Cell, 50, 67-81 (2013)[PubMed]
- Gou, L. T., Dai, P., Yang, J. H. et al.: Pachytene piRNAs instruct massive mRNA elimination during late spermiogenesis. Cell Res., 24, 680-700 (2014)[PubMed]
- Aravin, A. A., van der Heijden, G. W., Castaneda, J. et al.: Cytoplasmic compartmentalization of the fetal piRNA pathway in mice. PLoS Genet., 5, e1000764 (2009)[PubMed]
- Brennecke, J., Aravin, A. A., Stark, A. et al.: Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell, 128, 1089-1103 (2007)[PubMed]
- Li, C. F., Pontes, O., El-Shami, M. et al.: An ARGONAUTE4-containing nuclear processing center colocalized with Cajal bodies in Arabidopsis thaliana. Cell, 126, 93-106 (2006)[PubMed]
- Li, S., Le, B., Ma, X. et al.: Biogenesis of phased siRNAs on membrane-bound polysomes in Arabidopsis. Elife, 5 (2016)[PubMed]
- Choi, K., Zhao, X., Kelly, K. A. et al.: Arabidopsis meiotic crossover hot spots overlap with H2A.Z nucleosomes at gene promoters. Nat. Genet., 45.,1327-1336 (2013)[PubMed]
- Choi, K., Zhao, X., Tock, A. J. et al.: Nucleosomes and DNA methylation shape meiotic DSB frequency in Arabidopsis thaliana transposons and gene regulatory regions. Genome Res., 28, 532-546 (2018)[PubMed]
著者プロフィール
略歴:2015年 総合研究大学院大学生命科学研究科博士課程 修了,同年より国立遺伝学研究所 特任研究員.
研究テーマ:植物,とくに作物における減数分裂の制御機構およびその育種への応用.
野々村 賢一(Ken-Ichi Nonomura)
国立遺伝学研究所 准教授.
研究室URL:http://nonomuralab-nig.sakura.ne.jp/top_j.html
© 2019 小野聖二郎・野々村賢一 Licensed under CC 表示 2.1 日本